REGISTRO DOI: 10.5281/zenodo.10971014
Felipe Da Silva Mendonça De Melo
Júlia Lisboa Beserra E Silva
Tâmara Lorena De Souza Silva
Orientadora: Profa. Ma. Fabiana Nunes de Carvalho Mariz
RESUMO
A resistência bacteriana é um dos piores problemas de saúde pública enfrentados no século XXI. As Infecções Relacionadas à Assistência à Saúde (IRAS) causadas por Bactérias Multirresitentes (BMR) refletem as altas taxas de mortalidade de pessoas internadas em Unidades de Terapia Intensiva (UTI). O objetivo deste estudo foi buscar evidências na literatura sobre a cultura de vigilância epidemiológica realizada em pacientes adultos de UTI, além de relatar fatores de risco, implicações clínicas e as medidas de controle adotadas nestas unidades críticas. Para isso, foi realizada uma revisão de literatura narrativa para responder à pergunta “Como é realizada a cultura de vigilância epidemiológica em UTI adulto e quais são os fatores de risco, implicações clínicas e as medidas de controle adotadas?”. Foi utilizada a base de dados PubMed para a realização das buscas dos artigos. Inicialmente foram filtrados trinta e dois estudos, porém, ao final, foi realizada a leitura e análise de oito. Nos artigos estudados, a cultura de vigilância foi realizada após a coleta de swab retal, nasal além de outras amostras, que foram semeadas em meios de cultura específicos. Depois dos microrganismos identificados, foi realizado o teste de sensibilidade aos antimicrobianos, que foi imprescindível para os achados epidemiológicos. Ficou evidenciado a predominância da bactéria Acinetobacter baumannii, além de achados de Pseudomonas aeruginosa, Staphylococcus aureus resistente à meticilina,enterobactérias resistentes a carbapenêmicos, entre outras. O uso de antimicrobianos em ambiente hospitalar foi um dos maiores fatores de risco para a infecção por BMR, juntamente com a ventilação mecânica e o tempo prolongado de internação. Pacientes colonizados, foram mantidos com precaução de contato.O estudo possibilitou compreender que bactérias multirresistentes são um grave problema de saúde pública, principalmente quando se trata de ambientes de cuidado intensivo como a UTI, e que a realização da técnica de cultura de vigilância epidemiológica é importante e imprescindível na triagem de BRM em pacientes internados. Além disso, a implementação de um Programa de Gerenciamento de Antimicrobianos (PGA) se faz necessária, juntamente com a fiscalização e cuidados abrangentes em ambientes hospitalares.
Palavras–chave: farmacorresistência bacteriana múltipla; conduta expectante; centro de terapia intensiva; infecções nosocomiais.
ABSTRACT
Bacterial resistance is one of the most significant public health challenges faced in the 21st century. Healthcare-Associated Infections (HAI) caused by Multidrug-Resistant Bacteria (MDR) reflect high mortality rates among individuals hospitalized in Intensive Care Units (ICUs). The aim of this study was to search for evidence in the literature regarding the epidemiological surveillance culture conducted in adult ICU patients, as well as to report risk factors, clinical implications, and control measures adopted in these critical units. To achieve this, a narrative literature review was conducted to answer the question, “How is epidemiological surveillance culture performed in adult ICUs, and what are the risk factors, clinical implications, and control measures adopted?” The PubMed database was used for article searches. Initially, thirty-two studies were screened; however, in the end, eight were read and analyzed. The surveillance culture was carried out after collecting rectal and nasal swabs, among other samples, which were cultured in specific media. After identification, antimicrobial sensitivity testing was conducted, which was crucial for epidemiological findings. The predominance of Acinetobacter baumannii bacteria was evident, along with findings of Pseudomonas aeruginosa, methicillin-resistant Staphylococcus aureus, and carbapenem-resistant enterobacteria, among others. The use of antimicrobials in a hospital environment was one of the major risk factors for MDR infection, along with mechanical ventilation and prolonged hospitalization. Colonized patients were kept under contact precautions. The study helped to understand that multidrug-resistant bacteria pose a serious public health problem, especially in intensive care settings like the ICU, and that conducting the epidemiological surveillance culture technique is important and indispensable for screening MDR in hospitalized patients. Furthermore, the implementation of an Antimicrobial Management Program is necessary, along with comprehensive oversight and care in healthcare environments.
Keywords: drug resistance, multiple bacterial; watchuful waiting; intensive care units; cross infection.
1 INTRODUÇÃO
No ano de 2017 a Organização Mundial da Saúde (OMS) publicou em seu site uma lista contendo as 12 bactérias de maior risco à saúde humana, devido à resistência antimicrobiana e para as quais necessitam da criação de novos antibióticos urgentemente. A lista foi dividida em prioridades, sendo elas: crítica, alta e média (OMS, 2017). A Organização das Nações Unidas (ONU) publicou em 2019 que “doenças resistentes a medicamentos poderão causar 10 milhões de mortes por ano no mundo” se até 2050 a situação não for revertida os danos serão sanitários, econômicos e sociais (ONU, 2019).
Sabe-se que quando demonstrada a questão da resistência bacteriana, os impactos causados são notórios em diversas áreas, como por exemplo, o aumento do custo financeiro e logístico de tratamentos de pacientes, que fazem com que a estadia hospitalar seja prolongada, e possivelmente potencializando as chances de mortalidade, o que justifica e norteia a precedência do tema proposto. Levando em consideração que bactérias adquirem resistência a novos fármacos, a eficácia dos antimicrobianos é diminuída e limitada, e por consequência, aumenta-se o número de infecções de difícil tratamento. À medida que as opções terapêuticas se esgotam, infecções antes tratáveis com facilidade, poderão adquirir proporções mais severas, dada a limitação de recursos para seu combate eficaz (Rocha, 2015).
Para conhecer as bactérias resistentes que estão presentes no ambiente hospitalar realiza-se a cultura de vigilância epidemiológica. O protocolo dessa cultura resume-se na coleta de amostras de pacientes internados ou que possam necessitar de internação, principalmente em Unidades de Terapia Intensiva (UTI), para identificar a colonização por patógenos em sítios não estéreis e monitorar o aparecimento de Bactérias Multirresistentes (BMR) durante e após o período de permanência em âmbito hospitalar (Brasil, 2021).
Os impactos causados pelos eventos adversos oriundos das Infecções Relacionadas à Assistência à Saúde (IRAS) refletem nos altos índices de morbimortalidade de internados em UTI (Araújo et al, 2017). A repercussão gera ônus econômicos e sociais para a população, e para o sistema de saúde vigente, além de favorecer a disseminação de BMR para a sociedade, considerando-se, um sério desafio à saúde pública e preocupação constante para profissionais da saúde, pesquisadores e gestores hospitalares (Santos, 2004).
Sendo assim, essa preocupação intensifica-se em ambientes de alta complexidade, como as unidades de terapia intensiva, onde são realizados procedimentos que favorecem a exposição do paciente a microrganismos, como uso de suporte ventilatório e de cateteres venosos, por exemplo (Rocha, 2015).
Tendo em vista o grave problema que as bactérias multirresistentes representam para a saúde pública e para pacientes internados, o objetivo do estudo é buscar evidências na literatura sobre a cultura de vigilância epidemiológica realizada em pacientes adultos de unidades de terapia intensiva além relatar fatores de risco, implicações clínicas e as medidas de controle adotadas nestas unidades críticas.
Posto isso, o presente estudo segue o modelo Introdução, Revisão de Literatura, Material e Métodos, Resultados, Discussão e Conclusão (IRMRDC), e é composto por partes bem definidas. Este se inicia com a introdução, seguida da revisão de literatura, na qual é a apresentada a fundamentação teórica, com base em pesquisas bibliográficas que subsidiaram todo o desenvolvimento da investigação, seguida pelos materiais e métodos utilizados tanto na coleta quanto na análise dos dados encontrados. Por fim, a apresentação dos resultados obtidos, a discussão realizada pelos autores e a conclusão.
2 REFERENCIAL TEÓRICO
2.1 ESTRUTURA BACTERIANA
As células bacterianas possuem diversas estruturas, algumas essenciais e outras presentes apenas em algumas espécies (Alterthum, 2015a). Nessa sessão do referencial teórico, foi escolhido trabalhar as estruturas da célula bacteriana que são alvos de antimicrobianos.
2.1.1 Parede celular
A parede celular é uma estrutura complexa e semirrígida, é ela que protege o interior da célula de alterações externas e define sua forma. Tem como função a prevenção da ruptura da célula quando a pressão osmótica interna for maior que a externa (Tortora; Funke; Case, 2017). Ela também cumpre um importante papel na divisão celular e atua como modelo em sua biossíntese (Morse; Meitzner, 2014b). O peptideoglicano é a principal macromolécula que compõe a parede celular, porém existem diferenças entre as paredes das bactérias Gram-negativas e Grampositivas (Tortora; Funke; Case, 2017). Na Gram-positiva, a parede é constituída em sua maior parte por peptideoglicano, além de proteínas e ácido teicóico (Alterthum, 2015a). Já na Gram-negativa, a parede possui uma menor porção de peptideoglicano o que a torna mais suscetível a rompimentos (Tortora; Funke; Case, 2017).
Entretanto, a parede celular das bactérias Gram-negativas possui uma membrana externa, composta por Lipopolissacarídeos (LPS), lipoproteínas e fosfolipídeos. Essa membrana confere uma proteção contra detergentes, metais pesados, sais biliares, antibióticos e enzimas digestivas. Ainda assim, ela possui uma permeabilidade devido a canais como o de porina, que possibilita a entrada de moléculas como nucleotídeos, dissacarídeos, peptídeos, aminoácidos, vitamina B12 e ferro (Tortora; Funke; Case, 2017).
Nas bactérias Gram-negativas existe também um espaço entre as membranas plasmática e externa, chamado de espaço periplasmático, onde além de peptideoglicano, estão situadas enzimas e proteínas como: enzimas hidrolíticas (proteases, nucleases e lipases); enzimas inativadoras de drogas (β-lactamase); proteínas de transporte de solutos (Alterthum, 2015a).
2.1.2. Membrana celular
A membrana plasmática é uma estrutura vital para a célula bacteriana e é responsável pela separação do meio interno e externo. Em sua composição 60% corresponde às proteínas e os demais 40% a uma camada de lipídeos, dentre eles os fosfolipídeos. Na sua porção interna, os ácidos graxos fornecem sua característica hidrofóbica, enquanto na externa sua porção hidrofílica se expõe ao meio aquoso (Alterthum, 2015a).
Sua principal função é a de servir como barreira seletiva para entrada e saída de materiais da célula. Onde determinadas moléculas como água, oxigênio e dióxido de carbono conseguem atravessar, porém outras como proteínas, não conseguem. Outra função importante é a de digestão de nutrientes a partir de suas enzimas e assim a produção de energia (Tortora; Funke; Case, 2017).
2.1.3 Ribossomos e Nucleoide
Todas as células bacterianas possuem em seu citoplasma os ribossomos, onde ocorre a síntese proteica. Essa estrutura possui duas subunidades denominadas 50S e 30S que consistem em proteína e Ácido Ribonucleico (RNA) ribossomal (Tortora; Funke; Case, 2017).
Nucleoide é a estrutura onde fica localizado o cromossomo bacteriano, normalmente uma única molécula longa e contínua de DNA de fita dupla e que está frequentemente em arranjo circular (Tortora; Funke; Case, 2017).
2.1.4 Plasmídeo
Outra estrutura importante, porém, está presente em apenas algumas bactérias. Essa é uma molécula extracromossômica circular de DNA e que tem como função, conferir à bactéria vantagens seletivas, armazenando informações de degradação de substratos ou de síntese de enzimas que inativam antimicrobianos. Sua replicação pode ocorrer na divisão celular bacteriana ou no processo de conjugação (Padilha; Costa, 2015).
2.2. ANTIMICROBIANOS
O tratamento de doenças feito com substâncias químicas, é denominado quimioterapia. Algumas dessas substâncias, são sintetizadas em laboratório, e assim chamadas de quimioterápicos e outras produzidas por seres vivos que são chamadas de antibióticos. Tanto os antibióticos como os quimioterápicos, podem ser antimicrobianos e sua ação pode ser bacteriostático, ou seja, levar à inibição do crescimento antimicrobiano, ou bactericida levando à morte da bactéria (Alterthum, 2015b).
Antimicrobianos são fármacos que podem utilizar das diferenças bioquímicas existentes entre os microrganismos e os seres humanos. Eles são eficazes no tratamento de infecções, pois são seletivos e conseguem atingir o microrganismo sem causar grandes prejuízos às células do hospedeiro (Kisgen, 2016).
2.2.1. Mecanismos de ação dos antimicrobianos
A classificação dos antimicrobianos pode ser feita por sua estrutura química como β-lactâmicos e aminoglicosídeos, ou por seu mecanismo de ação, inibidores da síntese da parede celular, inibidores da função da membrana plasmática, inibidores da síntese de proteínas e inibidores da síntese do ácido nucleico (Kisgen, 2016).
2.2.1.1 Inibição da síntese da parede celular
A inibição da síntese da parede celular é um importante mecanismo de ação utilizado pelas classes de β-lactâmicos (penicilina, cefalosporinas, carbapenêmicos e monobactamicos) e glicopeptídeos (vancomicina e teicoplanina) (Alterthum, 2015b).
A síntese da parede bacterina ocorre em três etapas, a primeira no citoplasma, a segunda na membrana plasmática e a terceira na parte externa à membrana com a participação das Proteínas Fixadoras de Penicilina (PBP) na síntese de peptideoglicano. É durante a terceira etapa que os β-lactâmicos atuam, se fixando à PBP impedindo a síntese de peptideoglicano (Alterthum, 2015b).
Já os glicopeptídeos agem nas etapas iniciais, necessitando, portanto, penetrar na membrana plasmática, para serem eficazes (Carroll, 2014). Além de causar danos à membrana, sua ação principal, consiste em se ligar a uma unidade terminal do pentapeptideo peptoglicano, que é precursor do peptídeoglicano, e com isso impedindo ligações que o tornará enfraquecido e suscetível à lise (Deck; Winston, 2017a).
2.2.1.1 Inibição da função da membrana plasmática
Os antimocrobianos que atuam causando danos à membrana plasmática, são polimixina B e daptomicina, sua ação provoca alteração na permeabilidade da membrana, causando a perda de metabólitos importantes para a bactéria (Tortora; Funke; Case, 2017).
A polimixina B causa esse dano à membrana, por ser um peptídeo cíclico com atividade detergente contra o componente fosfatidiletanolamina que está presente na membrana. Enquanto a daptomicina é um lipopeptídeo que se liga à membrana de modo dependente de cálcio, o que causa a despolarização do potencial de repouso da membrana levando à perda de potássio para o meio extracelular (Carroll, 2014).
2.2.1.3 Inibição da síntese de proteínas
A síntese de proteínas ocorre nos ribossomos, dentre as classes que utilizam sua inibição como mecanismo de ação, estão os aminoglicosídeos, tetraciclinas, macrolídeos, estreptograminas, oxazolidinonas, lincosamidas, cloranfenicol, cetolídeos e glicilciclina além da mupirocina (Deck; Winston, 2017b).
A ação desses antimicrobianos, ocorre de diferentes formas. Os aminoglicosídeos invadem a célula atravessando a membrana externa pela difusão passiva nos canais de porina, em seguida invade de maneira ativa pela membrana plasmática e já dentro da célula, inibe a síntese de três maneiras: interferindo com o complexo de iniciação de formação do peptídeo; leitura errônea no mRNA, provocando a incorporação de aminoácidos incorretos e assim a produção de uma proteína afuncional ou tóxica; clivagem do polissomos em monossomos não funcionais (Deck; Winston, 2017b).
Já os macrolídeos, azalídeos, cetolídeos, clindomicina, licosamidas, cloranfenicol, estreptograminas e oxazolidinonas se assemelham no mecanismo de ação ao se ligarem à subunidade 50S do ribossomo e assim interferirem na formação dos complexos de iniciação para a síntese de cadeia peptídica. Enquanto as tetraciclinas e glicilciclinas se ligam à subunidade 30S impedindo a ligação da molécula de RNA transportador (RNAt) e assim, inibe a síntese de proteínas (Carroll, 2014).
2.2.1.4 Inibição da síntese de ácidos nucleicos
A inibição da síntese de ácidos nucleicos pode ocorrer de diversas maneiras, e os antimicrobianos que possuem esse mecanismo de inibição são rifampicina, trimetoprima, quinolonas, fluoroquinolonas e sulfonamidas (Carroll, 2014).
No caso da rifampicina, a inibição ocorre devido à sua forte ligação à enzima RNA polimerase DNA dependente, alterando a estrutura dessa enzima, inibindo assim a síntese de RNA. Já as quinolonas e fluoroquinolonas, bloqueiam a DNA girasse e topoisomerase, que são responsáveis pela replicação e reparo do DNA, dessa forma inibem a síntese de DNA (Carroll, 2014).
As sulfonamidas e trimetoprimas atuam na inibição da síntese de purina, metionina, timina e serina. As sulfonamidas bloqueiam a transformação de ácido paraminobenzóico em ácido diidropteróico, enquanto as trimetoprimas bloqueiam a transformação de ácido diidrofólico em ácido tetraidrofólico (Alterthum, 2015b).
2.2.1 Resistência aos antimicrobianos
Para que um antimicrobiano iniba o crescimento ou mate uma bactéria além do tempo de exposição e da concentração, é necessário que haja um alvo, que o antibacteriano atinja esse alvo e que o antimicrobiano não seja inativado antes de atingir o alvo (Alterthum, 2015b).
Quando o crescimento das bactérias não é inibido pela maior concentração de antimicrobiano tolerada pelo hospedeiro, considera-se que há uma resistência ao fármaco (Whalen; Finkel; Panavelil, 2016). Essa resistência pode ser intrínseca, ou adquirida (Alterthum, 2015b).
É definida como forma intrínseca, quando a resistência está atrelada às propriedades inerentes da bactéria, como por exemplo, a resistência aos glicopeptídeos que as bactérias Gram-negativas possuem, devido à impermeabilidade da sua membrana externa. A forma adquirida, é quando ocorre uma transferência de genes entre duas bactérias. Sendo assim, essa transferência pode se dar por três formas diferentes, transformação, transdução e conjugação (Christaki; Marcou; Tofarides, 2019).
Na transformação, ocorre uma recombinação genética, onde os fragmentos livres de DNA de uma bactéria são absorvidos por outras, que irão incorporá-los ao seu cromossomo. Na transdução, a transferência de material genético é realizada por um bacteriófago. E já na conjugação, ocorre um contato entre as bactérias por meio de um pili, e a informação genética contida no plasmídeo passada de uma bactéria para outra, que pode transportar um maior número de genes de resistência antimicrobiana (Christaki; Marcou; Tofarides, 2019).
Para compreender a aquisição de mecanismos de resistência aos antimicrobianos, é crucial considerar uma variedade de fatores causais, como os relacionados interações patógeno-medicamento, interações patógeno-hospedeiro, taxas de mutação do patógeno, surgimento de clones resistentes a antimicrobianos bem-sucedidos, taxas e transmissão de patógenos entre humanos, animais e meio ambiente (Holmes et al, 2016).
Estudos demonstram que a resistência antimicrobiana, surgiu em parte, por ocorrência da pressão seletiva, causada pela utilização de antimicrobianos fora da medicina humana, como em alimentação de animais e na agricultura (Holmes et al, 2016). A Organização Mundial de Saúde reforça essa multifatorialidade, e afirma que entre as principais ações que levam à resistência, estão o uso indevido e excessivo de antimicrobianos; falta de acesso a água potável e saneamento básico; medidas ineficazes na prevenção e controle de doenças e infecções em unidades de saúde; falta de consciência e conhecimento e descumprimento da legislação (OMS, 2021).
Os principais mecanismos de resistência antimicrobiana são: alteração da permeabilidade à entrada de antibacteriano; alteração do local de ação ou alvo; bomba de efluxo que é expulsão do antimicrobiano da célula e o mecanismo enzimático quando há produção de novas enzimas que não sofrem ação do antimicrobiano (Alterthum, 2015b).
2.2.1.1. Alteração da permeabilidade
A alteração da permeabilidade da membrana afeta os antimicrobianos que tem a membrana plasmática como alvo. Portanto as bactérias que utilizam esse mecanismo, alteram sua membrana e limitam o influxo de substâncias do meio externo evitando a entrada de compostos tóxicos. As substâncias mais afetadas, são as que possuem moléculas hidrofílicas, como β-lactâmicos, tetraciclinas e algumas fluoroquinolonas (Munita; Arias, 2016).
2.2.1.2. Alteração do local de ação
A alteração do local de ação é uma defesa aos antimicrobianos que possuem o mecanismo de inibição de síntese proteica, como os aminoglicosídeos, tetraciclinas e macrolídeos. Nessa forma de resistência ocorrem pequenas modificações no sítio de ação dos antimicrobianos para que não altere as funções celulares (Tortora; Funke; Case, 2017).
As modificações no alvo podem ocorrer de três formas, mutações pontuais nos genes que codificam o sítio alvo, alterações enzimáticas do sítio de ligação (por exemplo, adição de grupos metilo) e substituição ou desvio do alvo original (Munita; Arias, 2016).
Staphylococcus aureus Resistente à Meticilina(MRSA sigla em inglês) é um exemplo de sucesso desse mecanismo de resistência, que incluiu uma PBP, portanto os β-lactâmicos continuam se ligando à PBP normal, porém a PBP adicional exerce sua função na síntese da parede celular (Tortora; Funke; Case, 2017).
2.2.1.3. Bomba de efluxo
Nesse mecanismo de resistência, algumas proteínas da membrana plasmática funcionam como bombas ejetoras para retirar a substância tóxica, que no caso é o antimocrobiano, de dentro da célula (Tortora; Funke; Case, 2017).
A resistência à tetraciclina é o exemplo mais conhecido desse mecanismo. Porém diversas bactérias possuem essa resistência a classes como fluoroquinolonas, macrolídeos. Inclusive em Pseudomonas aeruginosa e enterobactérias, esse mecanismo é utilizado contra múltiplos grupamentos de antimicrobianos que inclui fluoroquinolonas, cloranfenicol, eritromicina e β lactâmicos (Tavares, 2014).
2.2.1.4. Mecanismo enzimático
No mecanismo enzimático, a bactéria produz enzimas que inativam o fármaco, adicionando porções químicas específicas que destroem a própria molécula do antimicrobiano, tornando-o incapaz de agir em seu alvo (Munita; Arias, 2016).
Esse é o mecanismo mais utilizado para resistência bacteriana, e a principal enzima produzia é a β-lactamase, ela hidrolisa a ligação de amida do anel β-lactâmico de penicilinas, cefalosporinas, monobactâmicos e carbapenemases (Tavares, 2014). Existem também outras enzimas produzidas para esse fim de resistência como carbapenemases utilizada por Klebsiella pneumoniae carbepenemase (KPC), adeniltransferases, nucleotidiltransferases e fofotransferases que inativam aminoglicosídeos e a cloranfenicol-acetiltransferase que inativa cloranfenicol (Tavares, 2014).
2.3. CULTURA DE VIGILÂNCIA
A cultura de vigilância epidemiológica é uma técnica de isolamento para a identificação das colonizações por Bactérias Multirresistentes (BMR). Essa técnica é utilizada não apenas para identificação dos microrganismos, mas também com o intuito de monitorar, compreender e rastrear a propagação desses patógenos, a fim de evitar transmissões cruzadas e o desenvolvimento de novas infecções (Almeida, 2016).
Sendo assim, as culturas de vigilância epidemiológicas desempenham um papel fundamental na prevenção e controle de agentes causadores de Infecções Relacionadas à Assistência à Saúde (IRAS) permitindo identificar e antecipar se ocorreu a introdução desses organismos nas unidades médicas (Silva et al, 2012). Posto isso, recomenda-se realizar a cultura de vigilância principalmente em algumas populações alvo, como por exemplo, pacientes internados em unidades de alto risco para a colonização por BMR (Brasil, 2021).
O principal exemplo de unidades críticas para esses patógenos, é a Unidade de Terapia Intensiva (UTI), onde infecções nosocomiais afetam cerca de 30% dos pacientes e estão diretamente relacionadas com morbidade e mortalidade. Além disso, pacientes de UTI são mais vulneráveis IRAS, do que comparado com as demais unidades, e sabe-se que, esses doentes têm de cinco a dez vezes mais probabilidades de contrair essas infecções, o que pode representar cerca de 20% das infecções totais de um hospital (Apud Vincent, 2003; Andrade; Leopoldo; Haas, 2006). De acordo com a Resolução n° 2.271, de 14 de fevereiro de 2020 do Conselho Federal de Medicina,
UTI: ambiente hospitalar com sistema organizado para oferecer suporte vital de alta complexidade, com múltiplas modalidades de monitorização e suporte orgânico avançados para manter a vida durante condições clínicas de gravidade extrema e risco de morte por insuficiência orgânica. Essa assistência é prestada de forma contínua, 24 horas por dia, por equipe multidisciplinar especializada (Resolução CFM Nº 2.271/2020).
Em vista disso, a cultura de vigilância realizada no ambiente hospitalar tem como seu principal objetivo, conhecer as circunstâncias e o status em que se encontra o paciente. Além disso, estabelece precauções de contato e manejo, identifica transmissões cruzadas entre unidades distintas ou áreas do mesmo hospital, avalia a epidemiologia de uma unidade de saúde e realiza a descontaminação de portadores (Oplustil et al, 2019).
Em 2015, o Governo do Distrito Federal implementou a nota técnica n° 01/2015, que diz respeito sobre “Orientações para medidas de prevenção e controle de bactérias multirresistentes do GDF”. O documento propõe às instituições coleta de culturas de vigilância para BMR em pacientes de UTI, considerando a epidemiologia local, e o perfil do paciente. Em outros aspectos, delimitam-se as, precauções de contato e atenção, rastreamento de contactantes e transporte de amostras e pacientes, além da coleta de material para cultura de vigilância, a fim de pesquisar e isolar bactérias consideradas resistentes (Distrito Federal, 2015).
2.3.1. Coleta de materiais para a cultura de vigilância
Na coleta para realização de cultura de vigilância, a escolha da amostra clínica e sítios anatômicos deve levar em consideração o tipo de microrganismo a ser pesquisado (Oplustil et al, 2019). Porém, cada unidade hospitalar que trabalha com cultura de vigilância deve adotar o seu próprio protocolo de implementação conforme o perfil epidemiológico de sua instituição (Franco, 2017). Segundo a Nota Técnica n° 01/2015, o Serviço de Controle de Infecção Hospitalar (SCIH) deverá elaborar um protocolo contendo todos os critérios e os procedimentos para a coleta das culturas de vigilância, que inclua a indicação da coleta, a escolha dos sítios anatômicos o número de amostras, entre outros (Distrito Federal, 2015).
O manual da Agência Nacional de Vigilância Sanitária (ANVISA) de 2021 sobre prevenção de infecções por microrganismos resistentes, sugere a coleta dos materiais em seus respectivos sítios da seguinte maneira: pesquisa de Staphylococcus aureus resistente à meticilina (MRSA) deve ser realizada com coleta de swab em ambas as cavidades nasais, e de forma adicional pode se coletar swab de orofaringe, perianal e de feridas; pesquisa de enterococos resistente à vancomicina (VRE) realiza a coleta swab retal ou perianal e se precisar uma amostra de fezes para coprocultura; isolamento de enterobactérias resistentes a carbapenêmicos (CRE) utiliza o swab perianal ou retal. Demais bactérias como Acinetobacter baumannii e Pseudomonas aeruginosa coleta-se na região orofaríngea, endotraqueal, retal, inguinal ou amostra de ferida (Brasil, 2021).
Essas amostras devem ser coletadas antes de qualquer lavagem ou desinfecção do local, utilizando swab ou recipientes estéreis. Em coleta por swab é recomendado os meios de transporte Amies ou Stuart. Essas amostras devem ser encaminhadas imediatamente ao laboratório de microbiologia, em temperatura ambiente, e dessa forma serão estáveis por no máximo 24 horas (Oplustil et al, 2019). Os momentos mais promissores para a coleta são na admissão do paciente na UTI, na transferência de unidades ou na triagem dos pacientes contactantes de um portador de BMR (Brasil, 2021).
2.3.2. Realização da cultura de vigilância
A cultura de microrganismos refere-se ao procedimento de propagação de seres microscópicos pelo fornecimento de condições ambientais e nutritivas apropriadas. Os meios de cultura, são formulados para permitirem o crescimento de diferentes espécies bacterianas, mas, também existem os meios que fazem distinção entre espécies e aqueles que isolam um único microrganismo presente na amostra (Morse; Meitzner, 2014a).
A metodologia na aplicação destas culturas de vigilância é feita a partir da coleta de materiais biológicos do paciente, com swab em maioria nasal e retal como já explicado anteriormente, seguida de identificação do gênero ou espécie bacteriana por técnicas manuais ou automatizadas, e posteriores testes de sensibilidade aos antimicrobianos, a fim de se detectar a possível resistência bacteriana (O’Brien; Stelling, 2011).
O procedimento para cultura de vigilância epidemiológica leva em consideração o microrganismo, o sítio anatômico de coleta e o possível microrganismo a ser pesquisado para a escolha do meio de cultura mais apropriado (Oplustil et al, 2019). Meios de cultura seletivos e específicos para os microrganismos que serão pesquisados são os mais utilizados para CVE, a técnica correta de semeadura é a de estrias sobre os meios de cultura sólido, seguindo a sequência dos mais ricos para os mais seletivos, como por exemplo: ágar chocolate, ágar sangue e MacConkey, respectivamente (Brasil, 2013).
A semeadura para cultivo qualitativo pode ser feita com o próprio swab (do meio de transporte), ou amostra do material removida com alça estéril flambada, ou alça de plástico não reutilizável e semeada de forma a obter um gradiente decrescente de concentração do inóculo, que permita o isolamento de todas as colônias diferentes. O cultivo quantitativo baseia-se na semeadura de um volume conhecido de material, medido em microlitros (µL) e finalizando a contagem do número de Unidades Formadoras de Colônias (UFC) (Brasil, 2013).
2.3.3. Teste de sensibilidade aos antimicrobianos
A bactéria isolada em meio de cultura, após a sua identificação, deverá ser testada diante um grupo específico de antibióticos, que serão selecionados de acordo com a espécie bacteriana e o material clínico, essa prática se chama Teste de Sensibilidade aos Antimicrobianos (TSA) (Oplustil et al, 2019). O método de discodifusão é uma abordagem antiga e manual, porém uma das mais utilizadas para realizar o teste. Essa forma é qualitativa, ou seja, só permite classificar a amostra bacteriana em sensível (S), sensível ampliando a exposição (I) ou resistente (R) ao antibiótico (BrCAST, 2017). Segundo o BrCAST, Bazilian Committee on Antimicrobial Susceptibility Testing, o TSA de disco-difusão deverá ser realizado em placa de meio sólido ágar Müeller Hinton (MH) (BrCAST, 2017).
Essa técnica, é adequada para avaliar a maioria dos patógenos bacterianos, é versátil em relação aos antimicrobianos que podem ser testados, além de não necessitar de automatização nem equipamentos especiais ou de custo elevados (BrCAST, 2017). O TSA por disco-difusão apresenta vantagens por ser um método prático, de fácil execução, e que possui reagentes e materiais econômicos. Entretanto, o método também possui limitações, como a dificuldade na avaliação da sensibilidade aos antimicrobianos que se difundem mal através do ágar, além da baixa interpretação para microrganismos anaeróbios ou fastidiosos (Jorgensen, 1997).
Fora o uso dos métodos manuais, muitos laboratórios de microbiologia utilizam testes automáticos para diagnóstico e triagem. Em relação ao TSA, o processo analítico é otimizado e gera uma resposta rápida à necessidade terapêutica (Abrantes; Nogueira, 2021). O TSA automatizado, semi-quantitativo é liberado não só pelas categorias sensível, sensível aumentando a exposição, ou resistente, mas também possui a liberação da Concentração Inibitória Mínima (CIM) aproximada (Amaral; Barbosa; Correia, 2017). O CIM é a menor concentração do antimicrobiano que será capaz de inibir a sua multiplicação e estabelecer os pontos de corte (breakpoints) para interpretação do TSA. (Martino, 2015).
Existem diversos equipamentos automatizados utilizados, com diferentes metodologias, por exemplo, o sistema VITEK da BioMérieux, utiliza cartões de testes, que são inoculados e incubados pelo equipamento, empregando a metodologia de turbidez para analisar os resultados. Uma tecnologia de fluorescência detecta o crescimento bacteriano e a ocorrência de alterações metabólicas nas microcavidades dos cartões (Franzolin, 2015).
Outra opção de equipamento automatizado é o MicroScan da Beckman Coulter, sua versão WalkWay 96 plus utiliza duas metodologias colorimétrica e fluorimétrica. O sistema consiste em microplacas (painéis) de 96 poços, impregnados com a série bioquímica para a identificação bacteriana e os agentes antimicrobianos com suas respectivas diluições para a susceptibilidade antimicrobiana com liberação do valor do CIM (Laft, 2023).
Diferentes estudos mostram as vantagens em se utilizar metodologias automatizadas, como o monitoramento contínuo do sistema, maior sensibilidade e rapidez das detecções de amostras positivas, criação de banco de dados, menor risco de contaminação laboratorial, além da economia de tempo e material. Porém, apesar das inúmeras vantagens, os métodos automatizados ainda possuem um custo financeiro elevado, e o custo-benefício só poderá ser avaliado pelo laboratório contratante (Amaral; Barbosa; Correia, 2017).
2.3.4. Testes moleculares e espectrometria de massa
A implantação de métodos moleculares já é uma realidade para a detecção de resistência antimicrobiana, e surgiu na intenção de otimizar o tempo dos resultados liberados (Endimiani et al, 2016).
A PCR consiste em realizar a amplificação de fragmentos específicos do DNA. Para isso, utiliza-se um primer ou iniciador que na presença da enzima DNA polimerase amplifique a sequência do DNA. A partir desse método, foram elaboradas variações para sua utilização, como o PCR em tempo real (qPCR), que permite quantificar o gene de interesse. Foi também desenvolvido o Multiplex PCR, esse utiliza mais de um primer para selecionar, e assim leva à amplificação de vários genes, facilitando a detecção de mais de um genótipo em um único processamento (Martinez; Taddei, 2015).
Normalmente, para a detecção de genes de bactérias multirresistentes, utilizase o Multiplex qPCR, com primers de diferentes genes de BMR. A amostra utilizada provém de uma colônia isolada de um crescimento em meio de cultura sólido (Pais et al, 2022).
A espectrometria de massa foi utilizada primeiramente em 1970, mas sem aplicabilidade na rotina microbiológica. A evolução se deu, quando a matriz utilizada para ionizar proteínas foi mudada para ionizar proteínas ribossomais, que são mais bem conservadas do que as de superfície, o que leva a uma mais rápida identificação de microrganismos (Oplustil, 2019).
A técnica se inicia nas bactérias originadas de uma cultura, que serão adicionadas a uma matriz fornecida pelo fabricante em determinada área da lâmina de teste. A lâmina é inserida no espectrômetro de massa, onde o material é ativado por um laser. A matriz irá absorver grande parte da energia emitida pelo laser, e irá convertê-la em calor. O calor vaporiza a parte externa do espécime (Procop, 2018).
As moléculas irão avançar por um espaço a vácuo com diferentes taxas de progressão, dependendo da razão massa:carga (m/z). Esse “tempo de voo” (time of flight) será determinado pela velocidade da chegada de diferentes moléculas ao detector. A soma dos tempos de voo de todas as moléculas presentes irá gerar um espectro. Logo após, esse mesmo espectro será comparado eletronicamente com todos os outros adicionados a um banco de dados, de forma a encontrar a melhor correspondência, que ao final identificará a cepa microbiana (Procop, 2018).
O tamanho e a sequência das proteínas ribossomais são bem específicas nas diferentes espécies, o que permite identificar e diferenciar as diferentes cepas (Hou et al, 2019).
Uma das principais vantagens do uso do MALDI-TOF é a economia de tempo, pois a identificação bacteriana poderá ser realizada em menos de uma hora, em vez de 24 a 48 horas (Croxatto; Prod’hom; Greub, 2012). Portanto, os benefícios da técnica resultam em estadias hospitalares mais curtas, principalmente na UTI (TorresSangiao; Rodriguez; García-Riestra, 2021).
A escolha do método de identificação de colonizados em hospitais depende dos patógenos prevalentes, das estratégias do serviço de controle de infecção hospitalar, da estrutura dos laboratórios de microbiologia clínicos e dos recursos disponíveis para monitoramento dessas vigilâncias (Cury, 2021).
2.4 MICRORGANISMOS RESISTENTES
Segundo a ANVISA alguns dos principais microrganismos investigados em culturas de vigilância para prevenção de MDR são Acinetobacter baumanni, Pseudomonas aeruginosa, Staphylococcus aureus resistente à meticilina (MRSA), Enterobactérias produtoras de β-lactamase de espectro estendido (ESBL), Enterobactérias produtoras de carbapenemases (KPC), Enterococcus spp. resistentes à vancomicina (VRE) (Brasil, 2021).
2.4.1 Acinetobacter baumannii
O gênero de bactérias Acinetobacter é atualmente responsável por cerca de 9% das infecções nosocomiais envolvendo Gram-negativos, sendo a maioria relacionada com o trato respiratório (Joly-Guillou, 2005). A pneumonia associada à ventilação e as infecções da corrente sanguínea apresentam algumas das maiores taxas de mortalidade e morbidade (Dijkshoorn; Nemec; Seifert, 2007). Devido ao aumento de sua incidência, o Acinetobacter baumannii foi classificado pela Infectious Diseases Society of America como um dos seis microrganismos multirresistentes mais problemáticos em termos de saúde pública e preocupação clínica (Talbot et al, 2006).
A. baumannii está frequentemente associado a pacientes submetidos a procedimentos cirúrgicos, cateteres intravasculares e ventiladores mecânicos, porém o fator mais relevante é tempo de internação (Peleg; Seifert; Paterson, 2008). Outros fatores de risco também incluem o etilismo, tabagismo e doenças pulmonares crônicas (Talbot et al, 2006).
Logo, tem se aumentado o interesse para entender seu mecanismo de resistência, já que o A. baumannii tem uma facilidade de adquirir e aglomerar resistência a fármacos, o que junto da sua capacidade de sobreviver por longos períodos em ambiente hospitalar leva ao aumento de surtos por MDR e de novas cepas multirresistentes, fazendo com que antibióticos que já foram a base do tratamento, como os carbapenêmicos, não sejam mais a principal escolha (Kempf; Rolain, 2012).
Por conseguinte, nota-se que com o tempo o A. baumannii acumulou uma diversa gama de resistência antimicrobiana, e grande parte das vezes por transferência horizontal de genes (Adams et al, 2008). Mesmo que tenha outros diversos mecanismos de resistência, como a modificação do alvo do antibiótico e bombas de efluxo (Fraimow; Tsigrelis, 2011).
2.4.2 Pseudomonas aeruginosa
Assim como o A. baumannii, a P. aeruginosa também faz parte da classe de bactérias Gram-negativas não fermentadoras, sendo encontradas no solo, na água e com alta frequência em ambientes de assistência à saúde como UTI, em superfícies e equipamentos, sendo capaz de colonizar tanto pacientes como profissionais da saúde. Por causa dessa capacidade, ela é a bactéria Gram-negativa não fermentadora mais comum nesses ambientes. (Fraimow; Tsigrelis, 2011).
Além disso, a P. aeruginosa é intrinsecamente resistente a muitos antimicrobianos devido aos seus mecanismos de resistência, como a produção da enzima metalobeta-lactamase (MBL), que é uma carbapenemase de classe B com função de degradar enzimaticamente os carbapenêmicos, utilizando o zinco como cofator enzimático, que possui a capacidade de hidrolisar todos os β-lactâmicos, exceto os monobactâmicos (Fraimow; Tsigrelis, 2011; Bush; Jacoby 2010).
2.4.3 Staphylococcus aureus resistente à meticilina (MRSA)
Após ser descoberto na década de 1880 o Staphylococcus aureus, foi classificado como uma bactéria potencialmente patogênica, identificada como causa de lesões cutâneas menores e infecções pós-operatórias chegando à marca de 80% de mortalidade para indivíduos colonizados, anterior ao advento da penicilina em 1940 (Deurenberg; Stobberingh, 2008).
Cerca de apenas dois anos após, foi encontrado a primeira cepa de S. aureus resistente a penicilina, se tornando mais comum com o passar do tempo. Já em 1959, foi liberado para o uso a meticilina e no mesmo período, após dois anos, foi desenvolvido resistência à meticilina pela aparição do gene mecA que codifica uma proteína de ligação à penicilina (PBP2a). Clones associados a hospitais surgiram, logo após os clones associados à comunidade se espalharem pelo mundo (Deurenberg; Stobberingh, 2008).
O Staphylococcus aureus é um patógeno oportunista atualmente faz parte da flora comensal da pele em cerca de 30% a 50% da população, sendo essa a principal bactéria Gram-positiva causadora de infecções nosocomiais. Sua cepa mais comum dificilmente causa patologias em indivíduos saudáveis, porém suas cepas resistentes a múltiplos antibióticos, como é o caso da MRSA, pode causar lesões cutâneas, pneumonia aguda, osteomielite e sepse (Poupel, 2018).
Por outro lado, a MRSA associada a comunidade possui o cromossomo IV do cassete estafilocócico curto, limitando seus genes que conferem resistência a antimicrobianos. Assim, mesmo que ela produza a leucocidina Panton-Valentine, um fator de virulência associado a infeções, essa cepa ainda é suscetível a antibióticos não-β-lactâmicos (Talbot et al, 2006).
2.4.4 Enterobactérias produtoras de β-lactamase de espectro estendido (ESBL)
Envolvendo as bactérias das famílias Enterobacteriaceae, as ESBL são microrganismos Gram-negativos que aparecem com maior frequência no trato gastrointestinal, no trato urinário, no trato biliar, em pneumonia adquirida em ambiente hospitalar, em meningite pós-operatória e feridas decorrentes de traumas (Talbot et al, 2006). São redutoras de nitrato, fermentam a glicose com prova de oxidase negativa e não purulentas (Medeiros, 2018).
Sendo assim, também estão presentes na lista dos seis microrganismos mais problemáticos da Infectious Diseases Society of America (Talbot et al, 2006). tendo em 1983 o primeiro caso relatado de β-lactamases capazes de hidrolisar cefalosporinas de espectro estendido, ficando conhecidas como as β-lactamases de Epectro Estendido (ESBL) e desde então foram descritas em todo o mundo (Arnold et al, 2012).
Por conta disso, entre as β-lactamases, a ESBL é uma das que gera uma maior preocupação atualmente, pois além de conseguir impedir a ação dos antibióticos βlactâmicos também conseguem atingir os de amplo espectro como as cefalosporinas de primeira a terceira geração, tão quanto os monobactâmicos e as penicilinas (Miranda, 2018).
2.4.5 Enterobactérias produtoras de carbapenemases (KPC)
A enzima carbapenemase, assim como as ESBL, é uma β-lactamase, que possui uma gama maior de antibióticos β-lactâmicos que é capaz de degradar. Atualmente, ela está dividida em três classes: as Metalo-β-Lactamases (MBL), do tipo Resistente a Imipenem, o tipo Metallo-β-lactamase codificado por integron de Verona ou o tipo New Delhi Metallo-β-lactamase; as OXA-carbapenemases; e as carbapenemases do tipo KPC, que são as mais relevantes, devido ao seu alto poder de disseminação (Miranda, 2018).
A enzima carbapenemase teve seu primeiro relato nos EUA em 1996 com um isolado de Klebsiella pneumoniae, originando o nome Klebsiella pneumoniae produtora de carbapenemase (KPC) porém essa enzima não é exclusiva da Klebsiella, podendo ser encontrada em diversas enterobactérias como na Enterobacter, Escherichia, Serratia, Citrobacter, Salmonella, Proteus e Morganella (Seibert et al, 2014).
Com o aumento da quantidade de organismos que adquiriram a capacidade de produzir essa enzima, se ocasiona um problema na saúde pública, já que carbapenêmicos como, imipenem, meropenem e ertapenem eram a principal escolha de tratamento para os casos graves de infecção por ESBL, o que diminui a quantidade de alternativas de tratamento (Oliveira; Stranieri, 2011).
2.4.6 Enterococcus ssp. resistente à vancomicina (VRE)
Enterococcus spp são bactérias Gram-positivas de catalase negativa, comuns no trato gastrointestinal, nas mucosas oral e vaginal e na pele. Os Enterococcus Resistentes à Vancomicina (VRE), são conhecidos como bactérias multirresistentes, da qual podem causar infecções oportunistas e possuem uma grande capacidade de se propagar no ambiente hospitalar (Distrito Federal, 2022).
Dentre os principais fatores de risco associados a infecções por VRE podemos observar pacientes com doenças de base severas, imunossuprimidos, pacientes com hipoalbuminemia e aqueles com prolongado tempo de internação, recebendo múltiplos antibióticos (Tacconelli; Cataldo, 2008).
Esses cocos possuem, de maneira intrínseca, resistência a diversos grupos de antibióticos como, cefalosporinas, clindamicina e aminoglicosídeos, e de maneira extrínseca por transmissão plasmidial ou por transposons, cluster do gene vanA. mudando o local de atuação e enfraquecendo a ligação com a vancomicina e diminuindo a eficácia do fármaco, já que o mesmo, terá dificuldades de bloquear a síntese da parede celular (Fraimow; Tsigrelis, 2011).
Devido a esses mecanismos, o Enterococcus faecalis é a espécie com maior prevalência em infecções humanas por Enterococcus, sendo encontrado em cerca de 80% a 90% dos casos, porém o E. faecium é o que tem uma maior quantidade de relatos de resistência à vancomicina (Distrito Federal, 2022).
2.5. CONDUTAS PARA CONTER A RESISTÊNCIA MICROBIANA
A Organização Mundial da Saúde (OMS) indicou, que entre as 10 principais ameaças à saúde pública mundial, está a resistência antimicrobiana (RA). O custo financeiro elevado para a economia dos países e seus sistemas de saúde também são um grande problema, pois a RA afeta a produtividade de pacientes quando as internações hospitalares se tornam longas, fato que encarece o tratamento e muitas vezes leva à incapacitação ou a morte (OMS, 2021).
Posto isso, a OMS em conjunto com os países membros da Organização das Nações Unidas, incluindo o Brasil, se mantem estimulando a elaboração de planos nacionais que contenham ações para o combater e conter a propagação da resistência microbiana, dentre as quais encontra-se a implementação de programas para o uso racional de antimicrobianos na saúde humana e animal. Sendo assim, a implementação de um Programa de Gerenciamento aos Antimicrobianos (PGA), tem a finalidade de prevenir, e controlar a seleção de BMR, além de maximizar os efeitos terapêuticos e minimizar algumas consequências do uso irracional (Brasil, 2023).
Existem componentes essenciais para a elaboração e implementação de um PGA de qualidade, que incluem: apoio de lideranças, definição de responsabilidades, educação, ação para melhorar o uso de antimicrobianos, monitoramento e divulgação dos resultados. O PGA reduz de forma comprovada o uso inadequado dos antimicrobianos, pois identifica prescrições incorretas, o que aumenta a segurança de pacientes tanto ambulatoriais quanto internados (Brasil, 2023).
Dessa maneira, os laboratórios de microbiologia possuem grande relevância e importância e devem redigir um manual de exames microbiológicos com informações sobre coleta, transporte e armazenamento das amostras biológicas, além de treinar os profissionais que atuam nessa área. (Brasil, 2023).
3 MATERIAIS E MÉTODOS
Para alcançar o objetivo anteriormente proposto pelos autores, o presente estudo realizou uma pesquisa de forma descritiva de cunho bibliográfico. Foi realizada uma revisão de literatura narrativa para responder à pergunta de pesquisa “Como é realizada a cultura de vigilância epidemiológica em UTI adulto e quais são os fatores de risco, implicações clínicas e as medidas de controle adotadas?”.
Para tal, foi utilizada a base de dados digital americana PubMed. A busca foi executada utilizando descritores encontrados no site digital da DECS/MESH, site de descritores em ciências da saúde, disponível na plataforma de buscas Google. Os descritores encontrados que mais se assemelhavam as palavras da pergunta de pesquisa e utilizados foram: “Unidades de Terapia Intensiva” e seus sinônimos.
“Conduta Expectante” e seus sinônimos e “Farmacorresistência Bacteriana Múltipla” e seus sinônimos, respectivamente.
Após selecionar os principais descritores em português e inglês e suas sinonímias, os autores acessaram a base de dados PubMed para realizar a pesquisa.
Foi utilizado o operador boleando “AND” que em inglês significa “E”, e serve para indicar para a base de dados que a busca de artigos científicos deve ser realizada com os termos “A” E o termo “B” E os termos “C”, nos campos de busca, para adicionar os três descritores em sequência na pesquisa.
Na primeira busca, realizada na data de 25 de agosto do ano de 2023, no horário de meio-dia e trinta e dois minutos, de Brasília, foram encontrados trinta e dois artigos publicados na base de dados dos anos de 2007 a 2022. Os artigos foram analisados pelos seus títulos e resumos por dois dos autores, e foram aplicados os filtros de seleção, exclusão e inclusão escolhidos pelos também pelos autores. Os critérios de exclusão se baseiam em, se o estudo ocorreu em UTI neonatal (critério 1); se o estudo não utilizou cultura de vigilância (critério 2); se o estudo não se passa em ambiente UTI (critério 3); se o artigo não responde a pergunta da pesquisa (critério 4); se o artigo trata-se de uma revisão integrativa (critério 5); se eram artigos pagos (critério 6) e um artigo excluído após leitura por não conter detalhes das condutas laboratoriais (critério 7) (Figura 1).
Além disso, a triagem inicial por títulos e resumos e a identificação dos estudos que atenderam aos critérios de inclusão e exclusão foram feitas por dois autores. Os estudos que atenderam aos critérios de inclusão foram submetidos à revisão completa do texto por um revisor independente. Qualquer desacordo foi resolvido por esse terceiro. Dessa forma, dos trinta e dois artigos filtrados inicialmente, apenas oito foram incluídos para a realização dos resultados, após os critérios citados.
Figura 1- Estrutura da seleção de artigos
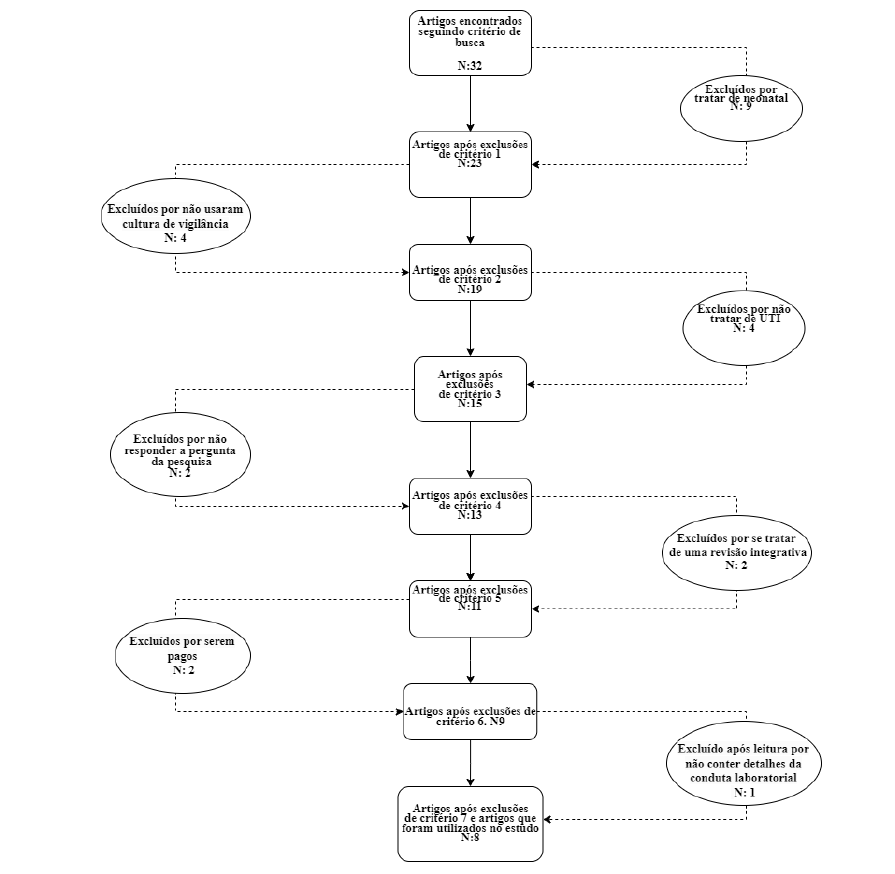
Elaborado pelos autores, (2023).
4 RESULTADOS
Para responder à pergunta norteadora do estudo, foi realizada a leitura e análise dos oito artigos selecionados anteriormente. Logo após, foram produzidas tabelas, para a coleta e síntese dos dados obtidos, com a finalidade de organizar de forma estruturada as informações obtidas e analisadas.
Na tabela 1, os artigos foram agrupados seguindo um roteiro esquematizado com as seguintes informações: autor e ano de publicação, local de realização do estudo, título original do artigo, delineamento da pesquisa, e objetivos dos estudos.
Os artigos foram enumerados por ordem de publicação, do mais recente para o mais antigo. São eles dos anos, 2020, 2019, 2018, 2017, 2013, 2011, 2008, e 2007, todos em língua inglesa. Quanto ao local de realização, encontrou-se os países: Marrocos, Coréia do Sul, Polônia, Estados Unidos, China, Espanha e Tailândia. A população abordada nos oito artigos foram pacientes admitidos em Unidades de Terapia Intensiva adulta, pacientes maiores de 18 anos.
Tabela 1 – Apresentação dos artigos
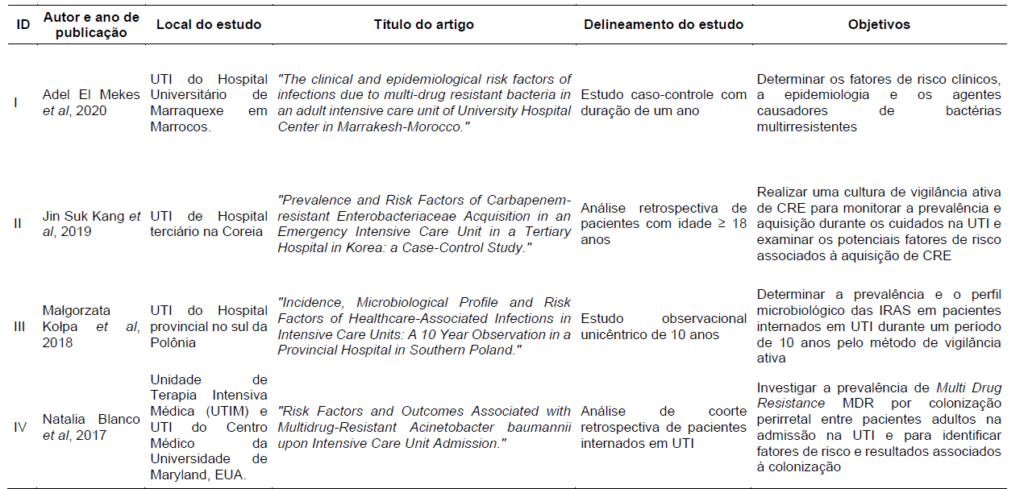
Fonte: Elaborado pelos autores
(Tabela 1- Continuação)

Fonte: Elaborado pelos autores
Na tabela 2 são apresentadas as análises microbiológicas realizadas nos estudos analisados. Foram utilizados diversos materiais biológicos para as análises microbiológicas. Os artigos II e IV utilizaram amostras perirretais, colhidas em swab e o artigo VI utilizou esfregaços retais (Kang et al, 2019; Blanco et al, 2018). Os artigos I e III foram os que mais utilizaram sítios anatômicos e amostras diferentes para pesquisa de MDR, dentre elas urina, lavado broncoalveolar e broncoaspirados, sangue, ponta de cateteres, esfregaço de feridas, fezes, pus e Líquido Cefalorraquidiano (LRC) (El makes et al, 2020; Kolpa et al, 2018). O artigo V utilizou swabs faríngeos, traqueais, escarro e aspirados endotraqueais, assim como o VIII utilizou também o swab nasal. (Huang et al, 2013; Jones et al., 2007). O artigo VII, utilizou aspirado traqueal e swab retal também (Apisarnthanarak et al, 2008).
Os artigos II, III, IV, e VII, relataram em seus textos o uso de meios de cultura sólidos ou líquidos e especificaram detalhes desses meios para a realização da cultura de vigilância ativa, os demais, a princípio, não informaram o meio de cultura utilizado (Kang et al, 2019; Kolpa et al, 2018; Blanco et al, 2018; Apisarnthanarak et al, 2008).
O sistema VITEK 2 da BioMérieux emprega a metodologia para identificação de turbidez na análise dos resultados, verificando a se possui utilização de açúcares, lipídeos e produção de enzimas. A detecção do crescimento bacteriano e a ocorrência de alterações metabólicas nas microcavidades. A galeria API da da BioMérieux, reúne diferentes reações bioquímicas em vários poços, também uma base de dados de identificação. Esses testes foram utilizados pelos artigos I e III (El makes et al, 2020; Kolpa et al, 2018). O artigo II, fez uso de espectrometria de massa com tempo de voo por dessorção a laser assistida por matriz, um espectro de massa da amostra é analisado e comparado com um banco de dados, fornecendo resultados de identificação em poucos minutos (Kang et al, 2019).
Todos os estudos discorreram sobre testes de identificação e a realização do Teste de Sensibilidade aos Antimicrobianos (TSA), comum entre todos, porém realizados de formas diferentes em cada estudo, podendo ser manuais ou automatizados. Os artigos I, II, IV, utilizaram teste de difusão em disco em placa sólida, teste esse manual e qualitativo (El makes et al, 2020; Kang et al, 2019; Blanco et al, 2018).
Os painéis de identificação MicroScan, utilizam tecnologia de CIM real e metodologia de microdiluição em caldo para fornecer os resultados mais precisos foi utilizado pelo artigo VI, e ambos são testes automáticos semi-quantitativos (Suarez et al, 2011).
Além disso, testes moleculares confirmatórios foram realizados em três artigos, os autores dos artigos II e VIII utilizaram a PCR, e o autor do artigo VI Eletroforese em gel de campo pulsado, ambos com finalidade de identificar o genótipo dos microrganismos isolados, além de traçar os perfis de resistência antimicrobiana (Kang et al, 2019; Jones et al, 2007).
Tabela 2- Análises microbiológicas

Fonte: Elaborado pelos Autores
Tabela 2- Continuação

Fonte: Elaborado pelos autores.
Na tabela 3 são apresentados os resultados das análises microbiológicas. Todos os autores relataram casos de bactérias resistentes a antimicrobianos na UTI adulto, com as mais diversas cepas bacterianas.
Desses estudos, o II, pesquisou apenas a incidência de enterobactérias resistentes a antibióticos da classe de carbapenêmicos (CRE) em uma área não endêmica, da qual o mais isolado foi a Klebsiella pneumoniae com 72,7% do total, além disso o CRE produtor de carbapenemase compreendeu 42,9% dos isolados totais e incluiu os genes bla KPC e bla NDM (Kang et al, 2019).
Três estudos diferentes (IV, V e VII), pesquisaram e isolaram apenas Acinetobacter baumannii com perfil de multirresistência. O artigo V traz que 14,2% dos pacientes desenvolveram pneumonia devido a MDRAB, destes, 27,8% com episódios de Pneumonia Associada a Ventilação (PAV) e 11,1% infecções secundárias da corrente sanguínea. Foi considerada MDR quando detectado resistência a ≥3 classes de antimicrobianos, incluindo cefalosporinas, aztreonam, carbapenêmicos, aminoglicosídeos, fluoroquinolonas e sulbactam (exceto polimixina B) (Blanco et al, 2018; Huang et al, 2013; Apisarnthanarak et al, 2008).
Já o artigo IV discute os fatores de risco com a presença de MDRAB na admissão na UTI adulto, em seus resultados pelo menos 83% dos isolados eram resistentes aos carbapenêmicos e 90% dos isolados eram resistentes a fluoroquinolonas, cefalosporinas, penicilinas antipseudomonas e tetraciclinas. A MDR considerada quando o isolado for resistente a um ou mais agentes em três ou mais categorias de antimicrobianos (Blanco et al, 2018).
Concluindo as MDRAB, o artigo VII busca intervenções para controlar as infecções e diminuir a incidência de isolados positivos para MDR.AB. Para isso, foi implementado a vigilância ativa e a limpeza ambiental como forma de controle, reduzindo a quantidade de infecções de 3,6 casos a cada 1000 pacientes para 0,85 casos a cada 1000 pacientes no final da intervenção, reduzindo também o custo de antibióticos em 36% – 42% e o custo de hospitalização em 25% – 36% mesmo que durante o estudo os critérios de prescrições de antibióticos tenham se mantido (Apisarnthanarak et al, 2008).
O estudo VI pesquisou e isolou somente Pseudomonas aeruginosa. A análise genotípica dos autores revelou que as cepas de P. aeruginosa multirresistente (MRPA) selecionadas, pertenciam a um único clone responsável por um grande surto sustentado, e que os pacientes eram reservatórios de tal bactéria, além de concluírem que a pressão antibiótica desempenhava um papel decisivo, alterando o nicho ecológico e proporcionando uma vantagem de crescimento seletivo para os organismos MRPA (Suarez et al, 2011).
O estudo VIII foi realizado utilizando isolados positivos para MRSA e identificouse que dessas, 13,2% também eram resistentes a mupirocina, o que ocasionava maior índice de mortalidade quando comparado a MRSA sensível a mupirocina. Após ser realizado uma análise molecular dos isolados resistentes ao antibiótico revelou que 72,5% dos isolados continham cassete estafilocócica cromossômica mec II que é compatível com a MRSA relacionado aos cuidados a saúde, e devido a variação de resistência encontrado sugere-se a existência de mais de um clone circulante (Jones et al, 2007).
O estudo I e o III, não pesquisaram e isolaram apenas um único gênero e espécie de microrganismo conhecido, portanto, encontraram resultados mais variados do que os demais (El mekes et al, 2020; Kolpa et al, 2018). O I, isolou em maior quantidade o Acinetobacter baumannii com 31% (Zahlane et al, 2020). Porém outros achados foram também relatados, como Enterobactereacae com 30%, Staphylococcus spp com 24%, Pseudomonas aeruginosa com 10% e outras cepas bacterianas com 5%. Os testes de resistência aos antibióticos revelaram que 40% (124/305) das cepas isoladas eram bactérias MDR. O mais comum foi A. baumannii resistente ao imipenem 70%, seguido por espécies multirresistentes de Enterobactereacae (18%), P. aeruginosa resistente à ceftazidima 7%, e S. aureus resistente à meticilina 5%. Todos os A. baumanni testados foram resistentes aos β-lactâmicos.
Já o estudo III também isolou em sua maioria o Acinetobacter baumannii, caracterizado pelas altas taxas de resistência a muitos grupos de antibióticos e tendência rápida de resistência especialmente para carbapenêmicos. Outras bactérias isoladas por esses pesquisadores foram Klebsiella pneumoniae e Pseudomonas aeruginosa (Kolpa et al, 2018).
Tabela 3- Resultados das análises
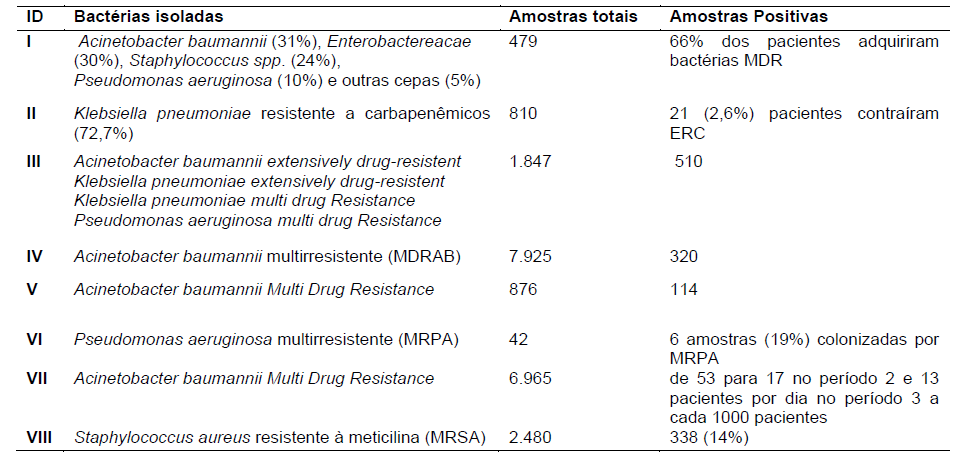
Fonte: Elaborado pelos autores.
5 DISCUSSÃO
A busca por microrganismos resistentes foi realizada de forma específica em seis artigos, e em dois deles de forma abrangente. Essa busca evidenciou a predominância de A. baumannii, sendo encontrado cepas em 62,5% dos estudos em comparação com a P. aeruginosa (25%), MRSA (12,5%) e ERC (12,5%). Essa alta prevalência de A. baumannii pode ser atribuída em parte à frequência de sua ocorrência em ambientes de cuidados intensivos.
Sendo assim, o setor onde foram realizadas as pesquisas deve ser levado em consideração já que esse local, unidade de terapia intensiva, é comumente caracterizado por pacientes debilitados e que necessitam de assistência de alta complexidade, incluindo constantemente o tratamento medicamentoso com antibióticos, e uso de dispositivos muitas vezes invasivos.
Sabendo disso, grande parte dos estudos trouxeram associações importantes entre os fatores de risco e a aquisição da colonização/infecção por BMR. O tratamento com antibioticoterapia, e o seu uso prolongado foi o fator mais comum entre os artigos para elevar as chances de se adquirir BMR, sendo citado nos estudos I, II, IV, VII (El mekes et al, 2020; Kang et al, 2019; Blanco et al, 2018; Apisarnthanarak et al, 2008). A ventilação mecânica usada na UTI, elevou os riscos nos artigos I, III e VII (El mekes et al, 2020; Kolpa et al, 2018; Apisarnthanarak et al, 2008). O histórico de internações prévias, transferência de hospitais ou de setores, e permanências prolongadas em UTI, configuraram o aumento de risco nos artigos I, II, IV e VII (El mekes et al, 2020; Kang et al, 2019; Blanco et al, 2018; Apisarnthanarak et al, 2008). Observou-se que a predominância de IRAS no sexo masculino (II, III), e a alimentação artificial ou nutrição parenteral (I, III) também elevaram os riscos de colonização/infecção (El mekes et al, 2020; Kang et al, 2019; Kolpa et al, 2018).
Determinados estudos trouxeram os relatos sobre mortalidades relacionadas com as colonizações e infecções por BMR. No artigo I a taxa de mortalidade causada por bactérias MDR foi de 12% e no artigo VI a taxa foi de 19% (El mekes et al, 2020; Suarez et al, 2011). O estudo II relata que infecções por CRE estão diretamente associadas com altas taxas de mortalidade, cerca de 40 a 50% (Kang et al, 2019). No estudo VIII a mortalidade hospitalar foi maior para pacientes colonizados com MRSA resistente à mupirocina (33% vs. 16%) (Jones et al, 2007).
O estudo IV trouxe que pacientes positivos para A. baumannii MDR na admissão na UTI tiveram maior probabilidade de morrer durante a hospitalização do que aqueles negativos na admissão concordando com o estudo VII que relatou que A. baumanni pode estar elevando a alta morbimortalidade dos pacientes (Blanco et al, 2018; Apisarnthanarak et al, 2008).
Contradizendo os estudos anteriores, o artigo III demonstrou que nos pacientes com IRAS a taxa de mortalidade foi de 42,5% e no grupo não infectado a taxa de mortalidade foi de 45,3%; essa diferença não foi estatisticamente significativa, porém o mesmo estudo traz que pacientes com IRAS permaneceram na UTI mais tempo do que aqueles sem IRAS em pelo menos 3 vezes mais (Kolpa et al, 2018). Assim, o artigo V chegou a resultados parecidos sobre mortalidade com o artigo III, eles demonstraram que as UTIs são responsabilizadas pela alta prevalência de infecções nosocomiais devido ao MDR.AB e pelo aumento dos custos médicos, porém a mortalidade relacionada não aumentou (Huang et al, 2013; Kolpa et al, 2018).
Além disso, após os achados dos artigos, alguns autores trouxeram em suas pesquisas as condutas após identificação de BMR em seus pacientes internados. Aplicaram-se medidas de higiene hospitalar e uso racional de antimicrobianos (I) e isolamento e precaução de contato imediatas (II, VII) (El mekes et al, 2020; Kang et al, 2019; Apisarnthanarak et al, 2008). O uso correto de EPI, supervisão de procedimentos de limpeza também foram adotados (VI, VII) (Suarez et al, 2011; Apisarnthanarak et al, 2008).
Sete dos oito artigos, destacaram a relevância do uso de cultura de vigilância. Os estudos I e V informam inclusive que a implementação da CVA é mandatória para controle de IRAS por BMR (El mekes et al, 2020; Huang et al, 2013). Os artigos III, IV e VII reforçam a importância desse método de controle para prevenção, controle e até diminuição a longo prazo, dos casos das infecções na UTI adulto (Kolpa et al, 2018; Blanco et al, 2018; Apisarnthanarak et al, 2008). É citado no artigo II que a cultura de vigilância ativa é um método rentável na diminuição de infecções e mortalidade por CRE (Kang et al, 2019). A CVA foi importante para que o resultado do artigo VI fosse alcançado (Suarez et al, 2011). Os autores do artigo IV, relembram inclusive que a CVA é uma abordagem consolidada e apoiada pelas diretrizes do CDC no controle de BMR (Blanco et al, 2018).
6 CONCLUSÃO
A realização da cultura de vigilância é essencial para a triagem e controle de IRAS causadas por bactérias multirresistentes em UTI adulto.
As bactérias A. baumannii, P. aeruginosa, MRSA e ERC foram isoladas e identificadas em meios específicos. Ainda foram utilizados para o TSA, métodos manuais como disco-difusão e métodos automatizados, como exemplo o sistema VITEK, e os métodos moleculares como a PCR e a eletroforese em gel de campo pulsado para a confirmação genotípica da BMR.
Além disso, também foi observado que fatores como a antibioticoterapia, o uso de ventilação mecânica e o elevado tempo de permanência na UTI foram fatores de risco para aquisição de BMR.
A implementação de programas de gerenciamento de antimicrobianos (PGA) em hospitais se torna uma necessidade atual e relevante. O PGA pode ajudar a controlar a disseminação de bactérias multirresistentes e melhorar a qualidade do tratamento em ambientes de UTI adulto, contribuindo para a segurança dos pacientes e a eficácia do sistema de saúde.
Foi percebida a necessidade de novas pesquisas, com objetivo de impulsionar o avanço das boas práticas em saúde, em benefício da população, visando diminuir e evitar a frequência de infecções nosocomiais relacionadas a BMR.
7 REFERÊNCIAS BIBLIOGRÁFICAS
ABRANTES, J. A; NOGUEIRA, J. M. R. Resistência bacteriana aos antimicrobianos:uma revisão das principais espécies envolvidas em processos infecciosos. Revista Brasileira de Análises Clínicas. Rio de Janeiro. 2021. DOI: 10.21877/24483877.202102156. Disponível em: https://www.rbac.org.br/artigos/resistenciabacteriana-aos-antimicrobianos-uma-revisao-das-principais-especies-envolvidas-emprocessos-infecciosos/. Acesso em: 28 set. 2023.
ADAMS, Mark D. et al. Comparative genome sequence analysis of multidrugresistant Acinetobacter baumannii. Journal of bacteriology, v. 190, n. 24, p. 80538064, dez. 2008. DOI: https://doi.org/10.1128/jb.00834-08. Disponível em: https://journals.asm.org/doi/10.1128/jb.00834-08?url_ver=Z39.882003&rfr_id=ori%3Arid%3Acrossref.org&rfr_dat=cr_pub++0pubmed Acesso em: 2 out. 2023.
ALMEIDA, Marília Virgo Silva. Perfil etiológico de cocos gram positivos isolados de cultura de vigilância epidemiológica de resistência. 2016. Monografia (Curso de Biomedicina) – Universidade Federal do Rio Grande do Norte, Natal, 2016.
ALTERTHUM, F. (2015a) Morfologia e Estrutura da Célula Bacteriana. In TRABULSI, L. R.; ALTERTHUM, F. (orgs), Microbiologia. 6. Ed (pp. 7-20). São Paulo, Rio de Janeiro, Belo horizonte: Atheneu.
ALTERTHUM, F. (2015b). Mecanismo de Ação dos Antibacterianos e Mecanismos de Resistência, In TRABULSI, L. R.; ALTERTHUM, F. (orgs), Microbiologia. 6. Ed (pp. 79-86). São Paulo, Rio de Janeiro, Belo horizonte: Atheneu.
AMARAL, P. S; Barbosa, S. R; Correia, S. M. B S.. A importância da automação nos laboratórios de análises clínicas. 2017. Disponível em:https://www.newslab.com.br/wpcontent/uploads/yumpu_files/A%20IMPORT%C3%82NCIA%20DA%20AUTOMA%C%87%C3%83O%20NOS%20LABORAT%C3%93RIOS%20DE%20AN%C3%81LISES%20CL%C3%8DNICAS.pdf. Acesso em: 29 set. 2023.
ANDRADE, D; LEOPOLDO, C. V; HASS, J. V.Ocorrência de bactérias multiresistentes em um centro de terapia intensiva de hospital brasileiro de emergências. Revista Brasileira de Terapia Intensiva. v. 18, n. 1, jan. Disponível em: https://www.scielo.br/j/rbti/a/bywVYGqdfYvSnR4QnFwk54s/?format=pdf&lang=pt. Acesso em: 2 out. 2023.
APISARNTHANARAK, Anucha et al. A multifaceted intervention to reduce pandrugresistant Acinetobacter baumannii colonization and infection in 3 intensive care units in a Thai tertiary care center: a 3-year study. Clinical Infectious Diseases, v. 47, n.6, p. 760-767, set. 2008. DOI: https://doi.org/10.1086/591134. Disponível em:https://academic.oup.com/cid/article/47/6/760/325207?login=false. Acesso em: 25 ago. 2023.
ARAÚJO, Beatriz Torres et al. Políticas para controle de Infecções Relacionadas à Assistência à Saúde (IRAS) no Brasil, 2017. 2017. Disponível em: https://bvsms.saude.gov.br/bvs/periodicos/ccs_artigos/v28_3_politica_controle_%20i nfeccao.pdf. Acesso em: 08 ago. 2023.
ARNOLD, Ryan S. et al. Emergence of Klebsiella pneumoniae carbapenemase (KPC)-producing bacteria. Southern medical journal, v. 104, n. 1, p. 40, jan. 2011.DOI: 10.1097/SMJ.0b013e3181fd7d5a. Disponível em:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3075864/?report=reader#__ffn_sectitl. Acesso em: 25 ago. 2023.
BLANCO, Natalia et al. Risk factors and outcomes associated with multidrugresistant Acinetobacter baumannii upon intensive care unit admission. Antimicrobial agents and chemotherapy, v. 62, n. 1, p. 10.1128/aac. 01631-17, dez. 2018. DOI:https://doi.org/10.1128/aac.01631-17. Disponível em: https://journals.asm.org/doi/full/10.1128/aac.01631-17. Acesso em: 10 set. 2023.
BRASIL. Agência Nacional de Vigilância Sanitária. Prevenção de infecções por microrganismos multirresistentes em serviços de saúde – Série Segurança do Paciente e Qualidade em Serviços de Saúde/Agência Nacional de Vigilância Sanitária. Brasília, 2021. Disponível em: https://www.gov.br/anvisa/ptbr/centraisdeconteudo/publicacoes/servicosdesaude/publicacoes/manual-prevencaode-multirresistentes7.pdf. Acesso em: 3 out. 2023.
BRASIL. Agência Nacional de Vigilância Sanitária. Microbiologia Clínica para o Controle de Infecção Relacionada à Assistência à Saúde. Módulo 4: Procedimentos Laboratoriais: da requisição do exame à análise microbiológica e laudo final/Agência Nacional de Vigilância Sanitária. Brasília. Anvisa, 2013.Disponível em: https://www.saude.go.gov.br/images/imagens_migradas/upload/arquivos/201702/modulo-4—procedimentos-laboratoriais—da-requisicao-do-exame-a-analisemicrobiologica-e-laudo-final.pdf. Acesso em: 3 out. 2023.
BUSH, Karen; JACOBY, George A. Updated functional classification of βlactamases. Antimicrobial agents and chemotherapy, v. 54, n. 3, p. 969-976, mar.2010. DOI:https://doi.org/10.1128/aac.01009-009. Disponível em:https://journals.asm.org/doi/full/10.1128/aac.01009-09 Acesso em: 3 de out. 2023.
CARROLL, Karen C.(2014). Quimioterapia Antimicrobiana. In BROOKS, G. F. et al. Microbiologia Médica de Jawetz, Melnick & Adelberg. 26. ed. (pp. 371-406) Porto Alegre: AMGH.
CURY, Ana Paula. (2021). Avaliação de método molecular para detecção de colonização por carbapenemases comparado com a cultura de vigilância diretamente do swab retal. Tese de Doutorado, Universidade de São Paulo, São Paulo, Brasil.
CFM. Resolução Nº 2.271 de 14 de fevereiro de 2020. Diário Oficial da União, 14 de fevereiro de 2020. Disponível em: https://www.in.gov.br/en/web/dou/-/resolucao-n2.271-de-14-de-fevereiro-de-2020- 253606068. Acesso em: 30 out. 2023.
CHRISTAKI, Eirini; MARCOU, Markella; TOFARIDES, Andreas. Antimicrobial Resistance in Bacteria: Mechanisms, Evolution, and Persistence Journal of Molecular Evolution p.26-40 pub 2019 Oct 28 DOI: 10.1007/s00239-019-09914-3.Disponível em: https://link.springer.com/article/10.1007/s00239-019-09914-3#citeas. Acesso em: 10 set. 2023.
CROXATTO, Antony; PROD’HOM, Guy; GREUB, Gilbert. Applications of MALDITOF mass spectrometry in clinical diagnostic microbiology. FEMS microbiology reviews, v. 36, n. 2, p. 380-407,Mar. 2012. DOI: https://doi.org/10.1111/j.15746976.2011.00298.x. Disponível em: https://academic.oup.com/femsre/article/36/2/380/565595. Acesso em: 30 out. 2023.
DECK, Daniel H.; WINSTON, Lisa G. (2017a). Antibióticos β-lactâmicos e outros antibióticos ativos na parede e membrana celularIn KATZUNG, B. G.; TREVOR, A.J. Farmacologia Básica e Clínica. 13. ed. (pp. 769-787) Porto Alegre: AMGH.
DECK, Daniel H.; WINSTON, Lisa G. (2017b).Aminoglicosídeos e espectinomicinaIn KATZUNG, B. G.; TREVOR, A.J. Farmacologia Básica e Clínica. 13. ed. (pp. 799-806) Porto Alegre: AMGH.
DEURENBERG, Ruud H.; STOBBERINGH, Ellen E. The evolution of Staphylococcus aureus. Infection, genetics and evolution, v. 8, n. 6, p. 747-763, dez. 2008. DOI: https://doi.org/10.1016/j.meegid.2008.07.007. Disponível em: https://www.sciencedirect.com/science/article/abs/pii/S156713480800141X. Acesso em: 2 out. 2023.
DIJKSHOORN, Lenie; NEMEC, Alexandr; SEIFERT, Harald. An increasing threat in hospitals: multidrug-resistant Acinetobacter baumannii. Nature reviews microbiology, v. 5, n. 12, p. 939-951, dez. 2007. DOI: https://doi.org/10.1038/nrmicro1789. Disponível em:https://www.nature.com/articles/nrmicro1789. Acesso em: 29 set. 2023.
DISTRITO FEDERAL. IGESDF. Manual Para Tratamento de Bactérias Gram Positivas. brasília, 2022. Disponivel em: https://igesdf.org.br/manual-paratratamento-de-bacterias-grampositivas/#:~:text=As%20principais%20esp%C3%A9cies%20de%20enterococos,de %20escolha%20na%20pr%C3%A1tica%20cl%C3%ADnica. Acesso em: 28 set. 2023.
DISTRITO FEDERAL. Secretaria de saúde do Distrito Federal. Orientações para medidas de prevenção e controle de bactérias multirresistentes na execução do plano de enfrentamento da resistência bacteriana nas áreas críticas dos hospitais públicos do GDF. Brasília, 2015. Disponível em:https://www.saude.df.gov.br/documents/37101/65181/NOTA-TECNICA-N%C2%B0-01-2015-GERIS-BMR.pdf/09177b41-785e-c5ad-3b95fc72e3892142?t=1648481372199. Acesso em: 25 set. 2023.
ENDIMIANI, A. e JACOBS, M. R. The Changing Role of the Clinical Microbiology Laboratory in Defining Resistance in Gram-negatives. Infect Dis Clin North Am, 2016 Jun 30(2) (p- 323-345.) Disponível em: https://www.sciencedirect.com/science/article/abs/pii/S0891552016300083?via%3Di hub. Acesso em: 27 out. 2023.
EL MEKES, Adel et al. The clinical and epidemiological risk factors of infections due to multi-drug resistant bacteria in an adult intensive care unit of University Hospital Center in Marrakesh-Morocco. Journal of Infection and Public Health, v. 13, n. 4, p. 637-643, apri. 2020. DOI: https://doi.org/10.1016/j.jiph.2019.08.012. Disponível em: https://www.sciencedirect.com/science/article/pii/S1876034119302928. Acesso em: 25 ago. 2023.
FRAIMOW, Henry S.; TSIGRELIS, Constantine. Antimicrobial resistance in the intensive care unit: mechanisms, epidemiology, and management of specific resistant pathogens. Critical care clinics, v. 27, n. 1, p. 163-205, jan. 2011. DOI: https://doi.org/10.1016/j.ccc.2010.11.002. Disponível em: https://www.criticalcare.theclinics.com/article/S0749-0704(10)00099-0/fulltext#tbl5. Acesso em: 12 out. 2023.
FRANCO, Mayara Maria Bastos. Etiologia e resistência bacteriana em unidades de terapia intensiva através de culturas de vigilância. 2017. Dissertação (Mestrado em Ciências Biológicas) – Centro de Biociências, Universidade Federal do Rio Grande do Norte, Natal, 2017.
FRANZOLIM, Marcia Regina. Fundamentos da Identificação Bioquímica das Bactérias. In TRABULSI, Luiz Rachid; ALTERTHUM, Flavio. Microbiologia (pp. 525- 539). São Paulo. Atheneu.
HOU, Tsung-Yun; CHIANG-NI, Chuan; TENG, Shih-Hua. Current status of MALDITOF mass spectrometry in clinical microbiology. Journal of food and drug analysis, v. 27, n. 2, p. 404-414, abr. 2019. DOI: https://doi.org/10.1016/j.jfda.2019.01.001 Disponivel em: https://www.sciencedirect.com/science/article/pii/S1021949819300146 Acesso em: 30 out. 2023.
HOLMES, Alison H et al. Understanding the mechanisms and drivers of antimicrobial resistance The Lancet Journal v. 387 p.176-187 jan.2016 DOI: http://dx.doi.org/10.1016/S0140-6736(15)00473-0 Disponível em: https://spiral.imperial.ac.uk/handle/10044/1/32225 Acesso em: 09 set. 2023.
HUANG, Jie et al. Sources of multidrug-resistantAcinetobacter baumanniiand its role in respiratory tract colonization and nosocomial pneumonia in intensive care unit patients. Chinese medical journal, v. 126, n. 10, p. 1826-1831, mai. 2013. DOI:
10.3760/cma.j.issn.0366-6999.20122358. Disponível em: https://journals.lww.com/cmj/fulltext/2013/05200/sources_of_multidrug_resistantacine tobacter.6.aspx. Acesso em: 25 ago. 2023.
JOLY-GUILLOU, M.-L. Clinical impact and pathogenicity of Acinetobacter. Clinical microbiology and infection v. 11, n. 11, p. 868-873, nov. 2005. DOI: https://doi.org/10.1111/j.1469-0691.2005.01227.x. Disponível em: https://www.clinicalmicrobiologyandinfection.com/article/S1198-743X(14)626008/fulltext. Acesso em: 01 out. 2023.
JONES, Jeffrey C. et al. Mupirocin resistance in patients colonized with methicillinresistant Staphylococcus aureus in a surgical intensive care unit. Clinical Infectious Diseases, v. 45, n. 5, p. 541-547, set. 2007. DOI: https://doi.org/10.1086/520663.Disponível em: https://academic.oup.com/cid/article/45/5/541/273805?login=false. Acesso em: 25 ago. 2023.
JORGENSEN, J.H. Laboratory issues in the detection. reporting antimicrobial resistance. Infectious Diseade Clinics os North America. Texas. v. 11, n. 4, dez.1997.DOI: 10.1016/s0891-5520(05)70390-1. Disponível em: https://pubmed.ncbi.nlm.nih.gov/9421700/. Acesso em: 02 out. 2023.
KANG, Jin Suk et al. Prevalence and risk factors of carbapenem-resistant Enterobacteriaceae acquisition in an emergency intensive care unit in a tertiary hospital in Korea: a case-control study. Journal of Korean medical science, v. 34, n. 18, mai. 2019. DOI: https://doi.org/10.3346/jkms.2019.34.e140. Disponível em: https://synapse.koreamed.org/articles/1122120. Acesso em: 25 ago. 2023
KEMPF, Marie; ROLAIN, Jean-Marc. Emergence of resistance to carbapenems in Acinetobacter baumannii in Europe: clinical impact and therapeutic options.International journal of antimicrobial agents, v. 39, n. 2, p. 105-114, fev. 2012.DOI: https://doi.org/10.1016/j.ijantimicag.2011.10.004. Disponível em: https://www.sciencedirect.com/science/article/abs/pii/S0924857911004225?via%3Di hub. Acesso em: 29 set. 2023.
KISGEN, Jamie. (2016). Princípios do tratamento antimicrobiano. In WHALEN, K.; FINKEL, R.; PANAVELIL, T. Farmacologia Ilustrada. (pp.471-482). Porto Alegre: Artmed.
KOŁPA, Małgorzata et al. Incidence, microbiological profile and risk factors of healthcare-associated infections in intensive care units: a 10-year observation in a provincial hospital in Southern Poland. International journal of environmental research and public health, v. 15, n. 1, p. 112, jan. 2018. DOI:https://doi.org/10.3390/ijerph15010112. Disponível em: https://www.mdpi.com/16604601/15/1/112. Acesso em: 25 ago. 2023.
LAFT, Comércio de Materiais – Microbiologia. Disponível em: https://laft.com.br/microbiologia.php. Acesso em: 28 out. 2023.
MARTINEZ, Marina B.; TADDEI, Carla R. (2015) Métodos de Diagnóstico In TRABULSI, L. R.; ALTERTHUM, F. (orgs), Microbiologia. 6. Ed (pp. 117-126). São Paulo, Rio de Janeiro, Belo Horizonte: Atheneu.
MARTINO, Marinês Dalla Valle. Métodos para Detecção do Perfil de Sensibilidade das Bactérias aos Antibióticos. In TRABULSI, Luiz Rachid; ALTERTHUM, Flavio. Microbiologia (pp. 525- 539). São Paulo. Atheneu.
MEDEIROS, Brunna Carvalho de. Cultura de vigilância: uma revisão bibliográfica. Natal, 2018. Trabalho de Conclusão de Curso. Universidade Federal do Rio Grande do Norte.
MIRANDA, Flaviane de Freitas Calasans. Mecanismos de resistência alactâmicos em enterobacteriaceae. Belo Horizonte, 2018. Trabalho de Conclusão de Curso. Universidade Federal de Minas Gerais.
MORSE, Stephen; MEITZNER, Timothy A. (2014a). Cultura de microrganismos. In BROOKS, Geo. F. et al., Microbiologia Médica de Jawetz, Melnick e Adelberg (pp. 67-77). Porto Alegre, RS: Lange
MORSE, Stephen, A.; MEITZNER, Timothy, A.(2014b). Estrutura Celular. In BROOKS, G. F. et al. Microbiologia Médica de Jawetz, Melnick & Adelberg. 26. ed. (pp. 11-42) Porto Alegre: AMGH.
MUNITA, Jose M; ARIAS, Cesar A. Mechanisms of Antibiotic Resistance Microbiology Spectrum v.4 n.2 pub. abr. 2016. DOI:https://doi.org/10.1128/microbiolspec.vmbf-0016-2015 Disponível em: https://journals.asm.org/doi/10.1128/microbiolspec.vmbf-0016-2015?url_ver=Z39.882003&rfr_id=ori%3Arid%3Acrossref.org&rfr_dat=cr_pub++0pubmed Acesso em: 09 set. 2023.
O’BRIEN, T. F.; STELLING, J. Integrated Multilevel of the World’s Infecting Microbes And Their Resistance To Antimicrobial Agents. Clinical Microbiology Reviews.
DOI: 10.1128/CMR.00021-10. v. 24, n. 2, p. 281–295, 2011. Disponível em:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3122493/pdf/zcm281.pdf. Acesso em: 5 out. 2023.
OLIVEIRA, Helder Cássio de; STRANIERI,Inês. Nota técnica 001/2011. Centro de informações sobre medicamentos-HUJM. Mato Grosso. Jan. 2011. Disponível em: https://www.gov.br/ebserh/pt-br/hospitais-universitarios/regiao-centro-oeste/hujmufmt/ensino-e-pesquisa/setor-de-gestao-de-pesquisa-e-inovacao-tecnologicasegpit/unidade-de-gestao-da-inovacao-tecnologica-em-saude/centro-deinformacoes-sobre-medicamentos-cim-1/2015/nota-tecnica-kpc.pdf. Acesso em: 12 out. 2023.
OPLUSTIL, Carmen Paz, et al. (2019). Procedimentos básicos em microbiologia clínica (4a ed.). São Paulo: Sarvier.
ORGANIZACIÓN MUNDIAL DE LA SALUD. Resistencia a los antimicrobianos. [Genebra]: OMS, nov. 2021. Disponível em: https://www.who.int/es/news-room/factsheets/detail/antimicrobial-resistance. Acesso em: 09 out. 2023.
PAIS, Ana Paula L. et al. Monitoring the bacterial resistance profile of healthcare-related infections (HAI) post-training for the phenotypic detection of kpc. DOI: 10.22533/at.ed.1592512201093. Disponivel em: https://www.atenaeditora.com.br/catalogo/artigo-revista/monitoring-the-bacterialresistance-profile-of-health-care-related-infections-hai-post-training-for-thephenotypic-detection-of-kpc. Acesso em: 30 out. 2023.
PADILHA, Gabriel; COSTA, Sergio O. P. (2015) Genética Bacteriana.In TRABULSI, L. R.; ALTERTHUM, F. (orgs), Microbiologia. 6. Ed (pp. 37-50). São Paulo, Rio de Janeiro, Belo horizonte: Atheneu.
POUPEL, Oliver. et al. SpdC, um novo fator de virulência, controla a atividade da histidina quinase em Staphylococcus aureus. PLOS Patógenos, 2018; DOI: 10.1371/journal.ppat.1006917 Disponível em:
https://journals.plos.org/plospathogens/article?id=10.1371/journal.ppat.1006917. Acesso em: 28 set. 2023.
PELEG, Anton Y.; SEIFERT, Harald; PATERSON, David L. Acinetobacter baumannii: emergence of a successful pathogen. Clinical microbiology reviews, v. 21, n. 3, p. 538-582, jul. 2008. DOI: https://doi.org/10.1128/cmr.00058-07. Disponível em: https://journals.asm.org/doi/10.1128/cmr.00058-07?url_ver=Z39.88-2003&rfr_id=ori%3Arid%3Acrossref.org&rfr_dat=cr_pub++0pubmed. Acesso em: 29 set. 2023.
PROCOP, Gary. et al. Diagnóstico microbiológico: texto e atlas 7. ed. – Rio de Janeiro. Guanabara Koogan, 2018.
ROCHA, Lucas. Pesquisadora fala sobre a resistência causada pelo uso indiscriminado de antibióticos. IOC/FIOCRUZ, 2015. Disponível em: https://portal.fiocruz.br/noticia/pesquisadora-fala-sobre-resistencia-causada-pelouso-indiscriminado-de-antibioticos. Acesso em: 08 ago. 2023.
SANTOS, Neusa de Queiroz. A resistência bacteriana no contexto da infecção hospitalar. Texto & Contexto-Enfermagem, v. 13, p. 64-70, 2004. DOI: https://doi.org/10.1590/S0104-07072004000500007. Disponível em:https://www.scielo.br/j/tce/a/KrkXBPPt83ZyvMBmxHL8yCf/?lang=pt. Acesso em: 08 ago. 2023.
SEIBERT, Gabriela et al. Nosocomial infections by Klebsiella pneumoniae carbapenemase producing enterobacteria in a teaching hospital. Einstein (São Paulo), v. 12, p. 282-286, 2014. DOI: 10.1590/S1679-45082014AO3131. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4872936/?report=reader. Acesso em: 28 set. 2023.
SILVA, André Ricardo Araújo et al. Dinâmica da circulação de bactérias multirresistentes em unidades de terapia intensiva pediátrica do Rio de Janeiro.Revista de Epidemiologia e Controle de Infecção, v. 2, n. 2 (p.41-45). 2012. Disponível em: https://www.redalyc.org/pdf/5704/570464024002.pdf. Acesso em: 2 out. 2023.
SUAREZ, Cristina et al. A large sustained endemic outbreak of multiresistant Pseudomonas aeruginosa: a new epidemiological scenario for nosocomial acquisition. BMC infectious diseases, v. 11, p. 1-8, out. 2011. Disponível em: https://link.springer.com/article/10.1186/1471-2334-11-272. Acesso em: 25 ago. 2023.
TACCONELLI, Evelina; CATALDO, Maria A. Vancomycin-resistant enterococci (VRE): transmission and control. International journal of antimicrobial agents, v.31, n. 2, p. 99-106, fev. 2008 DOI: https://doi.org/10.1016/j.ijantimicag.2007.08.026.Disponível em: https://www.sciencedirect.com/science/article/abs/pii/S092485790700516X?via%3Di hub. Acesso em: 29 set. 2023.
TALBOT, George H. et al. Bad bugs need drugs: an update on the development pipeline from the Antimicrobial Availability Task Force of the Infectious Diseases Society of America. Clinical infectious diseases, v. 42, n. 5, p. 657-668, mar 2006.
DOI: https://doi.org/10.1086/503200. Disponível em: https://academic.oup.com/cid/article/42/5/657/316652?login=false. Acesso em: 02 out. 2023.
TAVARES, Walter. (2014). Resistência Bacterianain TAVARES, Walter. (2014). Antibióticos e Quimioterápicos para o Clínico 3. ed. (pp.38-55). São Paulo, Rio de Janeiro, Belo horizonte: Atheneu.
TORTORA, G. J.; CASE, C. L.; FUNKE, B. R. (2016) Fámacos AntimicrobianosIn Microbiologia. 12. ed. (pp. 548-578). Porto Alegre: Artmed Editora.
TORRES-SANGIAO, Eva; RODRIGUEZ, Cristina; GARCÍA-RIESTRA, Carlos. Application and perspectives of MALDI–TOF mass spectrometry in clinical microbiology laboratories. Microorganisms, v. 9, n. 7, p. 1539, jul 2021. DOI:0.3390/microorganisms9071539. Disponível em:https://pubmed.ncbi.nlm.nih.gov/34361974/. Acesso em: 30 out. 2023.
Monografia apresentada ao curso de graduação em biomedicina da Universidade Católica de Brasília como requisito parcial à obtenção do título de Bacharel em Biomedicina
