REGISTRO DOI: 10.5281/zenodo.7991957
Silvia Mara Haluch1,
Flávia Caroline Haluch2,
Johnatan Duarte Lemes3.
RESUMO: O objetivo dessa pesquisa é verificar a presença de Enterobactérias resistentes aos antibióticos largamente utilizados em terapias medicamentosas, em águas subterrâneas, solos, águas de percolação ou nascente, visando investigação de passivo ambiental de aterros hospitalares e afins, com vistas em novo marco regulatório. Os isolados bacterianos foram identificados de acordo com procedimentos internacionais para contagem, identificação e perfil de resistência aos 10 antibióticos terapêuticos comerciais. Foram encontradas Enterobactérias altamente resistentes aos diversos fármacos. Essa presença de Enterobactérias, no local de estudo, evidencia a importância da inserção de parâmetros biológicos para caracterização de passivos ambientais em áreas possivelmente impactadas. Dentre o contexto, ao realizar testes genéticos, podemos apontar sobre nomenclaturas oficiais de duas bactérias, sendo que os testes de incubação para crescimento em 10º C, contribuíram em conjunto com a porcentagem genética de Klebsiella planticola e a Raoultella planticola, e dessa forma, podemos afirmar, que se trata de bactérias independentes e, portanto, coexistem entre si. Dessa forma, a Klebsiella planticola deve ser citada dentre as linhagens de Klebsiella e a Raoultella planticola deve ser mantida na linhagem de Raoultella. A identificação de patógenos nessas áreas traz a tona sobre manejo seguro de resíduos infectantes e auxilia nas futuras decisões de locais que suportem e garantam com as diretrizes de biossegurança, preservação da saúde coletiva e ambiental, com a inserção de parâmetros biológicos nas legislações nacionais e internacionais.
Palavras-Chave: Enterobactérias, Resistência, antibióticos, Klebsiella, Raoultella.
ABSTRACT: The of this work was to verify the resistance of Enterobacteriaceae to antibiotics, widely used in drug therapies. Such compounds are easly found in groundwater, soil, percolation or spring waterBacterial isolates were identified according to international procedures for counting, identification and resistance profiling to 10 commercial therapeutic antibiotics. Enterobacteriaceae are highly resistant to several drugs found in environment. The presence of Enterobacteriaceae in the study site highlights the importance of monitoring such organisms and reveal the importance in include biological parameters to characterize environmental liabilities in potentially impacted areas. Considering that, when carrying out genetic tests, we concluded that the official nomenclatures of two bacteria, and the incubation tests for growth at 10º C, contributed together with the genetic percentage of Klebsiella planticola and Raoultella planticola, therefore, , we can affirm that bacteria are independent, however and coexist with each other. Therefore, Klebsiella planticola must be restored among the Klebsiella lineages and Raoultella planticola must be maintained in the Raoultella lineage. The identification of pathogens in these areas shed lights on the safe management of infectious waste and helps in the future decisions of places that support and guarantee biosafety guidelines, preservation of collective and environmental health, with the insertion of biological parameters in national and international legislation.
Keywords: Enterobacteria, Resistance, antibiotics, Klebsiella, Raoultella.
INTRODUÇÃO
Um dos maiores problemas mundiais desse século na área da saúde humana é a resistência de agentes microbianos. Com o alto uso de antibióticos, tais microrganismos evoluem e adquirem resistência a esses fármacos (XIUJUAN, 2017). Além de chegarem aos compartimentos ambientais, os microrganismos e os fármacos utilizados no tratamento dos pacientes hospitalizados, permanecem nos tecidos de partes anatômicas removidos cirurgicamente e nos corpos de pacientes que vieram a óbito e em todo resíduo envolvido no processo. Ao destinar os resíduos hospitalares (luvas, seringas, curativos, ataduras, partes anatômicas, entre outros) em aterros, essa gama de substâncias e microrganismos é depositada em aterros controlados e muitos, apenas dispostos no solo descoberto (BRASIL, 2019; ANVISA, 2004).
Órgãos ambientais de cada país possuem suas legislações para pesquisa de passivos. Alguns países se tornaram referência internacional, como é o caso da Holanda, que em sua Lista 6530 traz padrões de referência, alerta e intervenção para solo e água subterrânea. Nos Estados Unidos com suas publicações de Resource Conservation, Clean Water Act e EPA–Environmental Protection Agency, o Canadá com CCME – Canadian Council of Ministers of Environment e CCMTR – Canadian Council of Ministers of the Environment atuam intensamente na preservação da qualidade ambiental.
A Lei americana de Controle de poluição da água (L. 92-500), Lei da água limpa (Pub. L. 95- 217) e Clean Water Act Amendments de 1978 (Pub. L. 95) estabelece que os critérios devam ser baseados nos fundamentos científicos e devem conter parâmetros suficientes na proteção para cada uso. Quando se trata de poluentes tóxicos: “… quando há preocupação dos os níveis de poluentes tóxicos, esses devem ser analisados e adotados novos critérios, aos quais se incluem inclusive, biomonitoramento com organismos aquáticos, a fim de garantir a proteção ambiental e humana (EPA, 2015; EPA 40, 2021).
Porém, quando se trata de parâmetros microbiológicos, apenas a EPA estabelece critérios pautados apenas na densidade de Enterococos. Demais países, os critérios de investigação de passivos ainda são totalmente baseados em parâmetros físicos e químicos, sem indicação de parâmetros microbiológicos e ecotoxicológicos (US GEOLOGICAL, 1989; EPA, 1994; RCA,1984).
No Brasil, a Resolução CONAMA 420/2019, estabelece as três etapas que devem compor o gerenciamento de áreas contaminadas: Identificação, Diagnóstico e Intervenção. A etapa de identificação engloba a Avaliação Preliminar de Passivo Ambiental e Avaliação Confirmatória. Esta última tem por objetivo a confirmação de contaminação do ar, água ou solo por substâncias químicas de origem antrópica em concentrações tais que limitem o uso dos recursos naturais da área em avaliação devido ao risco potencial destes à saúde humana. A definição de contaminação desta resolução não contempla a contaminação biológica em passivos ambientais, ainda que esta gere riscos à saúde humana. Porém a definição de perigo nesta resolução envolve ameaça à vida humana, ao meio ambiente e ao patrimônio público/privado por agentes patogênicos e reativos, entre outros agentes contaminantes, no solo ou em águas subterrâneas, mesmo em instalações não operantes (BRASIL, 2019).
MICRORGANISMOS PATOGÊNICOS
As bactérias da família Enterobacteriaceae (Gram negativas) são encontradas na natureza e em diversos materiais biológicos, principalmente no conteúdo do trato intestinal de humanos e animais. São representadas pelos gêneros Klebsiella, Enterobacter, Escherichia, Serratia, Citrobacter, Salmonella, Proteus e Morganella (ALMEIDA et al., 2012; GISKE, 2011). Tais bactérias desenvolveram mecanismos de resistência a fármacos tais como a produção de β-lactamases, enzimas que degradam o anel betalactâmico de forma a inativar o antimicrobiano, impedindo sua atividade, sendo que o grupo de enterobactérias mais preocupante compreende as bactérias betalactamases de espectro estendido – ESBL representado, por exemplo, pela Klebsiella spp (LIMBAGO, 2011).
As carbapenemases codificadas mais prevalentes nas Enterobactérias são expressadas por genes dos grupos A, blaKPC, encontrada na Klebsiella pneumoniae carbapenemase – KPC, grupo B, que são as metalobetalactamase, blaVIM, blaIMP e blaNdm, classificada em A e B, e D, que são as oxacillinase, blaOxa, representada pelo OXA-48 (GISKE, 2011).
Sabe-se atualmente, que as linhagens existentes de Klebsiella pneumoniae já possuem ß-lactamase AmpC que age hidrolisando a cefoxitina, carbapenemase e metalo-carbapenemase (ANVISA, 2003; TORTORA, 2017). KPC, termo associado a espécie de K. pneumoniae, foi constatada pela primeira vez, na Carolina do Norte, em 1996 (SPANU, 2012). As carbapenemases podem ser transmitidas entre bactérias distintas pela transmissão de partes do código genético, ou seja, plasmídeos que se autorreplicam interespécies, e essa transferência horizontal de genes, ocorre facilmente, causando o conhecido fenômeno de resistência microbiana de cefalosporinas, penicilinas, carbapenêmicos e monobactâmicos (ALMEIDA et al., 2012).
A Organização Mundial da Saúde – OMS (2017) realizou uma lista de prioridades de infecções hospitalares com base na resistência e morbimortalidade das mesmas, gerando três categorias. Os gêneros que ganham destaque são Pseudomonas, Acinetobacter e a família Enterobacteriaceae representada por Escherichia coli, Klebsiella pneumoniae, Proteus spp, Serratia spp, Enterobacter spp, Morganella spp e Providencia spp. A Tabela 1 relaciona as principais classes de risco de infecções
Tabela 1– Categorias de prioridades de infecções hospitalares de acordo com a resistência dos microrganismos e morbimortalidade relacionadas a estes
Prioridade Listados Crítica Acinetobacter baumannii resistente à carbapenema Pseudomonas aeruginosa resistente à carbapenema Enterobacteriaceae resistente à carbapenema e produtoras de ESBL. Alta Enterococcus faecium resistente à vancomicina Staphylococcus aureus (MRSA) resistente à meticilina e vancomicina. Helicobacter pylori resistente à claritromicina Campylobacter spp resistente às fluoroquinolonas Salmonella sp resistente às fluoroquinolonas Neisseria gonorrhoeae resistente à cefalosporina e fluoroquinolonas. Média Streptococcus pneumoniae sem sensibilidade à penicilina Haemophilus influenzae resistente à ampicilina Shigella spp resistente às fluoroquinolonas
Os mecanismos de resistência se iniciam no momento do contato com elementos traços dos fármacos dando início ao mecanismo de defesa da bactéria, que busca mudar seu código genético por inclusão de um plasmídeo ou transposons extracromossômico ou pela troca de material através de recombinação entre espécies usando o pillus sexuais ou simples englobamento, além de alterar a permeabilidade celular bloqueando entrada de fármaco, alteração do sítio ativo inativando o fármaco, bomba de efluxo (eliminação imediata por expulsão) e degradação enzimática (hidrólise da substância e outros), sendo inclusive, a chave do sucesso para as Enterobacterias contra ß-lactâmicos (ANVISA, 2004).
MATERIAIS E MÉTODOS
DESCRIÇÃO DO LOCAL DE ESTUDO
Localizado em Curitiba – Paraná, no Contorno Sul, Cidade Industrial, Avenida Juscelino Kubitschek de Oliveira, possui uma área de 92.400 m2 e recebeu aproximadamente 63 mil toneladas de material enquadrado como Resíduos infectantes e outros da área hospitalar de Curitiba e região metropolitana durante pelo menos 16 anos, desde sua abertura em 1989 até 2005. O volume de chorume produzido estimado foi de 0,864 litros por segundo. A geologia local consta um Geositiode grande importância internacional, devido aos achados arqueológicos, composto praticamente por rochas sedimentares conhecidos como formação Guabirotuba.

Ilustração formação Guabirotuba. Fonte: Autores.
O local é rico em água, visto que a menos de 2 km, encontramos uma importante represa de captação de água, o Passaúna e, a menos de 255 m o Rio Barigui, outros córregos perenes, nascentes e exposição freática.
Amostragem
A coleta de água subterrânea no local conhecido como vala séptica onde a probabilidade de ocorrência de bactérias resistentes, foram coletados e executados segundo a técnica de coleta com volume fixo e preservados conforme Standard Methods (APHA, 2017). As coletas das amostras foram divididas em 2 campanhas, sendo que a primeira coleta foi realizada sem esgotamento do poço (purga) e a segunda com o esgotamento do poço. As amostras foram inoculadas recém coletadas dos Poços de Monitoramento. O método utilizado seguiu a metodologia do Standard Methods para pesquisa de quantificação de Enterobactérias (APHA, 2017; OLPLUSTIL,2010).
Testes microbiológicos
Pela confecção de meios de cultura em meio sólido, com os ágares Mac Conkey e EMB, preparados em ambiente asséptico, as amostras são inoculadas por semeadura em estrias simples, no volume de 1 mL de cada amostra. Após semeadura, os meios serão incubados na temperatura de 35º C aproximadamente, por 48 horas. Após o tempo padrão, as placas serão retiradas para contagem e identificação pelas colônias características de coloração, forma celular, mucilagem, tamanho e alteração do meio.
A contagem celular foi realizada em placas com meio de cultura ágar MacConkey que possui na sua composição duas substâncias que inibem o crescimento de bactérias gram-positivas como cristal violeta e ácido biliar, desta forma, é o melhor meio quantificar e isolar culturas de Enterobactérias. O EMB traz características próprias para algumas bactérias, sendo possível, pelo padrão de crescimento, identificá-las e contá-las. (GOODWIN, 1996)
Identificação das bactérias e provas bioquímicas
A identificação conta com padrão de crescimento, coloração, mecanismo bioquímico da bactéria (BROOKS, 2014, TORTORA, 2017, APHA, 2017). As células escolhidas e com o padrão de crescimento no EMB como colônias verde brilhantes (E. coli), colônias com núcleo escuro (Enterobacter sp e Citrobacter sp), colônias rosadas mucóides (Klebsiella sp.), colônias avermelhadas (Serratia sp e R. planticola), colônias transparentes (Proteus sp, Salmonella sp e Shigella sp) e outras em comparações com Cepas ATCC padrões comerciais, realizada coloração de Gram e provas bioquímicas confirmatórias.
Com repiques das bacterias nos tubos contendo os meios, os mesmos foram incubados por 24 horas em 35 a 37º C, atmosfera mesófila. As provas bioquímicas seguiram uma série de testes que visam resultados positivos e negativos para compor um cenário de identificação da bactéria a ser estudada, sendo utilizado um kit de identificação da empresa Newprov®, com os valores tabelados. Foram realizadas colorações de Gram para observar bacilos isolados ou arranjados, como diplobacilos e estreptobacilos.
Teste de resistência
Após quantificadas e identificadas foram seguidos às técnicas de antibiogramas descritas na NCCLS (National Committee for Clinical Laboratory Standards) e CLSI (Clinical and Laboratory Standards Institute) (CLSI, 2011; NCCLS, 2003).
Duas a três colônias das bactérias de cada ponto coletado foram selecionadas e transferidas para tubos de ensaio contendo solução salina a 2,5% estéril. A turbidez foi comparada com uma solução contendo 0,5 McFarland de turbidez, a fim de garantir crescimento uniforme em toda extensão da superfície do meio de cultura Mueller- Hinton (MH). O espalhamento foi realizado pelo auxílio de um swab estéril, pelos movimentos suaves e em toda superfície. Após o espalhamento, foram colocados os discos adquiridos comercialmente e já validados pelo controle de qualidade com cepa padrão de E. coli ATCC 25922. A placa foi incubada por 18 horas em atmosfera comum e na temperatura de 37º C aproximadamente. Os testes foram realizados em duplicata.
Os Antibióticos selecionados para o teste, foram escolhidos levando em conta a CLSI e as prescrições médicas atuais. As substâncias são: CRO 30(ceftriaxone), CLO 30 (Chloramphenicol), SBA 10 e 10 (Ampicillin-sulbactam), CIP 5(Ciprofloxacin), AMI 30 (Amikacin), IPM 10 (Imipenem), AZI 30 (Azithromycin), AMO 10 (Amoxicilina), AMP 10 (Ampicillin), GEN 10 (Gentamicin). Vancomicina foi usado como controle negativo.
Depois de identificadas, os testes de resistência foram realizados e comparados os halos de formação visando a resistência, sensibilidade ou grau intermediário do antibiótico testado com valores tabelados do CLSI (2011). A Tabela 2 mostra o diâmetro padrão dos halos formados pelos antibióticos identificando sua eficiência no combate a bactérias Gram-negativas.
Tabela 2 – Diâmetro dos halos padrão de acordo com a eficiência do princípio ativo no combate a Enterobactérias (CLSI, 2011):
Antibióticos Halo padrão Resistente Intermediário Sensível CRO 30 – ceftriaxone ≤19 20-22 ≥23 CLO 30- Chloramphenicol ≤12 13-17 ≥18 SBA 10 e 10 – Ampicillin-sulbactam ≤11 12-14 ≥15 CIP 5 – Ciprofloxacin ≤20 21-30 ≥31 AMI 30 – Amikacin ≤14 15-16 ≥17 IPM 10 – Imipenem ≤19 20-22 ≥23 AZI 30 – Azithromycin ≤12 – ≥13 AMO 10 – Amoxicilina ≤13 14-16 ≥17 AMP 10- Ampicillin ≤13 14-16 ≥17 GEN 10 – Gentamicin ≤12 13-14 ≥15 VAN- Vancomicina Todas as gram-negativas são resistentes (resistência intrínseca)
Fonte: autores (2021).
Coloração de GRAM
A coloração de Gram é largamente utilizada como caracterização inicial de bactérias com a visualização da forma e arranjo da mesma, usando com auxílio de microscopia. Esse método de coloração foi criado por Hans Gram em 1884, porém até hoje, é utilizada em todos os laboratórios microbiológicos, diferenciado bactérias de gram positiva e negativa, bem como sua estrutura, morfologia e arranjo (BROOKS, 2014, TORTORA, 2017).
Testes moleculares
Para o sequenciamento genético foi usado o equipamento MiSeq Sequencing System Illumina com o método de alto desempenho. Para a montagem do genoma foi utilizado o pipeline A5 com correlações pelo uso do programa IDBA-UD em conjunto com o montador SPADES. As lacunas foram completadas através do programa GMCloser e para averiguar a qualidade das montagens, foi utilizado o programa Prokka. Para a identificação final foi utilizado o programa BLASTn, PROKKA JSpecies, com a melhor performance/hits para ensaio final (Coil et al., 2015; Peng et al., 2012, Bankevich et al., 2012; Kosugi et al., 2015; Richet et al., 2015, APHA, 2017).
RESULTADOS E DISCUSSÃO
Os resultados da contagem celular dos pontos amostrados estão descritos na tabela 3 a seguir:
Tabela 3: Resultados das contagens de coliformes das campanhas 1 e 2
Ponto de coleta (poço de monitoramento) Campanha 1 – sem esgotamento (UFC/ 100 mL) Campanha 2 – com esgotamento (UFC/ 100 mL) Coliformes totais Coliformes fecais Coliformes totais Coliformes fecais 1 2500 1700 9.000 2.500 2 1100 300 5.900 3.800 3 1900 1100 4800 1.100 4 1500 300 1600 800 5 3100 1500 800 300 6 <100 <100 1000 300 7 2600 1900 1100 600 8 5900 3700 <100 <100 9 1000 200 9.500 2.500 Ponto de coleta solos Sedimento de fundo da nascente Solo da nascente Jusante Solo da montante UFC/g UFC/g UFC/g Coliforme fecais 5,0 x105 2,5 x105 3,0×104 Coliformes totais 2,0 x106 5,5 x106 1,9 x105
Pelos resultados, tanto na campanha 1 como na campanha 2, ocorreram poços com ausência de Enterobactérias e no solo depositado no fundo da nascente, ocorreu alta concentração de coliformes fecais.
Comparando os resultados das contagens realizadas e o CONAMA 396: 2008 que “Dispõe sobre a classificação e diretrizes ambientais para o enquadramento das águas subterrâneas e dá outras providências”, conforme a tabela a seguir, notamos que a maioria das amostras estariam acima dos valores máximos permitidos para todos os usos preponderantes da água subterrânea. (BRASIL, 2008).
Tabela 4: Valores de referência para água subterrânea segundo CONAMA 398:2008.
Parâmetro Usos preponderantes da água Usos preponderantes da água Usos preponderantes da água Usos preponderantes da água Consumo humano Dessedentação de animais Irrigação Recreação E. coli Ausentes em 100 mL 200 em 100 mL Não consta 800 em 100 mL Enterococcos Não consta Não consta Não consta 100 em 100 mL Coliformes termotolerantes Ausentes em 100 mL 200 em 100 mL Não consta 1000 em 100 mL
A quantificação realizada mensurou a quantidade de bactérias encontradas na área amostrada, podendo comparar com a legislação nacional vigente e única, que consta a microbiologia, no entanto, a normativa se refere a usos preponderantes de uma água subterrânea. No Brasil (2009), o CONAMA 420, legislação própria para avaliação de passivo ambiental, trazem apenas parâmetros físicos e químicos para diagnósticos. Atualmente, tornando-se uma referência nacional, a Prefeitura Municipal de Curitiba em sua Secretaria de Meio Ambiente, adotou parâmetros ecotoxicológicos e microbiológicos em suas avaliações de áreas possivelmente impactadas de resíduos hospitalares.
Para o perfil de resistência aos antibióticos, após testes bioquímicos ou genéticos, os valores dos halos foram medidos e comparados com a CLSI, obtendo os dados tabelados a seguir:
Tabela 5: Resultados de identificação e resistência dos pontos amostrados.
PontoCampanha 1 Campanha 2 Enterobacteria isolada Perfil de resistência Enterobacteria isolada Perfil de resistência 1 Serratia sp Resistente à:SBA, AZI e AMP. Demais antibióticos sensível. Klebsiella sp Resistente à:ERI, IPM, AMO. Demais antibióticos sensível. Citrobacter sp Resistente à: CRO, CLO, SBA, CIP, AZI, AMO e AMP. Demais antibióticos sensível. Enterobacter sp Resistente à:CRO, ERI, CIP, IPM, AMO e AMP. Demais antibióticos sensível. Serratia sp Resistente à: CRO, ERI, IPM, AZI, AMO e AMP. Demais antibióticos sensível 2 Serratia sp Resistente à: : CRO, CLO, SBA, AMI, AZI, AMO, AMP e GEN. Demais antibióticos sensível. Enterobacter sp Sensível a todos os antibióticos testados. Enterobacter sp Resistente à: AMO e AMP. Consta intermediária para CIP. Demais antibióticos sensível 3 Klebsiella sp Resistente à:CIP. Intermediário para CRO e GEN. Demais antibióticos sensível. Klebsiella sp Resistente à: GEN. Demais antibióticos sensíveis. Serratia sp Resistente à: CRO, CLO, SBA, CIP, IPM, AZI, AMO, AMP. Consta intermediária para CIP. Demais antibióticos sensível 4 E. coli Resistente à: SBA, AZI, GEN. Intermediário para IPM. Demais antibióticos sensível. Morganella sp Resistente à: CRO, IPM, AMO e AMP. Demais antibióticos sensíveis. Enterobacter sp Resistente à: CRO, CLO, SBA, CIP, AZI, AMO, AMP e GEN. Demais antibióticos sensível. Enterobacter sp Resistente à: AMO e AMP. Demais antibióticos sensíveis. Shigella sp Resistente à: CLO, AMO e AMP. Demais antibióticos sensíveis. 5 Enterobacter sp Resistente à: CRO. Intermediário para CIP. Demais antibióticos sensível. Shigella sp Resistente à: CRO, ERI, AMI, AZI, AMO, AMP e GEN. Demais antibióticos sensíveis. klebsiella planticola Resistente a todos os antibióticos testados. Raoultella planticola Resistente à: CRO, CLO, SBA, CIP, IPM, AZI, AMO, AMP. Intermediário para AMI e GEN 6
Não houve crescimento de EnterobactériasMorganella sp Resistente à: CRO, IPM, AMO, AMP. Demais antibióticos sensíveis. 7 Enterobacter sp Resistente à: AMI e AZI. Demais antibióticos sensível. Enterobacter sp Resistente à: CRO, AMO, AMP. Demais antibióticos sensíveis. E coli Resistente à AZI. Demais antibióticos sensível 8 Enterobacter sp Intermediário para CIP. Demais antibióticos sensível.
Não houve crescimento de bactérias.Serratia sp Resistente à CRO, SBA, AZO, AMO e AMP. Demais antibióticos sensível. 9 Raoultella planticola Resistente à: AZI, AMO e AMPintermediário para SBA, CIP e IPM. Demais antibióticos sensível. Salmonella sp Resistente à: CRO, ERI, AZI, AMO e AMP. Demais antibióticos sensíveis. Enterobacter sp Resistente à SBA, AZI, AMO, AMP, GEN. Intermediário para CIP. Demais antibióticos sensível. E. coli sp Resistente à: AMO e AMP. Demais antibióticos sensíveis. Providencia sp Resistente à: ERI, IPM AZI, AMO e AMP. Demais antibióticos sensíveis. Solo fundo da nascente Klebsiela planticola Resistente a todos os antibióticos testados E. coli Sensível a todos os antibióticos testados Solo jusante na nascente Providencia sp Resistente à: ERI ,IPM ,AZI e CIP. Demais antibióticos sensível. Proteus vulgaris Resistente à CIP Demais antibióticos sensível. Morganella Resistente à CRO, AMO e AMP Solo montante E. coli Sensível a todos os antibióticos testados E. coli Sensível a todos os antibióticos testados Água da nascente Klebsiela planticola Resistente a todos os antibióticos testados. Klebsiela planticola Resistente a todos os antibióticos testados. E. coli Resistente à CIP, AM, ERI e AMP
Alguns autores já relatam em pesquisa de Enterobactérias, principalmente E. coli, por ser um indicador de contaminação fecal, em águas superficiais e subterrâneas. Rodrigues et al. (2009), encontrou Escherichia coli resistente à Tetraciclina no Rio Piracuama – SP. Schneider et al. (2009), encontrou Escherichia coli resistente à Ácido malidixico, amicacina, amoxaciclina/ácido clavulâmico, ampicilina, cefaclor, ciprofloxacina, cloranfenicol, estreptomicina, gentamicina, neomicina, sulfonamida, tetraciclina, tobramicina e trimetropina em Águas superficiais e subterrâneas em área de produção de suínos de diversas localidades. Barreto et al. (2014), encontrou Escherichia coli resistente à Tetraciclina, ácido clavulânico. nalidixico, ciprofloxacino e sulfazotrim no Açude Santo Anastácio – Ceará, Brasil. Caumo et al. (2010), encontrou Escherichia coli resistente à Amoxacilina/ácido clavulâmico, tetraciclina ampicilina, amicacina, piperacilina/tazobactam, sulfametoxazol/trimetropim, norfloxacina cloranfenicol na Lagoa dos Patos, RS. Bortoloti et al. (2018), encontrou Escherichia coli resistente Resistência à penicilina (PEN) e a amoxicilina (AMO) no Município de Itajubá está localizado no sul do Estado de Minas Gerais.
Em outro estudo no mesmo local pesquisado, Erbe et al (2010) relatam que as águas subterrâneas do local estão contaminados com fármacos diversos, ibuprofeno, diclofenaco e antibióticos, e que ocorre no local a presenta de Coliformes totais, E. coli, Enterococos, Clostridios sulfito redutores e Pseudomonas aeruginosa.
Com a realização da contagem, identificação e qualificação da resistência microbiana e a comparação com a lista de criticidade da Organização Mundial da Saúde e tomando base o CONAMA 396, é possível mensurar valores aceitáveis e valores de intervenção em áreas de estudo com perfil de possível contaminação hospitalar conforme tabela 6 a seguir.
Tabela 06: Valores propostos de recomendação e de intervenção para águas subterrâneas.
Agua subterrânea x Microrganismo Consumo humano Dessedentação de animais Irrigação Recreação Intervenção Pseudomonas sp Ausência em 100 mL Ausência em 100 mL Ausência em 100 mL 10 em 100 mL 100 em 100 mL Escherichia coli Ausência em 100 mL 200 em 100 mL 100 em 100 mL 800 em 100 mL 103 por mL Klebsiella pneumoniae Ausência em 100 mL Ausência em 100 mL Ausência em 100 mL 10 em 100 mL 100 em 100 mL Proteus spp Ausência em 100 mL 200 em 100 mL 100 em 100 mL 800 em 100 mL 103 por mL Serratia spp Ausência em 100 mL 200 em 100 mL 100 em 100 mL 800 em 100 mL 103 por mL Salmonella sp Ausência em 100 mL Ausência em 100 mL Ausência em 100 mL 10 em 100 mL 100 em 100 mL Shigella spp Ausência em 100 mL Ausência em 100 mL Ausência em 100 mL 10 em 100 mL 100 em 100 mL Morganella spp Ausência em 100 mL 200 em 100 mL 100 em 100 mL 800 em 100 mL 103 por mL Providencia spp Ausência em 100 mL 200 em 100 mL 100 em 100 mL 800 em 100 mL 103 por mL Enterobacter sp Ausência em 100 mL 200 em 100 mL 100 em 100 mL 800 em 100 mL 103 por mL Staphylococcus aureus (MRSA) resistente à meticilina e vancomicina. Ausência em 100 mL Ausência em 100 mL Ausência em 100 mL 10 em 100 mL 100 em 100 mL Streptococcus pneumoniae sem sensibilidade à penicilina Ausência em 100 mL Ausência em 100 mL Ausência em 100 mL 10 em 100 mL 100 em 100 mL Clostridium spp Ausência em 100 mL Ausência em 100 mL 100 em 100 mL 100 em 100 mL 1000 em 100 mL Coliformes totais Ausência em 100 mL Ausência em 100 mL 1000 em 100 mL — — Coliformes fecais Ausência em 100 mL Ausência em 100 mL 100 em 100 mL 1000 em 100 mL 10000 em 100 mL
Valores propostos recomendados e de intervenção para solos.
Solo x Microrganismo Orientativo APA Área de proteção ambiental Comercial Industrial / Cemitérios Agrícola Pseudomonas sp Ausência Ausência Ausência 103 UFC/g Ausência Klebsiella sp Ausência Ausência Ausência 103 UFC/g Ausência Salmonella sp Ausência Ausência Ausência 103 UFC/g Ausência Shigella sp Ausência Ausência Ausência 103 UFC/g Ausência Coliformes totais 105 UFC/g 105 UFC/g 106 UFC/g 109 UFC/g 108 UFC/g Coliformes fecais 103 UFC/g 103 UFC/g 105 UFC/g 106 UFC/g 106 UFC/g
Dessa forma, a resolução brasileira, CONAMA 420:2009, possui uma lacuna acerca de microrganismos de alto impacto na saúde coletiva e deve ser revista com a introdução de controles microbiológicos de igual forma que as internacionais.
Resultado do sequenciamento genético
O sequenciamento genético foi realizado para as bactérias que apresentaram o maior perfil de resistência aos antibióticos das águas subterrâneas. Como era esperado que o genoma 200310148457 fosse relacionado à Klebsiella pneumoniae e o genoma 200310148458 à Serratia spp, foram executadas análises de ANIb dos respectivos genomas usando como referência os genomas K. pneumoniae IS22 e Serratia sp. AS12.
Deste resultado, verificou-se que o genoma 200310148457 alinhou 3.793.418 pb (78,84%) de seu genoma com K. pneumoniae enquanto que 4.229.810 pb (70,0%) foram alinhados com o genoma de Raoultella planticola.
A expressão de um organismo se faz pela Dominância de seus genes, portanto se trata do gênero Klebsiella. Porém, com 70% de Raoultella planticola não se trata de K. pneumoniae e sim diante de uma nova linhagem de Klebsiella.
Também podemos concluir que todas estão conectadas em um ancestral comum e que devido ao mecanismo de conjugação e trocas genéticas através dos pilus sexuais, organela presente nas Enterobacterias, realizam trocas genéticas e esse é o fato, que despontam como perigo atual e futuro, devido à falta de medicamentos que possam atuar contra a infecção hospitalar.
Por sua vez, a outra bactéria, o genoma do isolado 200310148458 alinhou 2.449.421 pb (45,65%) com o genoma de Serratia sp. AS12, enquanto que 4.217.544 pb (78,60%) foram alinhados com o genoma de R. planticola FDAARGOS_64. Ou seja, o valor de 45,65% de Serratia, condiz no comportamento de crescimento em ágar e teste bioquímico indicando que a bactéria teria uma simetria com Serratia sp, porém essa bactéria se trata de R. planticola. Além da atribuição de espécie por comparação de genoma, também se realizou a identificação de espécie usando os genes 16S ribosomal RNA (16S rRNA) e DNA gyrase B (gyrB). Através de uma análise com a ferramenta blastn on-line (parâmetros default), usando como banco de dados o nt/nr, verificou-se que para o isolado com características de Serratia, apresentou a identidade de 99,67% com a bactéria Raoultella planticola (CP026047) e a Klebsiella planticola, por não estar na literatura internacional, não é possível uma comparação através desse artifício, pois não há consenso mundial sobre a nomenclatura da Klebsiella planticola para Raoutella planticola. Já existem estudos que evidenciam as diferenças entre os gêneros Klebsiella e Raoutella por meio dos genes ARNr 16S (Arenas et al., 2009; Kanki et al., 2002).
Portanto, de posse do resultado do sequenciamento genético, buscamos outras características e foram realizados testes de crescimento de bactérias a 10°C, uma vez que de acordo com a proposta de Drancourt et al. (2001) para classificação de Klebsiella plantícola e Raoultella planticola, entre as características que diferenciariam as espécies, a Raoultella apresentaria crescimento em meio de cultura à 10°C e a Klebsiella não apresentaria crescimento a temperatura de 10°C.
Ao colocar as placas semeadas com as bactérias e incubadas em estufa mesófila na temperatura de 35 e 10° C, observou-se que a bactéria que pareou seu genoma com K. pneumoniae, ocorreu crescimento apenas na placa incubada à 35º C e não se desenvolveu em 10º C, diferente da bactéria que alinhou com o genoma de Raoultella planticola se desenvolveu em ambas as temperaturas. O estudo de Drancourt et al. (2001), foi baseado diretamente na temperatura e agora com a contribuição genética, podemos verificar que ocorrem no Ecossistema ambas as bactérias.
Em incubação em ágar EMB (figura 1), por 48 horas, a klebsiella planticola apresenta o mesmo padrão de crescimento que a klebsiella pneumoniae, colaração ágar EMB rosa claro (figura B) e a Raoultella planticola rosa escuro apresenta o mesmo padrão de crescimento que Serratia sp (figura A).
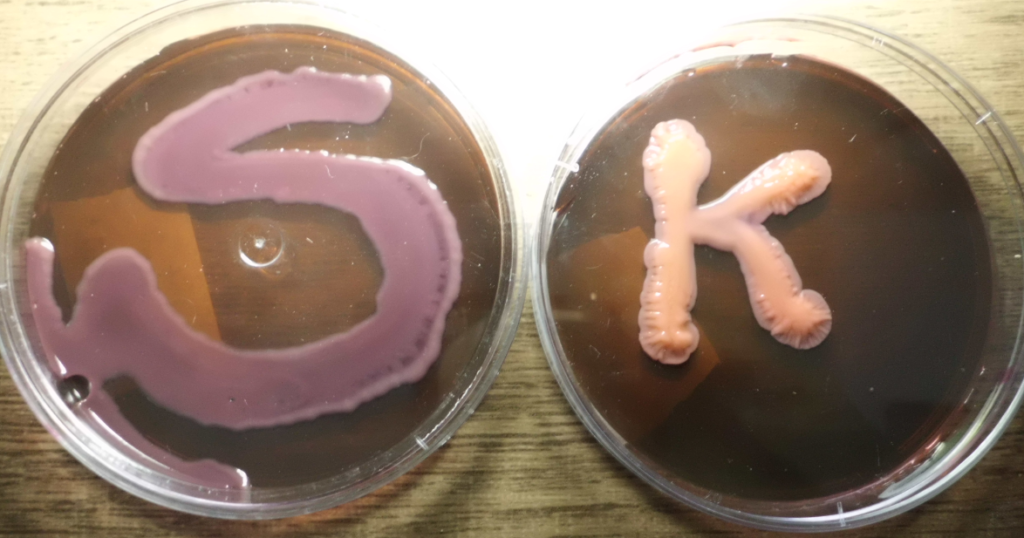
Figura 01: A:Raoultella planticola (48 horas) B: S(Raoultella) K (Klebsiella)
De acordo com Morais et al. (2009) infecções humanas causadas por bactérias do gênero Raoultella são raras. Observa-se, porém, o crescimento de relatos de casos de bacteremia relacionados com Enterobactérias desta linhagem. Castanheira et al. (2009), que relataram os 3 primeiros casos de infecção fatal de pacientes por blaKPC Raoutella spp. Nos EUA, mencionam que este tipo de bactéria é comumente encontrado em diversos compartimentos ambientais, entre estes águas estuarinas e peixes, e que a semelhança com Klebsiella spp pode ter facilitado a troca de material genético entre estas espécies de Enterobactérias.
Além dessas evidências, as bactérias que foram encontradas no mesmo poço PM5, apresentaram resistências à antibióticos de formas diferentes sendo a Klebsiella planticola resistente a todos os antibióticos testados, CRO 30(ceftriaxone), CLO 30 (Chloramphenicol), SBA 10 e 10 (Ampicillin-sulbactam), CIP 5(Ciprofloxacin), AMI 30 (Amikacin), IPM 10 (Imipenem), AZI 30 (Azithromycin), AMO 10 (Amoxicilina), AMP 10 (Ampicillin) e GEN 10 (Gentamicin), enquanto a Raoutella planticcola foi resistenteà CRO 30(ceftriaxone), CLO 30 (Chloramphenicol), SBA 10 e 10 (Ampicillin-sulbactam), CIP 5(Ciprofloxacin), IPM 10 (Imipenem), AZI 30 (Azithromycin), AMO 10 (Amoxicilina), AMP 10 (Ampicillin) e intermediário para AMI 30 (Amikacin) e GEN 10 (Gentamicin). Isso demonstra que ambas possuem, na parte genotípica de resistência, expressões diferentes, ou seja, contendo uma adaptação diferenciada.
Segundo ANVISA (2013; 2004), o tratamento medicamentoso contra gênero de Klebsiella consiste o uso não apenas de 1 mais a combinação de antibióticos, de forma a evitar desenvolvimento de uma nova mutação. O recomendado é administração de dois ou mais antibióticos, principalmente a gentamicina ou amicacina (classe de aminoglicosídeos) e meropenen ou doripenem (classe de carbapenêmicos) com o acréscimo de polimixina B ou E, visto que ainda é possível observar certa efetividade devido ao tamanho do fenômeno de resistência dessa bactéria, sendo conhecida popularmente como superbactéria.
Dessa forma, não é possível admitir que Klebsiella planticola não faça parte das linhagens de Klebsiella, visto que ambas as bactérias coexistem, e portanto, a Klebsiella planticola deve ser restituída dentre as linhagens de Klebsiella e a Raoultella planticola deve ser mantida na linhagem de Raoultella.
CONCLUSÕES
Os estudos retratam sobre a disseminação de bactérias resistentes à amoxicilina, eritromicina e ampicilina e de forma preocupante em alguns compartimentos ambientais e no sedimento de fundo de uma nascente próxima a um aterro, o que já ocorre em diversos países.
Foram evidenciadas em um dos poços subterrâneos duas bactérias similares à bactéria Klebsiella planticola que se mostrouresistente a todos os antibioticos testados e a Raoultella planticola contendo certa resistência o que demonstra uma lacuna nas legislações relacionados a águas, solos e sedimentos quando se trata de parametros biológicos.A norma de passivo ambiental no Brasil, CONAMA 420:2009, e de outros países, contemplam apenas parâmetros físicos e químicos e, portanto, precisa ser complementada com parâmetros biológicos, a fim de atender as tendências atuais, com vistas ao impacto ambiental de resíduos hospitalares e a introdução de microrganismos de interesse clínico.
Dentro da pesquisa é oportuno questionar e frisar sobre a nomeação da Enterobactéria realizada no ano de 2001, pois ambas foram encontradas no local do estudo, Klebsiella plantícola é uma subespécie de Klebsiella e não deveria ter sido renomeada para Raoutella plantícola, visto que cada uma delas apresenta características próprias e se tratam de bactérias independentes e para tanto, seria prudente, uma revisão dessa nomenclatura. Por fim, indicamos alguns parâmetros microbiológicos e valores orientadores para iniciar uma discussão sobre a tendência legislativa para os próximos anos.
AGRADECIMENTOS
Agradecemos a Empresa Latam Laboratórios pelo financiamento da publicação deste artigo, contribuindo para o avanço da Ciência no país. Agradecemos a Goldlab Ciência e Tecnologia por incentivar a pesquisa e tecnologia e criar meios para ensaios mais simples e robustos, em prol da introdução de ensaios técnicos em diversos ambientes que se fazem necessários. Agradecemos a Prefeitura Municipal de Curitiba e seus técnicos, em especial a Engenheira civil Lucy Schellin e ao Programa de Gestão Ambiental da Universidade Positivo, por sempre disponibilizar acesso aos pontos amostrados e apresentar dados de outros estudos e monitoramentos.
REFERÊNCIAS
ALMEIDA LP, CARVALHO FP, MARQUES AG, PEREIRA A S, BORTOLETO RP, MARTINO MD. Desempenho do disco de ertapenem como preditor da produção de Klebsiella pneumoniae carbapenemase por bacilos Gram-negativos isolados de culturas em um hospital municipal de São Paulo. Einstein; 2012
ANVISA. AGÊNCIA NACIONAL DE VIGILÂNCIA SANITÁRIA. Segurança e controle de qualidade no laboratório de microbiologia clínica: módulo II. Brasília, DF, 2004b. Disponível em: <http://www.anvisa.gov.br/servicosaude/microbiologia/mod_2_2004.pdf
ANVISA. Infecção Hospitalar – Controle. 2. Infecção em Serviços de Saúde. 3. Microbiologia Clínica. 4. Vigilância Sanitária em Serviços de Saúde. 5. Resistência microbiana. I. Brasil. ANVISA Ministério da Saúde. 2004.
ANVISA. Agência Nacional de Vigilância Sanitária (ANVISA). Nota Técnica n° 1/2013 – Medidas de prevenção e controle de infecções por enterobactérias multiresistentes. Brasília (DF): ANVISA; 2013.
APHA, AWWA, WEF in: L.S. Clesceri, A. E.; Greenberg, A. D. Eaton (Eds.), Standard Methods for the Examination of Water and Wastewater, 23th ed., American Public Health Association, American Water Works Association, Water Environment Federation,Washington,DC, USA, 2017.
ARENAS, N. H.; GUTIÉRREZ, A. J.; SALAZAR, L. M.; POLANCO, J. C.; GÓMEZ, A. Construcción de una filogenia molecular par alas especies de los géneros Klebsiella y Raoutella basada en los genes ARNr 16S y ARN polimerasa subunidad. Ciencias de la salud. v. 7, n. 2, 2009.
BANKEVICH, A., et al. SPAdes: A New Genome Assembly Algorithm and Its Applications to Single-Cell Sequencing. Journal of Computational Biology, v. 9, n. 5, p. 455-477, 2012. Seemann, T. Prokka: rapid prokaryotic genome annotation.Bioinformatics, v. 30, n. 14, p. 2068-2069, 2014.
BARRETO. Norma Suely Evangelista-Barreto; Priscila Coutinho Miranda; Danillo Cerqueira Barbosa; Ricardo Henrique Bastos de Souza4 ; Marly Silveira Santos. Condições higiênicas sanitárias da carne de sol comercializada no município de Cruz das Almas, Bahia e detecção de cepas com resistência antimicrobiana. Ciências Agrárias, Londrina, v. 35, n. 3, p. 1311-1322, maio/jun. 2014.
BORTOLOTI et al. Karina da Costa Sassi Bortoloti , Rogerio Melloni , Paulo Sérgio Marques , Brenda Mayra Fernandes de Carvalho, Mariléia Chaves Andrade. Qualidade microbiológica de águas naturais quanto ao perfil de resistência de bactérias heterotróficas a antimicrobianos. Eng Sanit Ambient | v.23 n.4 | jul/ago. 2018.
BRASIL. Resolução CONAMA nº 396. Conselho Nacional do Meio Ambiente. Brasília, 03 de agosto de 2008.
BRASIL. Resolução CONAMA nº 420. Conselho Nacional do Meio Ambiente. Brasília, 28 de dezembro de 2009.
BROOKS. Microbiologia médica de Jawetz, Melnick e Adelberg [recurso eletrônico] / Geo. F. Brooks … [et al.] ;– 26. ed. – Dados eletrônicos. Porto Alegre: AMGH, 2014.ISBN 978-85-8055-335-2. 2014
CANADÁ, CCME. Canadian Council of Ministers of Environment, Canadian EnvironmentaI Quality Guidelines. 1995. .[online]. Acesso: 14 de abril de 2021
CANADÁ, CCMTR. Canadian Council of Ministers of the Environment. Canadian EnvironmentaI Quality Guidelines. PN1299. Winnipeg, MN. 1999. .[online]. Acesso: 14 de abril de 2021
CASTANHEIRA, M.; DESHPANDE, L. M.; DIPERSIO, J. R.; KANG, J.; WEINSTEIN, M.; JONES, R. N. First description of blaKPC in Raoutella spp. (R. planticolla and R. ornithinolytica): report from the SENTRY Antimicrobial surveillance program. Journal of Clinical Microbiology. v. 47, N. 12, P. 4129-4139, 2009. Doi: 10.1128/jcm.01502-09.
CAUMO et al., K.; DUARTE, M.; CARGNIN, S.T.; RIBEIRO, V.B.; TASCA, T.; MACEDO, A.J. Resistência bacteriana no meio ambiente e implicações na clínica hospitalar. Revista Liberato, v. 11, n. 16, p. 183-190. 2010.
CLEAN WATER. Clean Water Act [33 U.S.C. § 1251-1387. Sec. 505] – LEI FEDERAL DE CONTROLE DA POLUIÇÃO DA ÁGUA, COMO ALTERADO PELA ÁGUA LIMPA. 1977.
CLSI publication M100-S21 Suggested Grouping of US-FDA Approved Antimicrobial Agents That Should Be Considered for Routine Testing and Reporting on Nonfastidious Organisms by Clinical Laboratories, 2011.
CURITIBA. Plano Municipal de Saneamento Básico, volume IV, Gestão Integrada de resíduos sólidos 2017. Disponível em:<https://mid.curitiba.pr.gov.br/2017/00211737.pdf>. Acesso em 25 abr. 2020.
COIL, D.; Jospin, G.; Darling, A. E. A5-miseq: an updated pipeline to assemble microbial genomes from Illumina MiSeq data. Bioinformatics, v. 31, n. 4, p. 587–589, 2015.
DRANCOURT, M.; BOLLET, C.; CARTA, A.; ROUSSELIER, P. Phylogenetic analysis of Klebsiella species delineate Klebsiella gen. nov., with description of Raoultella ornithinolytica comb. nov., Raoultella terrigena comb. nov. and Raoultella planticola comb. nov. International Journal of Systematic and evolutionary microbiology. n. 51, p.925-932. 2001.
Erbe, M.C.L, Stevanato R., Ferreira, J.F. F. Aplicação da eletrorresistividade na avaliação de passivo ambiental: vala séptica de Curitiba-PR. Copyright 2010, SBGf – Sociedade Brasileira de Geofísica. 2010
EPA. 600- R-94-173. Notas Técnicas sobre Métodos de Água Potável outubro de 1994. National Service Center for Environmental Publications (NSCEP), PO Box 42419, Cincinnati, OH 45242-0419. 1994. http://www.epa.gov/ nscep / acesso em abril 2021.
EPA. 48 FR 51405, 8 de novembro de 1983, 51047, 21 de agosto de 2015. Centro de Recursos Hídricos, USEPA, Código Postal RC4100, M Street SW , Washington, DC 20460 e a nota de §131.36 (b) (1). 2015.
EPA 40. Title 40. Protection of Environment. CAPÍTULO I – AGÊNCIA DE PROTEÇÃO AMBIENTAL (CONTINUAÇÃO), SUBCAPÍTULO N – DIRETRIZES E PADRÕES DE EFLUENTE, SUBCAPÍTULO D – PROGRAMAS DE ÁGUA /PARTE 131 – PADRÕES DE QUALIDADE DA ÁGUA;Autoridade: Secs. 307, 308, 501, Emendas da Lei Federal de Controle de Poluição de Água de 1972 (Pub. L. 92-500, 86 Stat. 816, (33 USC 1251). U.S. Superintendent of Documents . Washington, DC 20402–0001 Parts 1 to 49 Revised as of July 1, 2021. http://bookstore.gpo.gov, ACESSO em abril 2021.
GISKE, C. G.; GEZELIUS, L.; SAMUELSEN, W. M.; SUNDSFJORD, A.; WOODFORD, N. A sensitive and specific phenotypic assay for detection of metallo-βlactamases and KPC in Klebsiella pneumoniae with the use of meropenem disks supplemented with aminophenylboronic acid, dipicolinic acid and cloxacillin. Clin Microbiol Infect. 2011.
GOODWIN, M. A.; WALTMANN, W. D. Transmission of Eimeria, Viruses, and bacteria to the chicks: darkling beetles (Alphitobius diaperinus) as vectors of pathogens. Journal of Applied Poultry Research v. 5, p. 51-55, 1996.
KANKI, M.; YODA, T.;TSUKAMOTO, T.; SHIBATA, T. Klebsiella pneumonia produces no histamine: Raoutella planticola and raoutella ornithinolytica strains are histamine producers. Applied and environmental microbiology. V.68, n. 7, p. 3462-3466, 2002.
KOSUGI, S.; Hirakawa, H.; Tabata, S.: GMcloser: closing gaps in assemblies accurately with a likelihood-based selection of contig or long-read alignments. Bioinformatics, v. 31, n. 23, p. 3733–3741, 2015.
LIMBARGO BM, Rasheed JK, Anderson KF, Zhu W, Kitchel B, Watz N, et al. IMPproducing carbapenem-resistant Klebsiella pneumoniae in the United States. J Clin Microbiol. 2011
LISTA HOLANDESA – Lista 6530 de valores de águas e solos – Legislação Internacional para solos e águas subterrâneas – Padrão Internacional. Versão novembro de 1999. Anexos 1,2,3 e 4.[online]. Acesso: 14 de abril de 2021
MORAIS, V. P.; DAPORTA, M. T.; BAO, A. F.; CAMPELLO, M. G.; ANDRÉS, G. Q. Enteric fever-like syndrome caused by raoutella ornithinolytica (Klebsiella ornithinolytica) Journal of Clinical Microbiology. 2009. DOI: 10.1128/JCM.01709-08.
NCCLS. Performance Standards for Antimicrobial Disk Susceptibility Tests; Approved Standard—Eighth Edition. NCCLS document M2-A8 (ISBN 1-56238-485-6). NCCLS, 940 West Valley Road, Suite 1400, Wayne, Pennsylvania 19087-1898 USA, 2003
OLPLUSTIL, C. P. et al. Procedimentos Básicos em Microbiologia Clínica. 3.ed. Sarvier: São Paulo, 2010.
OMS. OMS pública lista inédita de bactérias resistentes a antibióticos. Direitos humanos,
2017. Disponível em: <https://nacoesunidas.org/oms-publica-lista-inedita-de-bacterias-resistentes- a-antibioticos/amp/>. Acesso em15 de fevereiro de 2020.
PATEL, J. B.; RASHEED, J. K.; BRANDONKITCHEL, M. Carbapenemases in Enterobacteriaceae: activity, epidemiology and laboratory detection. Clin Microb News. 2009.
PENG, Y.; Leung, H.C.; Yiu, S.M.; Chin, F.Y. IDBA-UD: a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth. Bioinformatics, v. 28, n. 11, p. 1420-1428, 2012.
RCRA. Resource Conservation and Recovery Act of 1976 (RCRA), Hazardous and Solid Waste Amendaments em 1984; (HSWA) – Do US Government Publishing Office, www.gpo.gov acesso em abril de 2021.
RICHTER, M.; Rosselló-Móra, R.; Glöckner, F.O.; Peplies, J.: JSpeciesWS: a web server for prokaryotic species circumscription based on pairwise genome comparison., Bioinformatics, 2015.
RODRIGUES, Jane Rose Dias Dionísio 1 JORGE, Antonio Olavo Cardoso 2 UENO, Mariko. Avaliação da qualidade das águas de duas áreas utilizadas para recreação do Rio Piracuama- recreação do Rio Piracuama-SP. REVISTA BIOCIÊNCIAS, UNITAU. Volume 15, número 2, 2009.
SCHNEIDER et al. Perfil de resistência antimicrobiana de isolados de Escherichia coli obtidos de águas superficiais e subterrâneas, em área de produção de suínos Roger Neto Schneider André Nadvorny Verônica Schmid. Biotemas, 22 (3): 11-17, setembro de 2009.
SPANU T. Fiori B, D’Inzeo T, Canu G, Campoli S, Giani T, et al. Evaluation of the New NucliSENS EasyQ KPC test for rapid detection of Klebsiella pneumoniae carbapenemase genes (blaKPC). J Clin Microbiol. 2012
TORTORA, Gerard J., FUNKE, Berdell R., CASE, Christine L. Microbiologia, 12th Edition. ArtMed. 2017.
US GEOLOGICAL. Método I-3720-85, Techniques of Water Resources Investigation of the US Geological Survey, Livro 5, Capítulo A-1, 3ª ed., 1989. Disponível em Information Services, US Geological Survey, Federal Center, Box 25286, Denver, CO 80225-0425. 1989.
XIUJUAN, M, Sidi L, Juping D, Xun H, Pengcheng Z, Xinrui X, et al. Risk factors and medical costs for healthcare-associated carbapenem-resistant Escherichia coli infection among hospitalized patients in a Chinese teaching hospital. BMC Infect Dis. 2017.
1 Mestre em Ciências, Biomédica, Química e Biotecnologia. Pesquisadora e Proponente do PPGEO/UFPR. haluchsil@hotmail.com
2 Estudante de Biomedicina da Universidade Pequeno Príncipe – FPP/PR Bolsista dos Programas desenvolvidos pela Goldlab Ciência e Tecnologia Ltda em parceira com Latam Laboratórios de Análises Toxicológicas e Ambientais Ltda. goldlablaboratorios@gmail.com
3 Biomédico Especialista em Microbiologia com ênfase em Resistência microbiana, Toxicologia e Química Analítica. Diretor da Latam Laboratórios de Análises Toxicológicas e Ambientais Ltda. johnatan@lablatam.com.br
