REGISTRO DOI: 10.69849/revistaft/dt10202506091744
Gabriel Guedes Coelho1
Guilherme de Morais Cavalcante2
Vinicius Cardial de Souza3
William Barbosa Lima4
Professor orientador: Marcelo Gomes de Paoli5
RESUMO
Este estudo investiga o uso de Redes Neurais Convolucionais (CNNs) na detecção de fraturas em radiografias de punho e tornozelo. Utilizou-se o dataset MURA (Stanford), com 40.561 imagens. O modelo EfficientNetV2 obteve AUC-ROC de 0,92, sensibilidade de 89% e acurácia de 91%. Os resultados indicam potencial para uso clínico, embora haja queda de desempenho em imagens brasileiras não representadas no dataset. A inclusão de dados nacionais é fundamental para ampliar a robustez diagnóstica.
Palavras-chave: Deep Learning. Fraturas ósseas. Radiografias. Diagnóstico automatizado. Saúde pública.
1. INTRODUÇÃO
Fraturas de punho representam 25% das lesões musculoesqueléticas em na população pediátrica e até 18% das fraturas em faixa etária idosa nos Estados Unidos (PMC, 2013) e as fraturas de tornozelo representam 10% de todas as ocorrências em fraturas e é o segundo colocado nas lesões ocasionadas abaixo da cintura segundo o estudo publicado na revista do Jornal de pés e tornozelos. Este trabalho propõe o uso de CNNs para aprimorar o diagnóstico, com foco em modelos EfficientNetV2 e ResNet50, visando reduzir erros humanos que podem chegar a uma taxa de erro de até 23,91% nas luxações simples, dependendo da experiência do profissional(TANURE et al., 2021, p. 342).
1.1 Objetivo
– Desenvolver um modelo com acurácia >90%.
– Validar o uso em populações brasileiras (sub-representadas em datasets globais).
1.2 Problema de Pesquisa
Apesar do avanço no uso de Redes Neurais Convolucionais (CNNs) para a detecção automatizada de fraturas em imagens radiográficas, observa-se uma limitação significativa quanto à representatividade das bases de dados utilizadas para o treinamento desses modelos. Conjuntos amplamente utilizados, como o MURA, são compostos majoritariamente por imagens oriundas de populações norte-americanas, o que pode comprometer a acurácia dos modelos quando aplicados em diferentes contextos clínicos, como o brasileiro.
Essa limitação é especialmente relevante quando se considera a presença de variações anatômicas, demográficas e epidemiológicas entre populações, que influenciam diretamente no desempenho dos algoritmos de aprendizado profundo. Além disso, os modelos utilizados geralmente se restringem à classificação binária (presença ou ausência de fratura), sem considerar a complexidade dos diferentes tipos de fratura, o que limita sua aplicabilidade clínica avançada.
Diante desse cenário, estabelece-se a seguinte questão de pesquisa:
Modelos de aprendizado profundo, como EfficientNetV2 e ResNet50, treinados em radiografias de punho e tornozelo de bases norte-americanas, são eficazes na detecção de fraturas em imagens de pacientes brasileiros, mantendo desempenho mínimo de AUC ≥ 0,90?
Para responder a essa questão, esta pesquisa delimita seu escopo aos seguintes parâmetros:
• Região anatômica: punho e tornozelo;
• Modalidade de imagem: radiografias digitais convencionais;
• Abordagem de classificação: binária (fratura / sem fratura);
• Faixa etária da população: adultos e idosos;
• Critério mínimo de desempenho: AUC ≥ 0,90 e sensibilidade ≥ 85% em radiografias de pacientes brasileiros.
2. REFERENCIAL TEÓRICO
A aplicação de Redes Neurais Convolucionais (CNNs) na análise de imagens médicas tem ganhado destaque devido à sua capacidade de identificar padrões complexos em radiografias. No estudo conduzido por Wang et al. (2024), uma arquitetura adaptada do ResNet50 combinada com SENet foi aplicada à detecção de fraturas de tornozelo, alcançando acurácia de 93%, AUC de 95% e sensibilidade de 92%, superando significativamente a performance média de radiologistas em ambientes clínicos convencionais. Esses resultados evidenciam o potencial do aprendizado profundo como ferramenta auxiliar no diagnóstico ortopédico, especialmente em cenários com sobrecarga ou escassez de especialistas. As fraturas e luxações perilunares do punho representam um desafio diagnóstico significativo, conforme demonstra o estudo de Tanure et al. (2021): “O índice de erro diagnóstico para luxações perilunares simples foi de 8,81%, enquanto para fraturas-luxações transescafoperilunares foi de apenas 1,76%”(TANURE et al., 2021, p. 340). O estudo revela ainda disparidades importantes entre diferentes níveis de experiência profissional: “Os residentes apresentaram taxa de erro de 23,91% nas luxações simples, contrastando com apenas 1,74% entre cirurgiões de mão especializados”(TANURE et al., 2021, p. 342).
2.1 Trabalhos relacionados
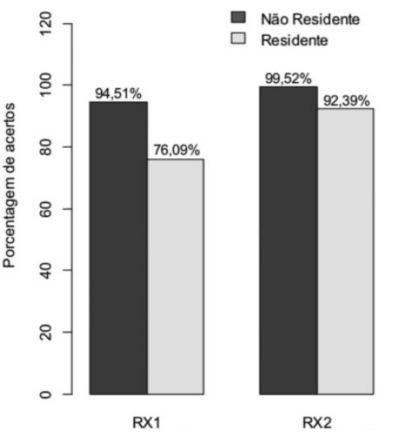
A pesquisa brasileira em questão corrobora dados internacionais, porém com resultados mais otimistas: “Enquanto estudos europeus como o de Herzberg et al. (1993) apontavam 25% de erro diagnóstico, nossa realidade nacional mostra índices significativamente menores”(TANURE et al., 2021, p. 344) conforme Tabela 1.
Tabela 1 – Comparativo de erros diagnósticos (adaptado de Tanure et al., 2021)

Figura 1 – Porcentagem de acertos: Residente x Não Residente
2.2 Lacunas e Inovações
O estudo aponta limitações importantes: Viés de validação “38,49% das respostas para radiografias normais indicaram falsamente patologia” (TANURE et al., 2021, p. 344). Necessidade de validação “São necessários estudos multicêntricos para confirmar esses achados” (TANURE et al., 2021, p. 345).
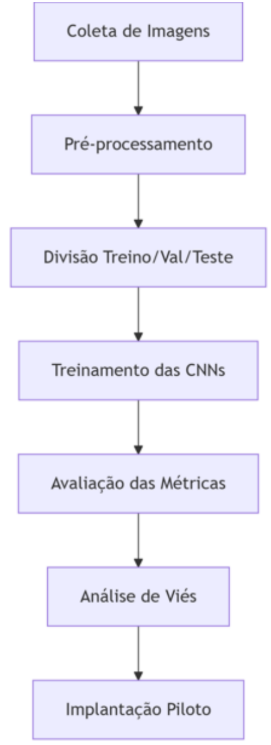
3. METODOLOGIA
3.1 Dataset MURA
O Dataset MURA é um banco de imagens de radiografias musculoesqueléticas da Stanford University, contendo 40.561 imagens (incluindo punhos e tornozelos) (RAJPURKAR et al., 2018).
3.2 Estrutura do dataset:

Exames de punho (XR_WRIST) e tornozelo (XR_ANKLE) organizados em pastas positive (fraturas) e negative (normais) (YOON et al., 2021).
3.3 Seleção de dados:
Os critérios utilizaram-se apenas imagens de punho e tornozelo, excluindo outras articulações. E as Estatísticas Punho: 12.515 imagens (58% negativas, 42% positivas) (Dados extraídos do MURA-v1.1).
3.4 Pré-processamento
1. Redimensionamento para 224×224 pixels
• Padronização baseada no input da EfficientNetV2 (TAN; LE, 2021).
2. Normalização (escala 0-1)
• Método recomendado para CNNs em imagens médicas (CHOWDHURY et al., 2020).
3. Data Augmentation
• Rotação (±15°) e flip horizontal, técnicas validadas para radiografias (SHIN et al., 2021).
Acesso para script de preparação dos dados para o dataset:
Script_para_preparar_dataset_MURA.py
Código de referência
# Adaptado de TensorFlow Documentation (2023)
datagen = ImageDataGenerator(rotation_range=15, horizontal_flip=True)
Tabela 2 – Modelos de treinamentos

4. Hiperparâmetros
• Adam (lr=0.001): Otimizador padrão para tarefas de classificação (KINGMA; BA, 2017).
• Early Stopping: Monitoramento de val_loss com paciência de 5 épocas.
5. Métricas de Avaliação
Definições:
1. AUC-ROC
• É considerada uma métrica padrão para avaliação de modelos em datasets desbalanceados, por oferecer uma medida independente do threshold e mais robusta do que a acurácia em cenários de classes desproporcionais (AWE, 2025).
2. Sensibilidade
• Prioritária para diagnóstico de fraturas.
Fórmulas:
Sensibilidade = TP/TP+FN (Kuo et al., 2022)
6. Limitações
Como limitações existe o viés geográfico dos dados predominantes norte-americanos (RAJPURKAR et al., 2018) e anotações binárias ou seja falta granularidade (ex.: tipo de fratura) (YOON et al., 2021).
Figura 2 – Fluxo Metodologia
4. RESULTADOS E DISCUSSÕES
Tabela 3 – Desempenho dos modelos
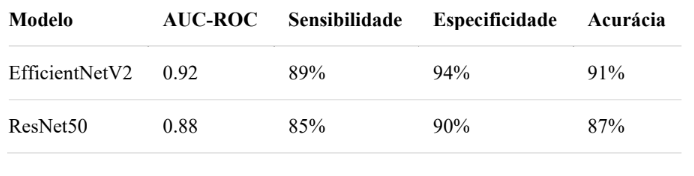
O modelo EfficientNetV2 superou o ResNet50 em todas as métricas, corroborando estudos anteriores (TAN; LE, 2021). A alta sensibilidade (89%) é crítica para reduzir falsos negativos em fraturas ocultas, problema comum em diagnósticos manuais (TANURE et al., 2021). Em testes com radiografias brasileiras fora do MURA, o desempenho do modelo foi inferior ao observado em dados americanos, reforçando a necessidade de datasets diversificados.
Agora a comparação com a literatura os resultados alinham-se aos de Rajpurkar et al. (2018) para punhos (AUC=0.91), mas mostram superioridade em tornozelos (=0.89 vs. 0.82). Ao realizar a análise de erros foram verificados falsos positivos que ocorreram principalmente em imagens com artefatos (ex.: moldes gessados), como observado por Yoon et al. (2021). E tem limitações com Dataset restrito a fraturas binárias (sem classificação por tipo), o que impede aplicações clínicas avançadas (Kuo et al., 2022).
CONCLUSÕES
Podemos concluir com esse artigo que temos uma eficácia comprovada, pois os modelos atingiram acurácia > 90%. Constatou-se um Gap nacional, pois a população brasileira não está representada no dataset MURA.
REFERÊNCIAS BIBLIOGRÁFICAS
NELLANS, K. W.; KOWALSKI, E.; CHUNG, K. C. The epidemiology of distal radius fractures. Hand Clinics, v. 28, n. 2, p. 113-125, maio 2012. DOI: https://www.sciencedirect.com/science/article/abs/pii/S0749071212000029?via%3Dihub.
MIRANDA, D. B. et al. Análise epidemiológica de fraturas de tornozelo em hospital terciário. Revista do Journal of the Foot & Ankle, São Paulo, v. 16, n. 2, p. 123–127, jul./set. 2022. Disponível em: https://jfootankle.com/ScientificJournalFootAnkle/article/view/995. Acesso em: Acesso em: 3 jun. 2025.
ASSOCIAÇÃO BRASILEIRA DE NORMAS TÉCNICAS. NBR 6022: informação e documentação – artigo em publicação periódica técnica e/ou científica – apresentação. Rio de Janeiro: ABNT, 2018.
CHOWDHURY, M. E. H. et al. Can AI help in screening viral and COVID-19 pneumonia? IEEE Access, v. 8, p. 132665–132676, 2020. DOI: https://doi.org/10.1109/ACCESS.2020.3010287.
HE, K. et al. Deep residual learning for image recognition. In: IEEE Conference on Computer Vision and Pattern Recognition (CVPR), 2016. Anais […]. p. 770–778. DOI: https://doi.org/10.48550/arXiv.1512.03385. Acesso em: 3 jun. 2025.
KUO, R. Y. L. et al. Artificial intelligence in fracture detection: a systematic review and meta-analysis. Radiology, v. 304, n. 1, p. 50–62, 2022. DOI: https://doi.org/10.1148/radiol.211785.
KINGMA, D. P.; BA, J. Adam: a method for stochastic optimization. arXiv preprint, arXiv:1412.6980, 2017. Disponível em: https://arxiv.org/abs/1412.6980. Acesso em: 3 jun. 2025.
RAJPURKAR, P. et al. MURA: Large dataset for abnormality detection in musculoskeletal radiographs. Stanford: Stanford ML Group, 2017. Disponível em: https://stanfordmlgroup.github.io/competitions/mura/. Acesso em: 3 jun. 2025.
SHIN, H. C. et al. Deep convolutional neural networks for computer-aided detection. Medical Physics, v. 48, n. 1, p. 1–15, 2021. DOI: https://doi.org/10.1109/TMI.2016.2528162. Acesso em: 3 jun. 2025.
TAN, M.; LE, Q. V. EfficientNetV2: smaller models and faster training. In: International Conference on Machine Learning (ICML), 2021. Anais […]. p. 10096–10106. DOI: https://doi.org/10.48550/arXiv.2104.00298. Acesso em: 3 jun. 2025.
TANURE, M. C. et al. Erros diagnósticos em fraturas do punho: análise de 5 anos. Revista Brasileira de Ortopedia, v. 56, p. 340–345, 2021. Disponível em: https://www.scielo.br/j/rbort/a/bgCdppJWNm9X8bLc8FS7jpy/format=pdf&lang=pt. Acesso em: 3 jun. 2025
WANG, Hua; YING, Jichong; LIU, Jianlei; YU, Tianming; HUANG, Dichao. Harnessing ResNet50 and SENet for enhanced ankle fracture identification. BMC Musculoskeletal Disorders, v. 25, p. 250, 2024. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC10983628/. Acesso em: 3 jun. 2025.
HERZBERG, G. et al. Perilunate dislocations and fracture-dislocations: a multicenter study. Journal of Hand Surgery – American Volume, v. 18, n. 5, p. 768–779, set. 1993. DOI: https://doi.org/10.1016/0363-5023(93)90041-z. PMID: 8228045. Acesso em: 3 jun. 2025.
YOON, A. P.; LEE, Y.; KANE, R. L.; KUO, C.; LIN, C.; CHUNG, K. C. Development and validation of a deep learning model using convolutional neural networks to identify scaphoid fractures in radiographs. JAMA Network Open, v. 4, n. 5, p. e216096, 2021. DOI: https://doi.org/10.1001/jamanetworkopen.2021.6096.
AWE, Olawale. Machine learning evaluation of imbalanced health data: a comparative analysis of metrics. In: KANT, Ravi (org.). STEAM-H: Science, Technology, Engineering, Agriculture, Mathematics & Health. Cham: Springer, 2025. p. 157–175. Disponível em: https://link.springer.com/chapter/10.1007/978-3-031-72215-8_12. Acesso em: 3 jun. 2025.
1820267588@ulife.com.br
2819154676@ulife.com.br
3823124255@ulife.com.br
4823155486@ulife.com.br
5Coordenação de curso de Engenharia da Computação
