REGISTRO DOI: 10.69849/revistaft/ni10202411250719
Gabriel Da Silva De Souza; Geovana Monike Pacheco De Sena; Jayanne De Lima Sousa; Orientadora: Amanda Bezerra Carvalho; Coorientador: Fábio Maués Carmona
RESUMO
Introdução: Os antibióticos beta-lactâmicos são medicamentos comumente administrados no tratamento de infecções bacterianas. Existem, pois, um inibidor da síntese da parede celular de bactérias que é capaz da sobrevivência das mesmas. A resistência bacteriana, especialmente à classe dos antibióticos β-lactâmicos, é um problema crescente na saúde pública, impactando a eficácia dos tratamentos e aumentando os custos hospitalares. Objetivo: Este trabalho tem como objetivo realizar uma revisão sistemática sobre resistência a beta-lactâmicos, abordando seus mecanismos e implicações clínicas. Metodologia: Foi realizada uma revisão sistemática seguindo os critérios PRISMA, com a seleção de 15 artigos completos publicados entre 2014 e 2024, obtidos das bases de dados BVS e MedLine. Os artigos foram analisados criticamente, e dados relevantes foram organizados em tabelas para discutir sobre a resistência a antibióticos beta-lactâmicos e abordar suas causas, mecanismos e implicações clínicas. Resultados: A utilização inadequada de antibióticos contribui para a seleção de cepas resistentes. Os estudos indicam a necessidade de intervenções inovadoras, voltadas para estirpes específicas e métodos alternativos de ação, para gerir e diminuir a resistência bacteriana de forma eficiente. Conclusão: A resistência bacteriana requer uma abordagem multifacetada, incluindo a compreensão dos mecanismos envolvidos e a implementação de novas estratégias de tratamento. A pesquisa destaca a importância de intervenções eficazes para combater infecções resistentes e melhorar os resultados clínicos.
Palavras chaves: bactérias resistentes, antibióticos, cepas resistentes, métodos alternativos.
ABSTRACT
Introduction: Beta-lactam antibiotics are commonly administered drugs for the treatment of bacterial infections. They act as inhibitors of bacterial cell wall synthesis, which is essential for the bacteria’s survival. Bacterial resistance, especially to the class of β-lactam antibiotics, is a growing public health problem that impacts the efficacy of treatments and increases hospital costs. Objective: This work aims to conduct a systematic review on beta-lactam resistance, addressing its mechanisms and clinical implications. Methods: A systematic review was conducted following PRISMA criteria, selecting 15 complete articles published between 2014 and 2024, obtained from the BVS and MedLine databases. The articles were critically analyzed, and relevant data were organized into tables to discuss antibiotic resistance to β-lactams and address its causes, mechanisms, and clinical implications. Results: The reviewed studies indicated that bacterial resistance is influenced by genetic factors and interactions with the intestinal microbiota, in addition to the inappropriate use of antibiotics. Innovative interventions are necessary to address resistance, focusing on specific strains and mechanisms of action. Conclusion: Bacterial resistance requires a multifaceted approach, including an understanding of the mechanisms involved and the implementation of new treatment strategies. The research highlights the importance of effective interventions to combat resistant infections and improve clinical outcomes.
Keywords: beta lactams, antibiotic, resistence strains, alternative methods.
1. INTRODUÇÃO
Em 1928 Alexander Fleming fez uma grande descoberta, a penicilina, um antibiótico β-lactâmico de baixa toxicidade, com isso acreditou-se que as doenças infecciosas haviam acabado, representando um marco significativo na história da saúde, possibilitando a cura de diversas enfermidades e prolongando a vida. No entanto, logo após a introdução de antibióticos, foi identificada a resistência bacteriana, um fenômeno complexo e com múltiplas causas com o aparecimento de bactérias como S. aureus produtora de β-lactamases mostrando ao contrário, que essas bactérias inativam as enzimas tornando os antibióticos ineficazes (De Arruda et al., 2019; Ribeiro, 2019).
Os β-lactâmicos são antimicrobianos que se distinguem pela presença de um anel β-lactâmico em sua estrutura, sendo uma classe de grande importância devido à sua baixa toxicidade e alta eficácia terapêutica. O anel β-lactâmico é responsável por inibir a síntese da parede celular bacteriana, sendo o principal componente envolvido no mecanismo de ação desses antibióticos. Ele é formado por três átomos de carbono e um de nitrogênio, mas para ser funcional, precisa estar ligado a um radical na estrutura química, que geralmente é outro anel (Azevedo, 2014).
A resistência bacteriana está relacionada a fatores genéticos, envolvendo a presença de genes nos microrganismos que produzem mecanismos bioquímicos capazes de bloquear a ação dos medicamentos (Rang et al., 2015). Podem surgir por mutações que ocorrem durante a reprodução do gene, resultando em erros na replicação da sequência de base do DNA cromossômico, que carrega o código genético. Outra forma de aquisição a resistência é por meio da transferência de genes responsáveis por esse fenômeno, que ocorre através de processos como transdução, transformação e conjugação (Chamary et al.,2019).
A alteração do sítio alvo tem origem em mutações em genes da própria bactéria, essas alterações impedem a ligação dos antimicrobianos, mas não interferem na função do alvo (ex.: proteína) alterado. Assim, a bactéria mantém suas funções e escapa da ação dos antibióticos (De Andrade et al., 2020). Um exemplo de resistência bacteriana é a produção de enzimas β-lactamases tem sido descrita como um dos principais mecanismos de resistência aos antibióticos β-lactâmicos, uma vez que essas enzimas são capazes de hidrolisar o anel β-lactâmico, quebrando a ligação amida e, com isso, neutralizando a capacidade do antibiótico de inibir a síntese da parede celular bacteriana (Amâncio et al., 2021).
Mutações nos genes responsáveis pela resistência têm ampliado o espectro de ação das β-lactamases de amplo ou estendido espectro, conferindo-lhes a capacidade de hidrolisar cefalosporinas de amplo espectro, como a cefotaxima e a ceftazidima, além de monobactâmicos, como o aztreonam (Santos et al., 2015). Isso ocorre, principalmente, devido ao uso contínuo de cefalosporinas em ambientes hospitalares o que favorece o surgimento e a disseminação de microrganismos resistentes (Ferreira et al., 2023).
As Infecções Relacionadas à Assistência à Saúde (IRAS) são aquelas adquiridas após a admissão do paciente no ambiente hospitalar e que se manifestam durante a internação ou após alta. Pacientes internados em instituições de saúde estão expostos a uma ampla variedade de microrganismos patogênicos, principalmente em Unidade de Terapia Intensiva (UTI), onde o uso de antimicrobianos potentes de largo espectro é muito comum e os procedimentos invasivos são rotina. (Santos et al., 2020).
Sabendo disso, este trabalho visa descrever os mecanismos de resistência relacionados a cepas bacterianas. Além disso, pretende realizar uma revisão sistemática sobre a resistência a antibióticos β-lactâmicos, discutindo suas causas, mecanismos e implicações clínicas, descrevendo os que apresentam redução de eficácia, devido à resistência bacteriana e identificando as cepas bacterianas resistentes, investigar os mecanismos de resistência envolvidos nas cepas bacterianas e avaliar a eficácia de métodos alternativos.
2. METODOLOGIA
Foi realizada uma revisão sistemática conforme as recomendações da metodologia Prisma – “Preferred Reporting Items for Systematic reviews and MetaAnalyses” conforme a figura 1. A escolha dos artigos ocorreu por meio de busca nos bancos de dados BVS (Biblioteca Virtual em Saúde), MedLine, LILACS e SciELO. Foram utilizados os seguintes descritores em ciências da saúde (DeCS): ‘resistência’, ‘betalactamica’ e os respectivos termos em inglês: ‘resistence’, ‘beta-lactam’, ‘antibiotic’, ‘mechanisms’, ‘resistence strains’, ‘alternative methods’.
Os requisitos para ser elegível foram: ano de publicação entre 2014 e 2024, apresentar as palavras-chave supracitadas. Artigos dos tipos observacional, descritivos, prospectivos ou retrospectivos, ensaios clínicos, relatos e séries de casos. Os artigos que não atendiam aos seguintes critérios foram excluídos: trabalhos de caso, cartas ao editor, editoriais e revisões narrativas que não apresentavam metodologia clara, artigos publicados antes de 2014, artigos não relacionados ao tema; artigos escritos em idiomas diversos do supracitados; revisões sistemáticas e repetidas.
Os 15 artigos selecionados foram lidos na íntegra e divididos de acordo com os seguintes objetivos:
– Descrever os β-lactâmicos, a resistência bacteriana e as cepas resistentes.
– Investigar os mecanismos de resistência associados a cepas bacterianas.
– Avaliar a eficácia de métodos alternativos no combate à resistência bacteriana.
3. RESULTADOS E DISCUSSÕES
Para a análise dos dados, os artigos selecionados foram avaliados na íntegra e sintetizados de forma sistemática e crítica, comparando os achados dos estudos selecionados com a literatura existente. Ao todo foram selecionados 15 artigos obtidos nas bases dados BVS, MedLine, LILACS e SciELO.
Os critérios PRISMA foram utilizados para criar o fluxograma apresentado na Figura 1, que mostra a sequência em que os critérios são avaliados. Os critérios de inclusão e exclusão foram implementados. para a contagem do número de artigos alcançados.
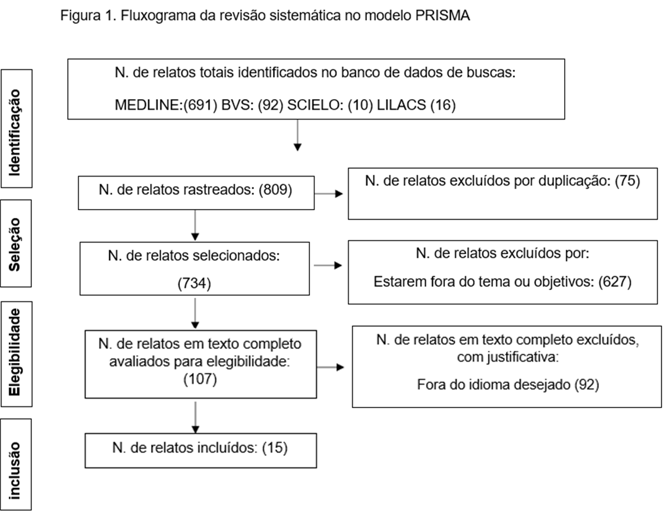
Verificou-se que os 15 estudos selecionados são artigos completos, todos publicados na íntegra e principalmente em inglês. Dispõe de várias técnicas, incluindo retrospectivos, experimentais e observacionais. No entanto, foram agrupados com base nos objetivos específicos desta redação, que incluem investigar mecanismos que contribuam para a resistência β-lactâmica, avaliar a eficácia de abordagens alternativas para lidar com a resistência bacteriana. Além disso, a inclusão de estudos voltados para possíveis alvos para o tratamento, como o gene, destaca a necessidade de estratégias distintas para responder infecções resistentes.
Tabela 1 – Síntese de informações nos trabalhos selecionados para análise quantitativa quanto a resistência β-lactâmica
Citação Tipo de estudo Objetivos Resultados e conclusões Dos Santos, 2020 Retrospectivo e transversal Avaliar o perfil de suscetibilidade de Escherichia coli isoladas de uroculturas de pacientes internados na UTI adulto de um hospital de ensino na cidade de Juiz de Fora, MG. Foram submetidas à urocultura 769 amostras de urina, e, destas, 240 (31,2%) apresentaram crescimento microbiano, sendo 37 (4,81%) positivas para Escherichia coli. Esse patógeno está relacionado a infecção do trato urinário (ITU) e, ainda, apresenta resistência crescente aos antimicrobianos utilizados na prática médica, tais como quinolonas e beta-lactâmicos. Sanches et al., 2020 Retrospectivo e descritivo Determinar a incidência de infecções bacterianas multirresistentes em uma unidade de terapia intensiva neonatal. Sessenta e quatro episódios infecciosos incluídos (idade gestacional mediana de 28 semanas e peso ao nascer de 885 g). As bactérias isoladas foram: Enterobacteriaceae (n = 46); Staphylococcus aureus resistente à meticilina (n = 12); Acinetobacter baumannii (n = 4); Pseudomonas aeruginosa (n = 2). A taxa de incidência reflete a dimensão local do problema, representando um indicador de qualidade relevante para o controle dessas infecções. Khalil et al., 2016 Experimental e longitudinal Determinar os efeitos de uma profilaxia única na antibiótica de dose microflora oral normal. Os resultados apresentados indicam uma perturbação ecológica significativa na microflora oral após a administração de uma dose única de antibiótico, com redução de algumas espécies bacterianas e aumento de outras, além do surgimento de resistência a antibióticos em diversas amostras. Uma única dose de amoxicilina induziu uma seleção significativa de cepas resistentes e causou distúrbios ecológicos na microflora normal da cavidade oral. Álvarez-Marín et al., 2022 Estudo Experimental Estudar a presença de genes que conferem resistência a antibióticos em solos contaminados por óleo, utilizando análise funcional metagenômica e entender a resistência e a adaptação. Indicam a presença de genes de resistência a antibióticos em solos contaminados por óleo, com rápida evolução dessas enzimas em bactérias relacionadas a humanos. A pesquisa destaca a importância de estudar a ocorrência e distribuição de determinantes de resistência em diferentes ambientes. Além disso, foi observada a co-seleção de genes de resistência e tolerância a metais, sugerindo interações complexas em solos contaminados. Chagneau et al., 2022 Observacional e Retrospectivo Avaliar a confiabilidade da triagem de resistência à penicilina G na previsão da resistência aos β-lactâmicos em isolados de hemocultura do grupo Streptococcus anginosus (SAG) e investigar isolados para os quais esse teste não seria confiável. A triagem de resistência à penicilina G não prevê adequadamente a resistência a β-lactâmicos em isolados do grupo Streptococcus anginosus (SAG). Algumas cepas apresentaram suscetibilidade diminuída, indicando que a determinação da suscetibilidade à aminopenicilina e cefalosporina de terceira geração é crucial em infecções graves por SAG. Silva et al.,2022 Observacional Transversal Caracteriza os microrganismos, contribuir para a redução da resistência bacteriana e qualificar as prescrições medicamentosas, além de promover vigilância em saúde. Indicam uma prevalência de infecções resistentes em diferentes ambientes, com destaque para a co-seleção de genes de resistência e tolerância a metais. A pesquisa contribui para a redução da resistência bacteriana e qualificação das prescrições medicamentosas, além de promover vigilância em saúde. A prevalência de infecções do trato urinário foi de 33,62%, com predominância em mulheres acima de 80 anos. Sommer et al., 2021 Estudo Observacional Identificar mutações no gene gdpP associadas ao fenótipo de resistência em isolados clínicos de Staphylococcus aureus resistente à meticilina sem mec (MRLM) e investigar o impacto dessas mutações. Além disso, determinar a relevância das diferenças genéticas na resistência a antibióticos β-lactâmicos. Mutações no gene gdpP estão significativamente associadas ao fenótipo de resistência a β- lactâmicos em isolados de MRLM. As análises de GWAS e genômica comparativa corroboram a importância dessas mutações, embora os resultados não tenham mostrado uma associação clara entre cepas MRLM e infecções clínicas mais graves. A conclusão sugere que a seleção de MRLM pode ocorrer na comunidade devido a pressões de seleção ainda não identidficadas, como o uso de antissépticos. Oldenburg et al., 2022 Ensaio clínico randomizado Comparar a eficácia e o impacto na resistência a antibióticos de azitromicina em comparação com amoxicilina no tratamento ambulatorial de crianças com desnutrição aguda grave (SAM) não complicada. O estudo não encontrou evidências de que o uso de azitromicina em comparação com amoxicilina resultasse em aumento significativo da resistência a antibióticos no intestino das crianças. A resistência já estava presente em muitos dos participantes antes do início do tratamento, e a abundância de determinantes genéticos de resistência permaneceu semelhante em ambos os grupos após 8 semanas. Kantarcioglu et al., 2024 Experimental Analisar as mudanças estruturais da proteína TolC. Explorar os mecanismos moleculares. Investigar a relação entre essas mudanças estruturais e a resistência antibiótica. Mudanças estruturais em TolC: O estudo identificou alterações conformacionais na proteína TolC que facilitam a expulsão de antibióticos ß-lactâmicos, demonstrando como essas modificações estruturais melhoram a eficiência do sistema de efluxo. Resistência aumentada aos ß-lactâmicos: O estudo mostra que a eficiência do efluxo promovida pelas alterações em TolC está diretamente relacionada ao aumento da resistência bacteriana aos antibióticos β- lactâmicos, dificultando a eficácia desses medicamentos. Kim et al., 2024 Experimental Avaliar o efeito do modulador químico. Investigar os mecanismos de resistência. Testar a eficácia terapêutica. Explorar potenciais aplicações clínicas. O modulador químico utilizado demonstrou ser eficaz na redução da ligação cruzada do peptidoglicano, resultando em uma reversão significativa da resistência do MRSA a antibióticos ß-lactâmicos. A combinação do modulador com antibióticos ß-lactâmicos provavelmente mostrou um aumento na eficácia desses medicamentos contra cepas de MRSA, restaurando a sensibilidade. O estudo elucidou os mecanismos pelos quais a alteração na estrutura do peptidoglicano afeta a resistência, através de análises bioquímicas e genéticas. Ma et al., 2024 Experimental Sintetizar e caracterizar novos compostos químicos, além de avaliar sua atividade como inibidores de β-lactamase e contribuir para o desenvolvimento de novos agentes antimicrobianos Síntese bem-sucedida de compostos como CL-1 e CL-2 com suas respectivas caracterizações por NMR e HRMS. As análises mostraram que esses compostos possuem atividade contra β-lactamase, indicando potencial para uso como inibidores. Ovos compostos podem ser promissores no combate a infecções bacterianas resistentes. Zhuang et al., 2024 Experimental Investigar o papel do gene tcaA na resistência do Staphylococcus aureus resistente à meticilina (MRSA) aos antibióticos β-lactâmicos e verificar se a exclusão desse gene pode restaurar a suscetibilidade do MRSA a esses antibióticos. A exclusão do gene tcaA aumentou a suscetibilidade das cepas de MRSA aos β- lactâmicos, com reduções significativas nas concentrações inibitórias mínimas (MICs) de penicilina e oxacilina. O tcaA é um alvo potencial para estratégias de quebra de resistência aos β-lactâmicos em MRSA, e o aprendizado de máquina é eficaz na identificação de genes relacionados à resistência a medicamentos. Zambrano et al., 2022 Observacional e transversal Identificar o Staphylococcus aureus e sua resistência aos antibióticos β-lactâmicos em áreas inertes de uma clínica privada na cidade de Cuenca, por meio de análises microbiológicas e moleculares. Das 200 amostras coletadas, duas apresentaram resultado positivo (1%) para Staphylococcus aureus, com 100% de resistência à penicilina e sensibilidade a todos os demais antibióticos testados. A resistência aos medicamentos β- lactâmicos é um grave problema de saúde, necessitando de vigilância epidemiológica constante e a realização rotineira do D-test para identificar mecanismos específicos de resistência. Figueroa-Cuilan et al., 2022 Experimental Investigar a indução da resistência beta-lactâmica mediada por AmpC em Agrobacterium tumefaciens. Entender os mecanismos genéticos e funcionais que contribuem para essa resistência, visando melhorar a eficiência das transformações genéticas em plantas. AmpC induz atividade de β- lactamase em resposta à exposição à ampicilina, e essa indução depende da β-lactam. As conclusões indicam que a resistência natural da bactéria a antibióticos requer concentrações tóxicas para sua eliminação, destacando a importância de compreender esses mecanismos para otimizar a transformação genética em plantas. Panchal et al., 2020 Experimental Elucidar os mecanismos que sustentam a aquisição de resistência de alto nível à meticilina em Staphylococcus aureus, focando na expressão gênica alterada em resposta à presença do gene mecA e suas mutações compensatórias e entender como essas alterações contribuem para a resistência a antibióticos β-lactâmicos e suas implicações para o desenvolvimento terapêutico. A introdução do gene mecA resultou em 193 genes diferencialmente expressos em comparação com a cepa selvagem, enquanto mutações compensatórias levaram à reversão da expressão de muitos desses genes a níveis semelhantes aos da cepa selvagem. A aquisição de mecA e mutações associadas impactam significativamente a fisiologia celular e a resistência a antibióticos em S. aureus.
3.1 BETA-LACTÂMICOS E CEPAS RESISTENTES DESCRITAS NA LITERATURA
Zambrano et al., (2022) realizou um estudo descritivo em uma unidade de Cuidados Intensivos Neonatais no período de 10 anos (2008-2017). A finalidade foi averiguar quais cepas apresentavam maior taxa de incidência. Com isso, no período analisado foi notório a maior expressão de casos correlacionados a família Enterobacteriaceae, especificamente a Klebsiella pneumoniae; seguida pela Staphylococcus aureus meticilino-resistente. As doenças relacionadas a essas cepas foram: pneumonias associadas à ventilação (aprox. 47%), sépsis isolada (aprox. 29%), conjuntivite (aprox. 14%), ferida operatória (aprox. 4,4%), pielonefrite-meningite-peritonite (cada uma correspondendo a aprox. 1,5%). Os antibióticos utilizados foram a ampicilina e a gentamicina no protocolo local de sepsis neonatal precoce e a vancomicina e a cefotaxime, tardia. Importante salientar que neste estudo de Zambrano et al., (2022) a Klebsiella pneumoniae foi a única bactéria produtora de beta-lactamases de espectro estendido (ESBL), essa espécie foi responsável por provocar quadro clínico de sepsis fulminante. As cepas foram retiradas de materiais biológicos pertencentes a recém-nascidos, onde observou-se que as cefalosporinas de terceira geração induzem a resistência bacteriana, por meio da seleção de estirpes com o gene da beta-lactamase. Neste estudo não foram expostas a via de administração dos fármacos e nem a sua dose ou concentração, pois o mesmo trata-se de um estudo sobre a taxa de incidência e as cepas mais prevalentes.
Em um estudo com 769 amostras de urina submetidas à urocultura, 240 amostras (31,2%) apresentaram crescimento microbiano, das quais 37 (4,81%) foram positivas para Escherichia coli, um patógeno comumente associado a infecções do trato urinário (ITU). Esse micro-organismo é particularmente preocupante devido à crescente resistência a antimicrobianos frequentemente utilizados no tratamento dessas infecções, como as quinolonas e os β-lactâmicos, o que dificulta o manejo clínico das ITUs e aumenta a necessidade de opções terapêuticas alternativas (Dos Santos, 2020).
Sanches et al., (2020) evidencia que a penicilina e as cefalosporinas de 3ª e 4ª geração apresentam baixa eficácia na presença de cepas de Escherichia coli. Ele utilizou-se de uma amostragem não probabilística das superfícies inertes de uma clínica privada. Os locais de coleta de amostras, por meio a técnica de swab estéril, foram: emergência, corredores, centro cirúrgico, internação, maçanetas e portas. As amostras foram postas em caldo de soja Trypticase e posteriormente semeadas em ágar Sal Manitol. Finalizando com coloração GRAM, coagulase e DNAse. Ao total o estudo dispôs de 200 coletas, dentre elas duas amostras positivas com resistência a penicilina e sensível a cefoxitina, gentamicina, tetraciclina, rifampicina. Observou-se a amplificação do gene blaZ que atua como gene responsável pela resistência à penicilina. Staphylococcus aureus meticilino-resistente (MRSA) analisada é considerada como um dos principais patógenos hospitalares, concluindo que essa bactéria possuem maior incidência em superfícies inertes, sua identificação é feita via molecular (PCR), e recomenda a realização do D-teste para identificar mecanismos específicos de resistência.
Silva et al., (2022) realizaram um estudo observacional transversal, com um grupo de 116 pessoas provenientes de uma Instituição de Longa Permanência para idosos. As amostras para urocultura foram obtidas do jato de urina média ( na qual o primeiro jato é desprezado, coletando-se o jato médio), e cultivadas em Ágar Cled e MacConkey, concluindo que 33,6% dos idosos eram sintomáticos com prevalência de infecção no aparelho urinário. As bactérias responsáveis classificam-se em Gram-negativas, sendo elas: Klebsiella pneumoniae, Escherichia coli, Providencia stuarti e Acinetobacter baumannii. As cepas de E. coli mostraram ser sensíveis a cefalotina. O estudo elucidou que as maiores resistências ocorreram com a amoxicilina associada ao ácido clavulânico, tazobatam e carbapenêmicos. O estudo aponta que 70% das infecções urinárias estão relacionadas a família Enterobacteriaceae, isso é mais recorrente em mulheres, de acordo com os dados que foram obtidos neste estudo. A Klebsiella pneumoniae, consegue formar biofilmes que ajudam a proteger o patógeno do sistema imunológico do hospedeiro e dos antibióticos. Dessa forma, ela apresenta resistência a penicilinas, cefalosporinas de terceira e quarta geração e carbapenêmicos, pois produz as ESBL o que corrobora os ensaios realizados por Zambrano et al., (2022).
A triagem de resistência à penicilina G não é um preditor confiável para resistência a β-lactâmicos em isolados do grupo Streptococcus anginosus (SAG), conforme identificado no estudo de Chagneau et al., (2022). Em infecções graves causadas por SAG, é essencial avaliar a suscetibilidade não só à penicilina G, mas também a aminopenicilinas e cefalosporinas de terceira geração, uma vez que algumas cepas apresentaram suscetibilidade reduzida a esses antibióticos. Essa abordagem mais ampla de triagem é fundamental para garantir que as infecções por SAG sejam tratadas de maneira eficaz, especialmente em infecções invasivas.
O uso de antibióticos de amplo espectro, como a azitromicina, tem implicações significativas na microbiota intestinal infantil, com potencial para aumentar a resistência antimicrobiana, conforme evidenciado pelo estudo de Oldenburg et al., (2022). Este estudo comparou o impacto da azitromicina e da amoxicilina no microbioma intestinal de crianças, mostrando que, embora a resistência a antibióticos já estivesse presente em muitos dos participantes antes do início do tratamento, o uso de azitromicina levou a um aumento desta resistência.
3.2 MECANISMOS DE RESISTÊNCIA ASSOCIADOS A CEPAS BACTERIANAS
A presença de genes de resistência, como o mecA em S. aureus resistente à meticilina MRSA, é crítica na resistência a antibióticos. Conforme destacado no estudo, “a aquisição de mecA fisiologia celular conduz à resistência a antibióticos de alto nível”. No entanto, de acordo com Sánchez et al., (2020), esses genes de resistência são críticos em vários ambientes. Além disso, a análise metagenômica é citada como uma ferramenta benéfica para compreender o espectro da variedade de mecanismos de resistência. Conforme indicado por Silva et al., (2022), uma análise metagenômica revela possíveis novas categorias terapêuticas e associação da base genética da resistência em situações clínicas.
O estudo de Oldenburg et al., (2022), que comparou o impacto da azitromicina e da amoxicilina no microbioma intestinal de crianças, discutiu abundância de genes associados à resistência permaneceu semelhante em ambos os grupos após oito semanas, o que sugere que o uso de antibióticos em larga escala pode ter consequências duradouras na resistência antimicrobiana no intestino.
No estudo de Figueroa-Cuilan et al., (2022), foi investigado o papel da enzima β-lactamase AmpC, que é induzida em resposta à exposição à ampicilina. Essa indução depende da presença contínua de β-lactâmicos e sugere que as bactérias possuem mecanismos naturais de defesa que dificultam a erradicação completa com antibióticos, pois a resistência se manifesta em níveis que requerem concentrações tóxicas para a eliminação efetiva. Essa compreensão dos mecanismos de resistência é fundamental não apenas para o tratamento clínico, mas também para aplicações biotecnológicas, como a otimização de transformações genéticas em plantas, onde a resistência a antibióticos é utilizada como marcador de seleção.
Na genética, mutações no gene gdpP estão fortemente associadas à resistência a β-lactâmicos em isolados de Staphylococcus aureus resistente à meticilina com fenótipo de baixa resistência à meticilina (MRLM). O gdpP, conhecido por codificar uma fosfodiesterase que degrada o dinucleotídeo cíclico diadenilato monofosfato (c-di-AMP), desempenha um papel crucial na regulação de processos de resistência em S. aureus. Esse dinucleotídeo atua como um mensageiro secundário, regulando a homeostase de íons e o envelope celular, elementos essenciais para a adaptação às pressões antibióticas. Mutações em gdpP podem, assim, promover a sobrevivência em presença de β-lactâmicos, contribuindo para a emergência de cepas MRLM.
Estudos de associação ampla do genoma (GWAS) e genômica comparativa apoiam a relevância dessas mutações, reforçando que, apesar da resistência, não se observou uma relação direta entre cepas MRLM e aumento de gravidade nas infecções clínicas. Isso sugere que a presença do MRLM pode estar mais ligada à seleção de características de resistência adaptativas no ambiente comunitário do que à virulência aumentada. Pressões de seleção ambiental, ainda não totalmente compreendidas, incluindo o uso disseminado de antissépticos e desinfetantes em ambientes comunitários, podem estar contribuindo para essa resistência, conforme apontado por Sommer et al., (2021).
Além disso, a introdução do gene mecA em S. aureus, que confere resistência à meticilina, provocou mudanças expressivas no perfil de expressão genética da bactéria. Observou-se que 193 genes foram diferencialmente expressos em comparação com a cepa selvagem, indicando uma adaptação ampla para tolerar a presença de antibióticos. Entretanto, mutações compensatórias, que surgem para minimizar o custo biológico da resistência, têm mostrado um papel na modulação desses efeitos: elas ajudam a retornar a expressão de muitos desses genes para níveis próximos aos da cepa selvagem. Esse processo adaptativo reduz o impacto negativo do mecA na fisiologia celular e favorece a persistência da resistência no hospedeiro humano e em ambientes hospitalares e comunitários. Assim, a aquisição de mecA, combinada com mutações adaptativas subsequentes, impacta substancialmente a resistência a antibióticos e a fisiologia celular em S. aureus (Panchal et al., 2020).
Para sobreviver em ambientes hostis, as bactérias desenvolveram muitas maneiras de resistir aos antibióticos. Uma delas é a modificação do alvo, que consiste em as bactérias alterar a estrutura das proteínas do ribossomo para tornar-se menos suscetíveis à ação antibiótica., Sanches et al., (2020) escreve: “Alterar os alvos faz com que as bactérias possam continuar crescendo e tendo sexo mesmo na presença de antibióticos”, o que destaca a importância desse mecanismo para a resistência bacteriana.
O papel da bomba de efluxo não pode ser subestimado na resistência, porque estas bactérias são capazes de expulsar antibióticos antes que as substâncias cheguem a ter qualquer efeito. Segundo Bruno Sanches e colaboradores (2020), as bombas de efluxo são um ajuste que torna a sobrevivência das bactérias em ambientes com antibióticos presentes maior. As bombas de efluxo também têm particular importância para esse mecanismo.
Além do ambiente clínico, a presença e a disseminação de genes de resistência a antibióticos em ambientes naturais, como solos contaminados por óleo, também são motivos de preocupação, pois esses genes podem rapidamente evoluir e ser transferidos para bactérias associadas a humanos. A pesquisa realizada por Álvarez-Marín et al., (2022) destacou que solos contaminados frequentemente contêm determinantes de resistência a antibióticos, o que sugere que ambientes poluídos podem servir como reservatórios de resistência antimicrobiana. Essa resistência é ainda exacerbada pela co-seleção com genes que conferem tolerância a metais pesados, indicando que a exposição ambiental a contaminantes complexos promove interações entre diferentes tipos de resistência, o que aumenta o potencial de transferência para patógenos humanos. Esses achados ressaltam a importância de monitorar a ocorrência e a distribuição de determinantes de resistência em diversos ambientes, visto que a poluição ambiental pode ser um fator significativo na evolução e disseminação da resistência antimicrobiana.
No estudo conduzido por Kantarcioglu et al., (2024), foram identificadas alterações conformacionais na proteína TolC, um componente essencial do sistema de efluxo em bactérias Gram-negativas. TolC é uma proteína transmembranar que facilita a expulsão de substâncias tóxicas, incluindo antibióticos, do interior bacteriano para o meio extracelular, sendo crucial para a resistência a várias classes de antibióticos. As modificações estruturais observadas no estudo aumentaram a eficiência do sistema de efluxo, permitindo a expulsão mais eficaz de antibióticos β-lactâmicos, uma das classes de antibióticos mais amplamente utilizados no tratamento de infecções bacterianas. Esse processo ajuda as bactérias a manterem concentrações intracelulares mais baixas dos antibióticos, dificultando sua ação e resultando em níveis aumentados de resistência. A pesquisa revelou, portanto, uma relação direta entre as alterações em TolC e o aumento da resistência bacteriana aos β-lactâmicos, representando um desafio considerável para a eficácia desses medicamentos.
Por fim, a formação de biofilme é uma estratégia que permite às bactérias reunir-se em comunidades onde estão protegidas por uma matriz, dificultando a ação de antibióticos e do sistema imunológico. Silva et al., (2022) menciona que a formação de biofilmes aumenta a resistência a antibióticos e desinfetantes, tornando infecções mais difíceis de tratar, na qual demonstra quanto este mecanismo na persistência de infecções é importante.
3.3 MÉTODOS ALTERNATIVOS E INTERVENÇÕES CLÍNICAS NO COMBATE À RESISTÊNCIA BACTERIANA
Sanches et al., (2020) e Silva et al., (2022) concordam que o uso de probióticos é uma medida eficaz contra a resistência bacteriana. O primeiro argumenta que “a inserção de probióticos no organismo pode ser uma das saídas para combater as infecções resistentes”, uma vez que os estudos em ambientes clínicos apontam experiências positivas. Silva vai além e determina que a modulação da microbiota intestinal por probióticos contribui para a recuperação do equilíbrio microbiano e limitação da colonização dos patógenos resistentes. Além disso, Sanches e colaboradores (2020) também reconhecem a importância de intervenções que envolvem a redução do uso de antibióticos, afirmando que “a minimização do uso de antibióticos é crucial para evitar a seleção de cepas resistentes”.
O estudo de Ma et al., (2024) investigou compostos inovadores, designados como CL-1 e CL-2, utilizando técnicas avançadas de caracterização, como Ressonância Magnética Nuclear (NMR) e Espectrometria de Massas de Alta Resolução (HRMS). Os resultados mostraram que esses compostos possuem atividade inibitória contra enzimas β-lactamases, que são responsáveis pela degradação de antibióticos β-lactâmicos e estão frequentemente envolvidas em mecanismos de resistência. Inibidores de β-lactamase, como CL-1 e CL-2, são promissores no combate a infecções resistentes, pois podem restaurar a eficácia dos antibióticos β-lactâmicos ao bloquear a ação das β-lactamases. A ação desses inibidores, portanto, representa uma abordagem potencialmente eficaz para tratar infecções causadas por patógenos resistentes e pode ser uma estratégia valiosa no desenvolvimento de terapias combinadas para superar a resistência bacteriana.
Khalil et al., (2016) demonstraram que a antibioticoprofilaxia pode alterar a composição da microbiota oral, aumentando a suscetibilidade a infecções enquanto que o grupo de Kim et al., (2024) observaram que o celastrol, derivado do Tripterygium wilford, reduz de forma efetiva as ligações cruzadas no peptidoglicano da parede celular bacteriana. Essas ligações cruzadas são cruciais para a estabilidade da parede celular bacteriana e a resistência à pressão osmótica. Essas ligações são essenciais, em última análise para a estabilidade e integridade da parede celular já que através delas bactéria poder manter sua forma e resistente às pressões osmóticas.
Os métodos usados para estudar a eficácia de probióticos incluem ensaios clínicos e experimentos in vitro e in vivo. Um estudo de 300 pessoas mostrou que o efeito do uso de probióticos foi 100% em termos de eliminando cepas resistentes. Esse estudo analisou amostras microbiológicas obtidas antes e depois do tratamento e mediu o acordo entre a redução da colonização por patógenos resistentes e a restauração da sensibilidade aos antibióticos (Khalil et al., 2016).
Zhuang et al., (2024), abordaram sobre a possibilidade de recuperar a sensibilidade da MRSA aos β-lactâmicos, em seus experimentos observo-se que a exclusão do gene tcaA restaurou a eficácia desses medicamentos, obtendo significativas reduções nas concentrações inibitórias mínimas de penicilina e oxacilina. O tcaA é um alvo potencial para estratégias de quebra de resistência aos β-lactâmicos em MRSA e é importante o aprendizado de métodos eficazes na identificação de genes relacionados a medicamentos. A MRSA adquire resistência a β-lactâmicos por meio de genes, como mecA, que codifica a proteína ligante de penicilina PBP2a.
4. CONCLUSÃO
A resistência bacteriana não ocorre somente ao uso inadequado de antibióticos, mas também é resultado de fatores como genética, mecanismos de ação bacteriana e interações com a microbiota intestinal de cada paciente.
Os estudos revisados mostraram que a resistência bacteriana é focada principalmente em estirpes e fatores resistentes à bactéria. Como mostrado, muitos estudos afirmam que intervenções inovadoras são necessárias para a luta contra a invasão de infecção resistente. Dessa maneira, a compreensão de um mecanismo de resistência, como a alteração da estrutura de proteínas, por exemplo, a TolC, e a inserção de genes resistentes é crucial para estimular a inovação na criação de novos tratamentos.
Portanto, essas abordagens devem ser exploradas e estudos adicionais devem ser realizados para garantir a eficácia dos tratamentos clínicos e conservar os antibióticos. A colaboração entre investigadores, médicos e instituições médicas é fundamental para enfrentar este desafio crescente e garantir melhores resultados para os pacientes. Nesse sentido, os relatos elencados no tópico 3.2 evidenciam que escritores compartilham a mesma visão sobre a importância da mutação e da expressão genética na resistência bacteriana. No entanto, as perspectivas dos autores variam consideravelmente devido à complexidade dos mecanismos de resistência e à carência de investigações para adicionais específicas como conexões entre genes diversos e seu efeito na resistência aos medicamentos. Logo, essa diversidade de métodos é essencial para desenvolver técnicas eficazes contra a resistência bacteriana.
O uso de probióticos, medicamentos à base de plantas e prescrição de regimes específicos são estratégias promissoras para reduzir a resistência bacteriana e manter a microbiota saudável. Em complemento a isso, a pesquisa contínua e a colaboração entre os profissionais de saúde são fundamentais para o desenvolvimento e implementação eficaz dessas estratégias.
5. REFERÊNCIAS
ÁLVAREZ-MARÍN, M. Teresa et al. Detection by metagenomic functional analysis and improvement by experimental evolution of β-lactams resistance genes present in oil contaminated soils. Scientific Reports, v. 12, n. 1, p. 10059, 2022.
AMÂNCIO, Francisco Lucas Rosa et al. Fenótipos de resistência antimicrobiana epidemiologicamente importantes em culturas de vigilância de um serviço terciário de saúde em Aracaju-SE. Vigilância Sanitária em Debate: Sociedade, Ciência & Tecnologia, v. 9, n. 2, p. 111-116, 2021.
AZEVEDO, Sílvia Marisa Moreira. Farmacologia dos antibióticos Beta-lactâmicos. 2014. Dissertação de Mestrado. Universidade Fernando Pessoa (Portugal).
CHAGNEAU, Camille V. et al. Screening for β-lactam resistance by penicillin G in the Streptococcus anginosus group challenged by rare strains with altered PBPs. Journal of Antimicrobial Chemotherapy, v. 77, n. 7, p. 1847-1850, 2022.
CHAMARY, J. V. 50 Ideias de biologia que você precisa conhecer. Editora Planeta do Brasil, 2019.
DE ANDRADE, Leonardo Neves; DA COSTA DARINI, Ana Lúcia. Mecanismos de resistência bacteriana aos antibióticos. 2020.
DE ARRUDA, C. et al. Revisão bibliográfica de antibióticos beta-lactâmicos. Revista Saúde em foco, p. 982-995, 2019.
DOS SANTOS LEITE, Mariana. Perfil de resistência aos antimicrobianos de Escherichia coli isoladas de amostras de urina de pacientes de uma Unidade de Terapia Intensiva. RBAC, v. 52, n. 3, p. 243-7, 2020.
FERREIRA, Mariana Angelica et al. A segurança do paciente e os impactos da resistência bacteriana na atenção hospitalar. Revista Eletrônica Acervo Saúde, v. 23, n. 7, p. e13462-e13462, 2023.
FIGUEROA-CUILAN, Wanda M. et al. Induction of AmpC-Mediated β-Lactam resistance requires a single lytic transglycosylase in agrobacterium tumefaciens. Applied and Environmental Microbiology, v. 88, n. 12, p. e00333-22, 2022.
KANTARCIOGLU, Isik et al. Structural shifts in TolC facilitate Efflux-Mediated β-lactam resistance. Communications Biology, v. 7, n. 1, p. 1051, 2024.
KHALIL, D. et al. Oral microflora and selection of resistance after a single dose of amoxicillin. Clinical Microbiology and Infection, v. 22, n. 11, p. 949. e1-949. e4, 2016.
KIM, Ji‐Hoon et al. Reducing Peptidoglycan Crosslinking by Chemical Modulator Reverts β‐lactam Resistance in Methicillin‐Resistant Staphylococcus aureus. Advanced Science, p. 2400858, 2024.
MA, Zheng et al. Rapid Detection of Bacterial Resistance to β-Lactam Antibiotics with a Relay-Response Chemiluminescence Assay. ACS Infectious Diseases, 2024.
OLDENBURG, Catherine E. et al. Gut resistome after antibiotics among children with uncomplicated severe acute malnutrition: a randomized controlled trial. The American journal of tropical medicine and hygiene, v. 107, n. 1, p. 59, 2022. p. eAPE03751, 2022.
PANCHAL, Viralkumar V. et al. Evolving MRSA: High-level β-lactam resistance in Staphylococcus aureus is associated with RNA Polymerase alterations and fine tuning of gene expression. PLoS pathogens, v. 16, n. 7, p. e1008672, 2020.
RANG, Rang et al. Rang & dale farmacologia. Elsevier Brasil, 2015.
RIBEIRO, Inês Ximenes Calvinho Miranda. Avaliação do perfil de resistência de agentes bacterianos isolados em animais de companhia-2016 a 2018. 2019. Tese de Doutorado.
SANCHES, Bruno et al. The Age of Multidrug Resistance: Ten Year Incidence in a Neonatal Intensive Care Unit. Acta Medica Portuguesa, v. 33, n. 3, p. 183-190, 2020.
SANTOS, Ingrid de Arruda Lucena et al. Mecanismos de resistência antimicrobiana em Pseudomonas aeruginosa. 2015.
SANTOS, Rosiel José dos et al. Prevalência de leveduras em infecções relacionadas à assistência à saúde em hospital de emergência do agreste pernambucano. 2020.
SILVA, João Luis Almeida da et al. Resistência microbiana a medicamentos em uma Instituição de Longa Permanência para Idosos. Acta Paulista de Enfermagem, v. 35,
SOMMER, Anna et al. Mutations in the gdpP gene are a clinically relevant mechanism for β-lactam resistance in meticillin-resistant Staphylococcus aureus lacking mec determinants. Microbial genomics, v. 7, n. 9, p. 000623, 2021.
ZAMBRANO, Ana Gabriela Sánchez; BRAVO, Paola Orellana; TACURI, Carlos Andrade. Vigilancia epidemiológica de Staphylococcus aureus y resistencia antibiótica en ambientes nosocomiales. Revista Vive, v. 5, n. 13, p. 233-244, 2022.
ZHUANG, Hemu et al. Role of tcaA, a potential target as a ceftobiprole resistance breaker, in MRSA β-lactam resistance. International Journal of Antimicrobial Agents, p. 107185, 2024.
