REGISTRO DOI: 10.5281/zenodo.11643750
Enzo Carmine Picchiello;
Nathália Agnes Sene André;
Thaís Ukstin Paiva Goulart Silva;
Poliana Maria de Souza
Resumo
Este trabalho de conclusão de curso visou à implementação de um sistema avançado de monitoramento genômico ambiental no Aeroporto Internacional de São Paulo/Guarulhos, focando na detecção e quantificação precisa de organismos patogênicos específicos: Neisseria meningitidis, Streptococcus pneumoniae, Haemophilus influenzae, e H. pylori. Utilizando protocolos padronizados e tecnologias de sequenciamento de última geração, o estudo mapeou a presença e distribuição desses microrganismos em um ambiente de alto tráfego, destacando as potenciais rotas de transmissão e avaliando a eficácia das práticas de limpeza e desinfecção atuais. Os resultados oferecem insights valiosos para a elaboração de estratégias mais efetivas de prevenção e controle de doenças infecciosas em espaços públicos, contribuindo significativamente para a saúde pública.
Keywords: Monitoramento genômico ambiental, Detecção de patógenos, Aeroporto Internacional de São Paulo/Guarulhos, Prevenção de doenças infecciosas.
Abstract
This thesis aimed at implementing an advanced environmental genomic monitoring system at São Paulo/Guarulhos International Airport, focusing on the accurate detection and quantification of specific pathogenic organisms: Neisseria meningitidis, Streptococcus pneumoniae, Haemophilus influenzae, and H. pylori. Employing standardized protocols and cutting-edge sequencing technologies, the study mapped the presence and distribution of these microorganisms in a high-traffic environment, highlighting potential transmission routes and assessing the effectiveness of current cleaning and disinfection practices. The findings provide valuable insights for developing more effective strategies for the prevention and control of infectious diseases in public spaces, significantly contributing to public health.
Keywords: Environmental genomic monitoring, Pathogen detection, São Paulo/Guarulhos International Airport, Infectious disease prevention.
1. INTRODUÇÃO
Este trabalho de conclusão de curso tem como objetivo o estudo de um sistema avançado de monitoramento genômico ambiental, com foco específico no Aeroporto Internacional de São Paulo/Guarulhos. Este local foi escolhido devido à sua importância como um dos principais eixos de tráfego aéreo no Brasil e na América Latina, tornando-o um local estratégico para o monitoramento de patógenos devido ao seu alto fluxo de passageiros internacionais (Danchin & Timmis, 2020).
A introdução do sequenciamento de nova geração (NGS) por Grabherr et al. (2011) expandiu as capacidades de análise microbiológica, permitindo uma visão detalhada da biodiversidade microbiana em ambientes complexos, como terminais de aeroportos. Essas tecnologias são cruciais para o monitoramento genômico ambiental, fornecendo meios eficazes para vigilância em tempo real e prevenção de surtos.
Danchin e Timmis (2020) discutiram a evolução contínua dos patógenos e as implicações para a vigilância global, destacando a necessidade de sistemas de monitoramento precisos e adaptáveis. Este requisito é especialmente crítico em ambientes de aeroportos, onde a rápida disseminação de variantes patogênicas pode impactar significativamente a saúde pública mundial.
Através do uso de tecnologias de sequenciamento genético de última geração, este estudo visa identificar e quantificar organismos patogênicos como Neisseria meningitidis, Streptococcus pneumoniae, Haemophilus influenzae, e H. pylori, que são conhecidos por representar significativas ameaças à saúde pública (Altschul et al., 1990; Grabherr et al., 2011).

Figura 1 – Illumina NextSeq® 550 desktop next-generation sequencing (NGS) system. Fonte: Altschul et al. (1990).
Esta pesquisa sublinhou a importância da colaboração interdisciplinar na biomedicina e na saúde pública, unindo conhecimentos em genética, microbiologia e epidemiologia para enfrentar desafios complexos na prevenção de doenças.
Com o aumento das ameaças de patógenos emergentes e reemergentes, o monitoramento genômico ambiental se torna uma ferramenta fundamental para a identificação rápida de riscos potenciais, permitindo intervenções mais efetivas e tempestivas (Weiss et al., 2019).
De tal modo, este estudo representa um avanço importante em direção a um sistema de saúde pública mais resiliente e adaptativo, capaz de responder de maneira eficaz às dinâmicas globais de doenças infecciosas. A necessidade de sistemas avançados de vigilância, especialmente em portões de entrada internacional como aeroportos, é reforçada pela emergência e rápida propagação global de doenças infecciosas.
Martins et al (2020) analisaram o microbioma presente na atmosfera de três terminais de ônibus em Fortaleza, destacando-se uma diversidade significativa de bactérias e fungos, com as análises revelando que as contagens de bactérias eram superiores às dos fungos em todos os terminais, com predominância de cocos Gram positivos e bastonetes Gram positivos. Os fungos identificados incluíram gêneros como Aspergillus e Fusarium, mostrando que a presença de um microbioma aéreo variado pode influenciar a saúde respiratória das pessoas que frequentam esses locais.
Ferramentas genômicas, exemplificadas pela introdução do BLAST por Altschul et al. (1990), revolucionaram a capacidade de detectar patógenos em amostras ambientais, estabelecendo um marco na identificação rápida de organismos potencialmente perigosos.
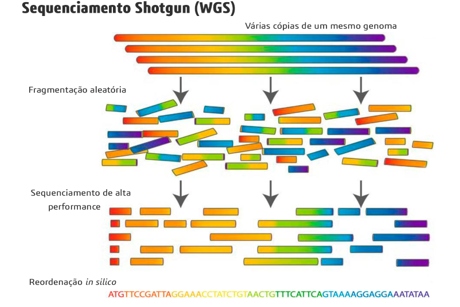
Figura 2 – Exemplo de tipos de cobertura de sequenciamento genômico. Fonte: Commins et al (2009).
Weiss et al. (2019) investigaram o microbioma de cabines de avião, revelando a complexidade das comunidades microbianas presentes e a predominância de microrganismos comensais humanos. Esses achados sublinham a importância de métodos de limpeza e desinfecção baseados em evidências para mitigar o risco de transmissão de doenças em locais de alto tráfego.
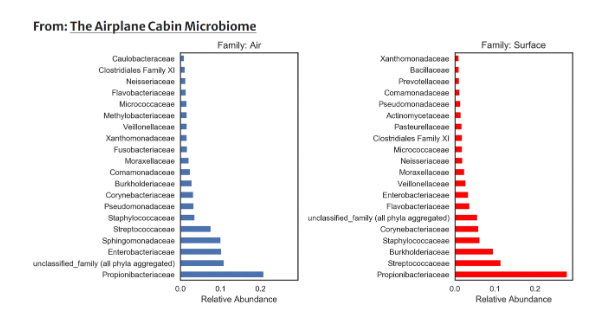
Figura 3 – Exemplo de microbioma da cabine de uma aeronave. Fonte:Weiss et al. (2019).
Cave et al. (2021) investigaram a prevalência de bactérias em ambientes públicos, utilizando técnicas modernas de vigilância genômica para mapear a distribuição e resistência desses microrganismos em áreas de alta circulação de pessoas. O estudo detalha a identificação de várias espécies de bactérias patogênicas, incluindo Staphylococcus, Escherichia coli, e de Klebsiella. O estudo analisou também a variabilidade na prevalência desses patógenos entre diferentes ambientes, mostrando como o monitoramento contínuo e detalhado pode ajudar na prevenção de surtos de doenças.
Em se tratando da política de saúde pública na prevenção de doenças e promoção da saúde, Quah (2016) explorou o papel da legislação e da política de saúde pública na prevenção de doenças e promoção da saúde, além da importância da cooperação supranacional e internacional para lidar com questões de saúde pública que ultrapassam fronteiras nacionais.
Katoh et al. (2002) e Li e Durbin (2009) realçaram a importância das ferramentas bioinformáticas para o alinhamento e análise de sequências genéticas. Tais tecnologias são indispensáveis para o processamento de dados de sequenciamento em alta profundidade, permitindo uma compreensão detalhada da diversidade genética dos patógenos detectados em ambientes como aeroportos. Estas ferramentas bioinformáticas facilitam identificações precisas e rápidas, cruciais para a implementação de medidas de controle e prevenção eficazes.
Assim, a metodologia adotada, que inclui coleta de amostras ambientais e análises genômicas, propõe uma abordagem inovadora para a vigilância epidemiológica em ambientes de alto risco. Este estudo não apenas mapeia a presença de patógenos em um dos maiores hubs aéreos do país, mas também avalia a eficácia das atuais estratégias de prevenção e controle de doenças infecciosas nesses ambientes (Katoh et al., 2002; Li & Durbin, 2009).
Espera-se que os resultados gerem dados para o aprimoramento das políticas de saúde pública, promovendo a prevenção de surtos e a segurança de viajantes e funcionários.
A combinação de avanços tecnológicos em sequenciamento genético com estratégias proativas de vigilância epidemiológica marca um avanço significativo na saúde pública. Essa abordagem não apenas possibilita a detecção e quantificação de patógenos conhecidos, mas também abre caminho para a identificação de novos agentes infecciosos, oferecendo uma base sólida para a implementação de políticas de prevenção de doenças e promoção da saúde em locais de alto risco, como terminais de aeroportos.
A aplicação dessas metodologias e tecnologias no contexto do Aeroporto Internacional de São Paulo/Guarulhos não só enriquece o corpo de conhecimento científico na área de monitoramento genômico ambiental, mas também estabelece um modelo replicável para futuras investigações e práticas de vigilância em saúde pública. Essas iniciativas são fundamentais para aprimorar a resposta global a ameaças emergentes de doenças infecciosas, visando não apenas melhorar a segurança dos viajantes, mas também proteger a saúde da população em geral.
2. metodologia
A coleta de amostras para monitoramento genômico ambiental em terminais de aeroportos é uma etapa crítica que demanda rigor e precisão. O objetivo é capturar uma representação fiel da diversidade microbiana presente, o que requer protocolos detalhados que minimizem a contaminação e garantam a integridade do material genético. Antes da coleta, definiu-se os locais de amostragem, que incluíram áreas de alto tráfego e pontos de contato frequente, como totens de check-in, áreas de espera, carrinhos de bagagem e corrimãos. A seleção desses pontos foi baseada em estudos preliminares e avaliação in loco antes do seu início.
O kit de coleta incluiu swabs estéreis, solução de preservação de RNA/DNA (como o DNA/RNA Shield), luvas descartáveis, máscaras e recipientes estéreis para transporte das amostras. A escolha da solução de preservação foi crucial para manter a estabilidade das amostras durante o transporte e armazenamento.

Figura 4 – Kit de coleta utilizado na pesquisa. Fonte: Autor.
Cada local de amostragem foi abordado com um protocolo consistente: usando luvas e máscaras para evitar contaminação, um swab estéril é umedecido na solução de preservação e então passado sobre a superfície escolhida com movimentos uniformes para coletar material biológico. Após a coleta, o swab foi colocado em um tubo contendo mais solução de preservação e devidamente etiquetado.

Figura 5 – Exemplo de coleta ambiental realizada em carrinho de passageiros no terminal 2 de passageiros. Fonte: Autor.
Para cada amostra coletada, foi fundamental registrar detalhadamente o local, a data e a hora da coleta, as condições ambientais (como temperatura e umidade, se possível) e qualquer observação relevante sobre o local de amostragem. Essa documentação foi crucial para análises subsequentes e interpretação dos resultados, e toda registrada em planilha específica.
Os locais de coleta consistiram em 8 pontos específicos, sendo 4 no terminal 2 (totens de check-in das companhias aéreas LATAM e GOL, sola do sapato e carrinhos de bagagem) e 4 no terminal 3 de passageiros (sola do sapato e 3 pontos de carrinhos de bagagem), sendo realizadas repetidamente nos dias 15/10/2021, 21/10/2021, 28/10/2021, 05/11/2021, 11/11/2021, 19/11/2021, 26/11/2021 e 03/12/2021.
As colunas da Tabela 1 contêm o ID Tube (código identificador do tubo de coleta), data, cidade, hora e minuto da coleta (horário local de Brasília), localização, tipo da superfície, o objeto amostrado, a área aproximada da superfície de coleta, a pessoa responsável pela coleta, a temperatura, umidade e coordenadas geográficas.
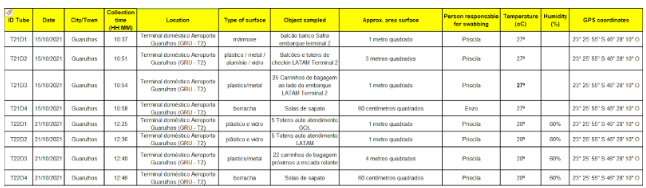
Tabela 1 – Coleta de amostras de superfície no Terminal 2 de passageiros do aeroporto de Guarulhos – 15/10/2021. Fonte: Autor.
As amostras foram transportadas ao laboratório central de sequenciamento do projeto (localizado na facility de sequenciamento genômico do A.C. Camargo Câncer Center, em São Paulo) o mais rápido possível, via correio (SEDEX), levando-se em torno de 1 dia útil entre a origem e o destino, mantendo-se em temperatura ambiente. No laboratório, as amostras foram armazenadas em freezers até o momento do processamento.
No laboratório, o material coletado foi submetido a processos de extração de DNA/RNA, seguindo protocolos rigorosos para evitar contaminação cruzada e garantir a extração eficiente do material genético. A qualidade e a quantidade do DNA/RNA extraído foram também avaliadas antes da análise subsequente.
Após a extração, o material genético foi submetido a sequenciamento de nova geração (NGS) e outras análises genômicas. Estas incluíram a quantificação de microrganismos específicos, análise de diversidade microbiana e identificação de patógenos potenciais.
A análise dos dados gerados pelo sequenciamento gerou a necessidade de aprendizagem em linguagem de programação Python, conforme Figura 8. Os resultados foram comparados com bancos de dados genômicos para identificar os microrganismos presentes nas amostras. Esta etapa foi crucial para entender a composição microbiana dos terminais de aeroportos e para identificar possíveis riscos à saúde pública.
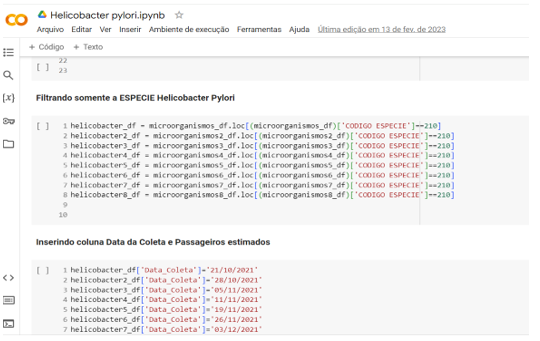
Figura 6 – Exemplo de processamento e análise de dados do microorganismo Helicobacter Pylori em linguagem de programação Python. Fonte: Autor.
Os resultados do monitoramento genômico puderam nortear medidas de prevenção e controle de infecções em aeroportos, servindo de base para a possível otimização de procedimentos de limpeza e desinfecção. Além disso, podem contribuir para políticas públicas de saúde, melhorando a resposta a surtos de doenças infecciosas.
3. resultados e discussão
Para uma comparação mais direta entre os terminais 2 e 3, consolidou-se os dados dos dois terminais e se criou uma visualização que destaque as diferenças ou semelhanças na presença relativa desses micro-organismos ao longo do período de coleta. Isso ajudou a entender se existem padrões específicos de prevalência de micro-organismos associados a cada terminal, o que pode ser indicativo de variações ambientais ou de fluxo de pessoas e atividades entre os terminais.
A figura 7 comparou a presença média relativa de Neisseria meningitidis, Streptococcus pneumoniae, Haemophilus influenzae e Helicobacter pylori entre os Terminais 2 e 3. Essa visualização permitiu identificar diferenças na prevalência média desses micro-organismos entre os terminais, oferecendo insights potenciais sobre as condições ambientais, o fluxo de pessoas e outras variáveis que podem influenciar a composição microbiana de cada local.
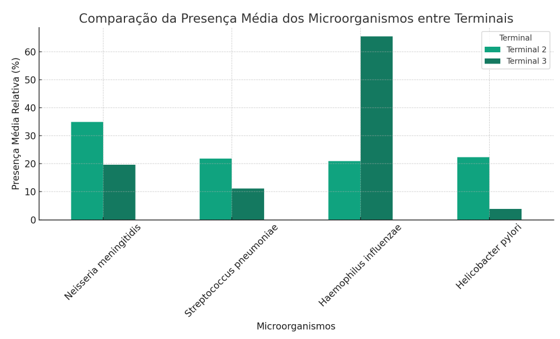
Figura 7 – Comparação da presença média dos microrganismos entre terminais. Fonte: Autor.
Quanto a correlação entre os micro-organismos foco deste trabalho, se calculou e visualizou a correlação na presença dos micro-organismos entre os terminais, para identificar se as variações em um terminal podem estar relacionadas às observações no outro, de acordo com a Figura 8.
Figura 8 – Comparação da diversidade microbiana entre os Terminais 2 e 3. Fonte: Autor.
Para a análise de diversidade, considerou-se a variedade e equilíbrio na presença dos quatro micro-organismos estudados como uma representação simplificada da diversidade microbiana de cada terminal. Calculou-se uma medida simplificada de diversidade, baseada na equitabilidade da presença relativa desses micro-organismos.
Por fim, explorou-se a correlação na presença dos micro-organismos entre os terminais, buscando identificar padrões que possam sugerir interações ou influências ambientais compartilhadas. Essa análise poderá revelar se mudanças na prevalência de um micro-organismo em um terminal estão relacionadas a mudanças similares no outro terminal.
A comparação da diversidade microbiana entre os Terminais 2 e 3, representada pela entropia, mostrou que o Terminal 2 possui uma maior diversidade (entropia = 1.96) em comparação com o Terminal 3 (entropia = 1.40). Esta medida sugere uma distribuição mais equilibrada na presença relativa dos microorganismos analisados no Terminal 2, indicando uma maior diversidade microbiana, ao menos entre os microorganismos especificados.
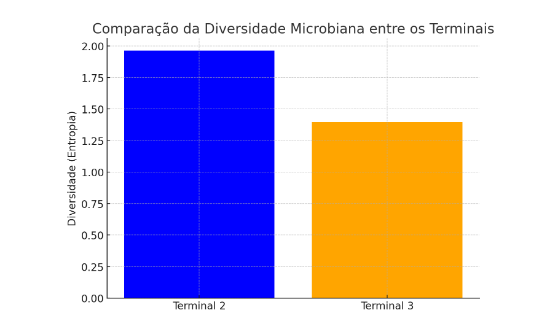
Figura 9 – Comparação da diversidade microbiana entre os Terminais 2 e 3, representada pela entropia. Fonte: Autor.
Ao se analisar a correlação entre microrganismos, explorou-se a correlação na presença dos micro-organismos entre os terminais. Esta análise visou identificar se as variações na prevalência de um micro-organismo em um terminal estão de alguma forma relacionadas às variações no outro terminal. Isso pode indicar influências ambientais semelhantes afetando ambos os terminais ou interações entre os micro-organismos que se manifestam de maneira consistente nos dois locais. Para isso, calculou-se a correlação entre as presenças médias relativas dos microorganismos nos dois terminais.
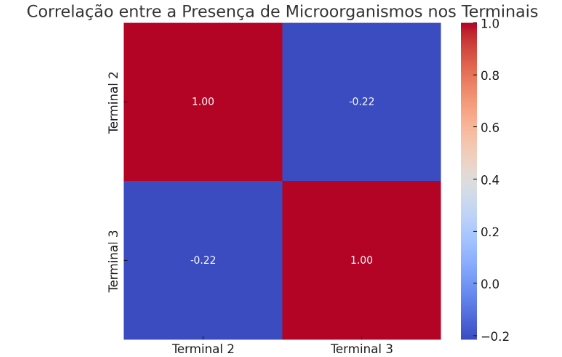
Figura 10 – Correlação entre a presença de microrganismos nos terminais. Fonte: Autor.
A matriz de correlação mostra a relação entre a presença média dos microrganismos nos Terminais 2 e 3. O valor de correlação entre os terminais é de aproximadamente -0.22, indicando uma correlação negativa muito fraca. Isso sugere que, na média, não existe uma relação linear significativa na variação da presença dos micro-organismos especificados entre os dois terminais.
Em resumo, estas análises revelam diferenças notáveis na presença e diversidade dos microrganismos selecionados entre os Terminais 2 e 3. A diversidade microbiana é maior no Terminal 2, enquanto a correlação na presença de micro-organismos entre os terminais é fraca, indicando que os fatores que influenciam a composição microbiana podem variar consideravelmente entre os dois locais.
Na Figura 11 pode-se verificar a variação temporal da presença relativa de cada microrganismo, separada por terminal. Isso nos permite identificar padrões temporais e comparar como esses padrões diferem entre os terminais.
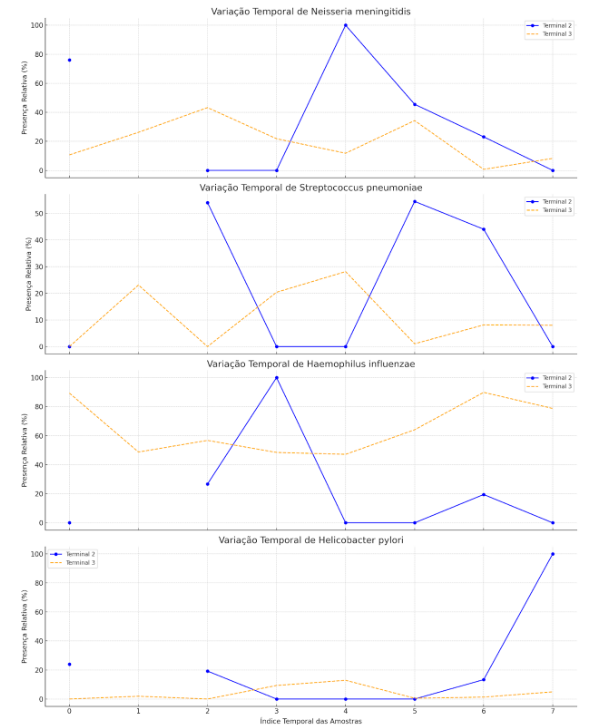
Figura 11 – Índice temporal das amostras nos terminais 2 e 3. Fonte: Autor.
As visualizações mostram a variação temporal da presença relativa de Neisseria meningitidis, Streptococcus pneumoniae, Haemophilus influenzae, e Helicobacter pylori nos Terminais 2 e 3. Cada gráfico representa um microorganismo específico, permitindo observar como sua prevalência varia ao longo do tempo em ambos os terminais.
Algumas observações importantes podem ser feitas a partir desses gráficos:
- Para alguns microrganismos, observou-se variação significativa na presença relativa ao longo do tempo, indicando possíveis flutuações ambientais ou de outras condições que afetam sua prevalência.
- A comparação entre os terminais revela que, embora alguns padrões temporais possam ser similares, existem diferenças notáveis na presença relativa dos microrganismos, sugerindo variações nas condições ou nas populações microbianas entre os terminais.
- O comportamento temporal de cada microrganismo oferece insights sobre sua dinâmica no ambiente estudado, podendo refletir respostas a fatores ambientais, interações microbianas ou influências humanas.
Estas análises temporais fornecem uma base para entender melhor a ecologia microbiana dos Terminais 2 e 3, assim como para investigar fatores que influenciam a composição e a dinâmica das comunidades microbianas nesses locais.
4. CONCLUSÕES
Os dados aqui apresentados destacam a importância e o impacto da implementação de um sistema avançado de monitoramento genômico ambiental no Aeroporto Internacional de São Paulo/Guarulhos. O estudo demonstrou a eficácia dessa abordagem inovadora na detecção e quantificação de organismos patogênicos específicos, contribuindo significativamente para a saúde pública ao oferecer insights valiosos para o desenvolvimento de estratégias de prevenção e controle de doenças infecciosas em espaços públicos de alto tráfego.
Os resultados obtidos sublinham a relevância da adoção de tecnologias de sequenciamento de última geração e protocolos padronizados no monitoramento ambiental, permitindo uma vigilância epidemiológica mais precisa e efetiva. A identificação das rotas de transmissão potenciais e a avaliação da eficácia das práticas de limpeza e desinfecção atuais são fundamentais para a elaboração de políticas públicas mais eficientes e direcionadas ao combate e prevenção de surtos de doenças infecciosas.
Além disso, este estudo reforçou a necessidade de uma abordagem interdisciplinar na biomedicina e saúde pública, integrando conhecimentos em genética, microbiologia e epidemiologia. A colaboração entre diferentes áreas do conhecimento é essencial para enfrentar os desafios impostos por patógenos emergentes e reemergentes, tornando o monitoramento genômico ambiental uma ferramenta indispensável no arsenal de vigilância em saúde pública.
Por fim, a implementação deste sistema de monitoramento no Aeroporto Internacional de São Paulo/Guarulhos estabeleceu um modelo replicável para futuras investigações e práticas de vigilância em saúde pública. As informações geradas pelo estudo fornecem uma base sólida para o aprimoramento contínuo das medidas de prevenção e controle de infecções, melhorando a segurança de viajantes e funcionários e protegendo a saúde da população em geral. Este trabalho não apenas enriqueceu o corpo de conhecimento científico na área de monitoramento genômico ambiental, mas também destaca a importância de tais iniciativas para responder eficazmente às dinâmicas globais de doenças infecciosas.
5. REFERÊNCIAS
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. (1990). Basic local alignment search tool. Journal of Molecular Biology, 215(3), 403-410.
CAVE, Rory; COLE, Jennifer; MKRTCHYAN, Hermine V. Surveillance and prevalence of antimicrobial resistant bacteria from public settings within urban built environments: Challenges and opportunities for hygiene and infection control. Environment international, v. 157, p. 106836, 2021.
Commins, J., Toft, C., Fares, M. A. – “Computational Biology Methods and Their Application to the Comparative Genomics of Endocellular Symbiotic Bacteria of Insects.” Biol. Procedures Online (2009).
Danchin A, Timmis K. SARS-CoV-2 variants: Relevance for symptom granularity, epidemiology, immunity (herd, vaccines), virus origin and containment? Environ Microbiol. 2020 Jun;22(6):2001-2006.
Katoh K, Misawa K, Kuma K, Miyata T. (2002). MAFFT: A novel method for rapid multiple sequence alignment based on fast Fourier transform. Nucleic Acids Research, 30(14), 3059-3066.
Li H, Durbin R. (2009). Fast and accurate short read alignment with Burrows–Wheeler transform. Bioinformatics, 25(14), 1754-60.
MARTINS, Ana Paula Alves. Microbioma urbano: microrganismos aerolizados em terminais de integração de ônibus da cidade de Fortaleza-CE. 2020.
QUAH, Stella R. International encyclopedia of public health. Academic press, 2016.
Weisblum Y, Schmidt F, Zhang F, DaSilva J, Poston D, Lorenzi JC, Muecksch F, Rutkowska M, Hoffmann HH, Michailidis E, Gaebler C, Agudelo M, Cho A, Wang Z, Gazumyan A, Cipolla M, Luchsinger L, Hillyer CD, Caskey M, Robbiani DF, Rice CM, Nussenzweig MC, Hatziioannou T, Bieniasz PD. Escape from neutralizing antibodies by SARS-CoV-2 spike protein variants. Elife. 2020 Oct 28;9:e61312.
Weiss H, et al. (2019). The Airplane Cabin Microbiome. Microbial Ecology, 77, pp. 87–95.
Universidade Anhembi Morumbi, Bacharelado em Biomedicina
