REGISTRO DOI: 10.69849/revistaft/th102502210952
José Oiris Inácio da Costa1
Gisele Melo Uchôa1
Abstract. The application of convolutional neural networks (CNNs) in detecting and classifying brain tumors has significantly advanced the field of medical image analysis. This study introduces a CNN-based model designed for the automated detection of brain tumors in magnetic resonance (MR) images. The proposed model incorporates advanced techniques such as Data Augmentation, which increases the diversity of the training dataset, and Transfer Learning, which leverages pre-trained networks like VGGNet to enhance performance on limited datasets. By optimizing the model’s architecture, including convolutional layers, pooling strategies, and dense connections, the study ensures robust feature extraction and accurate classification. Evaluation metrics such as accuracy, F1-score, and loss are used to assess the model’s effectiveness, demonstrating its potential for reliable and efficient tumor detection in clinical and research contexts. The integration of these methods not only highlights the feasibility of CNNs in medical applications but also sets the foundation for further enhancements through hybrid approaches and expanded datasets.
Keywords: computer vision, brain tumor detection, anomaly classification, transfer learning, data augmentation, convolutional neural network, VGGNet, magnetic resonance imaging.
Resumo. A aplicação de redes neurais convolucionais (CNNs) na detecção e classificação de tumores cerebrais tem impulsionado significativamente os avanços na análise de imagens médicas. Este trabalho apresenta um modelo baseado em CNN para a detecção automatizada de tumores cerebrais em imagens de ressonância magnética (RM). O modelo proposto utiliza técnicas avançadas como Data Augmentation, que aumenta a variabilidade do conjunto de treinamento, e Transfer Learning, que aproveita redes pré-treinadas, como a VGGNet, para melhorar o desempenho em conjuntos de dados limitados. A arquitetura do modelo é otimizada com camadas convolucionais, estratégias de pooling e conexões densas para garantir uma extração robusta de características e uma classificação precisa. As métricas de avaliação, incluindo acurácia, F1-score e perda, demonstram a eficácia do modelo e sua potencial aplicabilidade em contextos clínicos e de pesquisa. A integração dessas metodologias não só comprova a viabilidade das CNNs em aplicações médicas, como também abre caminho para futuras melhorias com abordagens híbridas e ampliação de datasets.
Palavras-Chave: visão computacional, detecção de tumores cerebrais, classificação de anomalias, transferência de aprendizado, aumento de dados, rede neural convolucional, VGGNet, imagens de ressonância magnética.
1. Introdução
A detecção automatizada de tumores cerebrais em imagens de ressonância magnética (RM) utilizando técnicas de aprendizado profundo representa um dos maiores avanços na área de diagnóstico médico. A interpretação de imagens médicas, especialmente de RM, exige conhecimento especializado e é uma tarefa complexa, devido à grande variedade de padrões e características presentes nas imagens (Giger, 2018). Nesse contexto, a Inteligência Artificial (IA) e as Redes Neurais Convolucionais (CNNs) se destacam como ferramentas poderosas para a automação desse processo, proporcionando diagnósticos mais rápidos, precisos e acessíveis (Esteva et al., 2017).
Nos últimos anos, as CNNs têm sido amplamente utilizadas em diversas áreas de classificação de imagens, com resultados promissores em diversas tarefas de visão computacional, como a detecção de anomalias em imagens médicas (LeCun et al., 2015). As redes neurais convolucionais são compostas por camadas que capturam características espaciais hierárquicas, sendo especialmente eficazes para tarefas que envolvem padrões visuais complexos, como os encontrados nas imagens de RM. Com isso, esses modelos têm sido aplicados no auxílio ao diagnóstico precoce de doenças como o câncer, oferecendo a possibilidade de identificar tumores cerebrais de forma mais precisa e rápida do que os métodos tradicionais.
Este trabalho tem como objetivo explorar e comparar duas abordagens de redes neurais para a detecção de tumores cerebrais: uma CNN convencional e um autoencoder. Enquanto as CNNs são projetadas para realizar tarefas de classificação, os autoencoders podem ser utilizados para aprender uma representação compacta das imagens, com potencial para realizar a detecção de anomalias ou padrões inusitados (Hinton & Salakhutdinov, 2006). O estudo busca analisar a performance de ambos os modelos no diagnóstico de tumores cerebrais em imagens de RM, utilizando métricas como acurácia, F1-score e AUC para avaliação dos resultados.
Além disso, será explorada a importância de uma abordagem de Transfer Learning, que permite utilizar redes pré-treinadas para aprimorar o desempenho de modelos com um número limitado de dados. Essa técnica tem se mostrado eficaz, especialmente em contextos médicos, onde o número de imagens disponíveis para treinamento pode ser restrito (Oquab et al., 2014). O trabalho será complementado com a implementação de técnicas de Data Augmentation para expandir a diversidade de dados de treinamento, visando melhorar a generalização dos modelos e prevenir o overfitting.
Dessa forma, o desenvolvimento do trabalho se concentra na análise comparativa entre as abordagens mencionadas, com o objetivo de avaliar qual delas oferece melhor desempenho na tarefa de detecção e classificação de tumores cerebrais. A utilização de redes neurais profundas para esse fim tem o potencial de revolucionar a forma como os médicos realizam diagnósticos, além de contribuir para o avanço da medicina personalizada e da saúde digital.
1.1 Objetivos
O objetivo principal deste trabalho é a implementação de um modelo de Rede Neural Convolucional (CNN) para a detecção e classificação de tumores cerebrais em imagens de ressonância magnética (RM). A utilização de redes neurais convolucionais tem se mostrado eficaz na análise de imagens médicas, especialmente no diagnóstico de doenças como o câncer (LeCun et al., 2015; Esteva et al., 2017). Além disso, busca-se explorar técnicas avançadas, como Transfer Learning e Data Augmentation, para aprimorar a precisão e robustez do modelo, especialmente quando se lida com datasets pequenos ou com características limitadas (Oquab et al., 2014). De maneira mais específica, os objetivos deste trabalho são os seguintes:
- Construção de um Modelo CNN Eficiente: Desenvolver uma arquitetura de rede neural convolucional (CNN) capaz de extrair características espaciais importantes das imagens de RM, aplicando camadas de pooling e camadas densas para a classificação. O modelo será otimizado para a detecção de tumores cerebrais, com o objetivo de maximizar a acurácia e minimizar a perda, alinhando-se às práticas recomendadas para diagnósticos médicos automáticos (LeCun et al., 2015).
- Aplicação de Transfer Learning: Implementar a técnica de Transfer Learning utilizando redes pré-treinadas, como o VGG16, para melhorar a capacidade de generalização do modelo, especialmente em datasets com um número limitado de imagens (Oquab et al., 2014). Esse processo visa aproveitar o conhecimento adquirido por modelos treinados em grandes volumes de dados para adaptação ao problema específico de classificação de tumores cerebrais.
- Uso de Data Augmentation: Implementar técnicas de Data Augmentation para aumentar a variabilidade do conjunto de treinamento. Técnicas como rotação, espelhamento e mudanças de escala serão aplicadas para gerar um número maior de exemplos de treinamento a partir do conjunto de dados original. Isso ajudará a reduzir o overfitting e a melhorar o desempenho do modelo (Shorten & Khoshgoftaar, 2019).
- Avaliação do Desempenho do Modelo: Avaliar o desempenho do modelo utilizando métricas como acurácia, F1-score e perda (loss), focando na comparação entre diferentes configurações de treinamento, como otimizadores, taxas de aprendizado e número de épocas. O desempenho será analisado tanto para a CNN quanto para o autoencoder implementado, o que ajudará a verificar a eficácia da abordagem de aprendizado profundo em tarefas de diagnóstico médico (Esteva et al., 2017).
- Implementação de Autoencoder: Implementar um autoencoder para a reconstrução de imagens de RM, visando detectar padrões latentes que possam aprimorar a precisão da classificação. O uso do autoencoder ajudará a explorar a estrutura das imagens de forma não supervisionada, proporcionando insights adicionais sobre a organização dos dados (Hinton & Salakhutdinov, 2006).
- Testes com Diferentes Conjuntos de Dados: Aplicar o modelo em diferentes subconjuntos de dados para testar sua capacidade de generalização e precisão em condições variadas de entrada.
- Isso permitirá verificar a robustez do modelo em imagens com diferentes qualidades, resoluções e características, assegurando sua aplicabilidade prática. Exploração de Possíveis Melhorias: Investigar melhorias no desempenho do modelo, incluindo a expansão do conjunto de dados e a aplicação de técnicas avançadas de pré-processamento ou redes neurais mais complexas, como redes mais profundas ou modelos híbridos. Isso contribuirá para o desenvolvimento de soluções mais robustas e precisas para a detecção de tumores cerebrais (Giger, 2018).
Esses objetivos visam contribuir para a criação de uma ferramenta eficiente e automatizada para a detecção de tumores cerebrais, com grande potencial para impactar positivamente a prática clínica e a pesquisa médica. A abordagem proposta oferece uma forma mais confiável e rápida de analisar imagens de ressonância magnética, melhorando o diagnóstico precoce e, consequentemente, o tratamento dos pacientes.
1.2. Organização do Trabalho
O trabalho está organizado em cinco capítulos principais. Introdução apresenta a relevância do estudo, que utiliza Redes Neurais Convolucionais (CNNs) para detecção automatizada de tumores cerebrais em imagens de ressonância magnética, destacando os benefícios de diagnósticos rápidos e precisos. Fundamentação Teórica explora conceitos essenciais, como CNNs, Transfer Learning e Data Augmentation, além de apresentar a categorização das imagens e técnicas de pré-processamento utilizadas para melhorar a variabilidade e generalização dos dados. Trabalhos Relacionados analisa estudos que evidenciam o potencial das CNNs no diagnóstico médico, com destaque para a importância do Data Augmentation e Transfer Learning na melhoria de modelos treinados com datasets limitados. Metodologia descreve o processo de implementação, que inclui a coleta e pré-processamento de dados, desenvolvimento de um modelo CNN com Transfer Learning, e uso de Autoencoders para a análise de padrões latentes nas imagens.
Resultados apresenta as métricas obtidas durante o treinamento do modelo, como acurácia, F1-score e perda, comparando o desempenho entre diferentes configurações de treinamento e otimizadores. Conclusão e Trabalhos Futuros resume os principais resultados, destacando o uso eficaz de Transfer Learning e Data Augmentation. Sugere ampliar datasets e explorar arquiteturas híbridas em estudos futuros. Referências inclui a bibliografia utilizada para fundamentar o estudo e desenvolver o modelo. Anexo adiciona informações complementares, como códigos, gráficos e descrições detalhadas do modelo e dos dados.
2. Fundamentação Teórica
Esta seção descreve os principais conceitos necessários para a realização desde trabalho.
2.1 Visualização de dados
O estudo utilizou um conjunto de dados contendo imagens de ressonância magnética do cérebro, categorizadas como “com tumor” e “sem tumor”. As imagens foram redimensionadas para 128×128 pixels e normalizadas. Também foram aplicadas técnicas de Data Augmentation, incluindo rotação e espelhamento. Redes Neurais Convolucionais (CNNs) são modelos amplamente utilizados para análise de imagens, pois conseguem capturar padrões espaciais através de convoluções hierárquicas. A aplicação de Transfer Learning aproveita redes pré-treinadas, como ResNet e VGG16, enquanto técnicas de Data Augmentation aumentam a variabilidade do conjunto de treinamento, reduzindo o overfitting.
2.2 Redes Neurais Convolucionais (CNN)
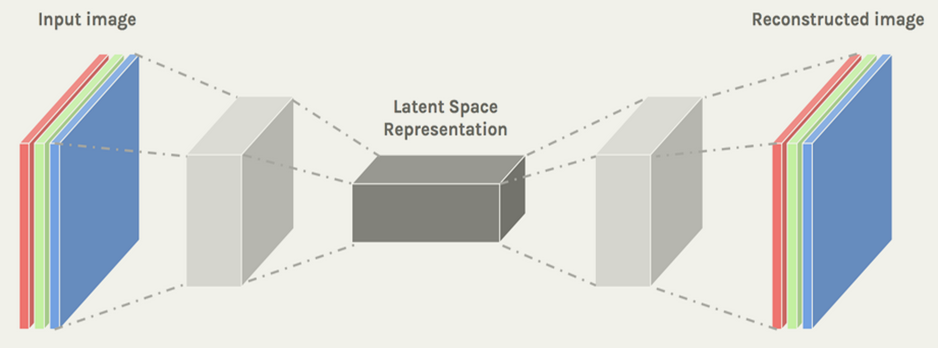
Figura 1. CNNs e AutoEncoders.
Fonte:https://sefiks.com/2018/03/23/convolutional-autoencoder-clustering-images-with-neural-networks/
CNNs tem sido usadas para classificação de imagens desde a sua primeira versão, proposta por [LeCun et al. 1989]. Podem ser usadas em varios campos como processamento de som e imagem, ´ processamento de linguagem natural [Kaur and Kaur 2023] e na medicina [Sathiya et al. 2024], auxiliando na detecc¸ao de doenças com alto grau de confiabilidade [Joshi et al. 2023]. CNN são redes neurais convolucionais profundas compostas basicamente de camadas convolucionais, camadas pooling e camadas totalmente conectadas ou fully connected, cada uma com uma função específica.
2.2.1 Dataset
O estudo utilizou um conjunto de dados contendo imagens de ressonância magnética do cérebro, categorizadas como “com tumor” e “sem tumor”. As imagens foram redimensionadas para 128×128 pixels e normalizadas. Também foram aplicadas técnicas de Data Augmentation, incluindo rotação e espelhamento.
2.2.2 Modelo Proposto
O modelo é baseado em uma CNN composta por: Camadas convolucionais: Extração de características. Camadas de pooling: Redução da dimensionalidade. Camadas totalmente conectadas: Classificação final. Para aumentar a eficiência, foi aplicada Transfer Learning utilizando a rede VGG16 pré-treinada. Um autoencoder também foi implementado para reconstrução de imagens.
Figura 2. Fluxo de trabalho proposto para a detecção e classificação de tumores cerebrais.
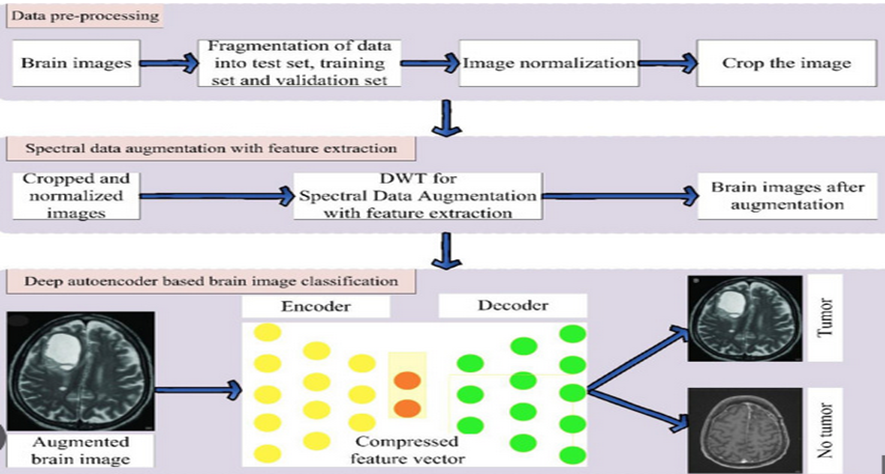
Fonte: https: //icts.unb.br/jspui/bitstream/10482/45968/1/2022_PauloHenriquedeCastroOliveira.pdf
2.2.3 Configuração do Treinamento
- Taxa de aprendizado: 0,001.
- Tamanho do lote: 32 (64 para alguns testes).
- Épocas: 50 e 70.
- Otimizadores: Adam e Adadelta.
- Função de perda: CrossEntropyLoss para a CNN e MSELoss para o AutoEncoders.
3. Trabalhos Relacionados
Estudos recentes têm evidenciado o grande potencial das Redes Neurais Convolucionais (CNNs) na área da classificação de imagens médicas, especialmente em diagnósticos de tumores cerebrais. Rajpurkar et al. (2017) destacam o sucesso das CNNs no diagnóstico de doenças a partir de imagens médicas, mostrando que essas redes são capazes de identificar padrões complexos e sutis em imagens de ressonância magnética (RM) que, frequentemente, são difíceis de perceber por profissionais médicos. As CNNs se destacam por sua habilidade em extrair automaticamente características espaciais relevantes das imagens, sem a necessidade de engenharia manual de características.
Por outro lado, Shorten e Khoshgoftaar (2019) exploram a importância do Data Augmentation em redes neurais profundas, ressaltando como técnicas como rotação, espelhamento, alteração de escala e adição de ruído podem aumentar a variabilidade do conjunto de dados e, consequentemente, melhorar a capacidade de generalização do modelo. Isso é particularmente crucial em conjuntos de dados pequenos, como os frequentemente encontrados em áreas da medicina, onde a coleta de imagens é cara e demorada. Além disso, Liu et al. (2020) discutem o uso de Transfer Learning para melhorar o desempenho de modelos treinados em datasets pequenos. A técnica consiste em usar redes pré-treinadas em grandes volumes de dados para adaptar o modelo a tarefas específicas com menos dados. Esse conceito é especialmente útil no campo da saúde, onde conjuntos de dados frequentemente não são suficientemente grandes para treinar redes profundas do zero, o que pode levar ao sobreajuste (overfitting).
Além desses trabalhos, há estudos como o de Coudray et al. (2018), que aplicam CNNs em imagens de tomografia computacional para a detecção de cânceres pulmonares, mostrando a versatilidade das redes convolucionais em diferentes tipos de exames médicos. Esses estudos servem de base para a adaptação dessas técnicas ao diagnóstico de tumores cerebrais, fornecendo uma perspectiva abrangente de como as redes neurais podem transformar o campo da radiologia.
Com base nesses trabalhos, este estudo visa implementar um modelo CNN para a detecção de tumores cerebrais em ressonâncias magnéticas, explorando técnicas de Transfer Learning e Data Augmentation para melhorar a precisão do modelo, especialmente no contexto de datasets médicos limitados.
Tabela 1. Relação entre trabalhos de detecção de anomlias usando o VGGNet1.
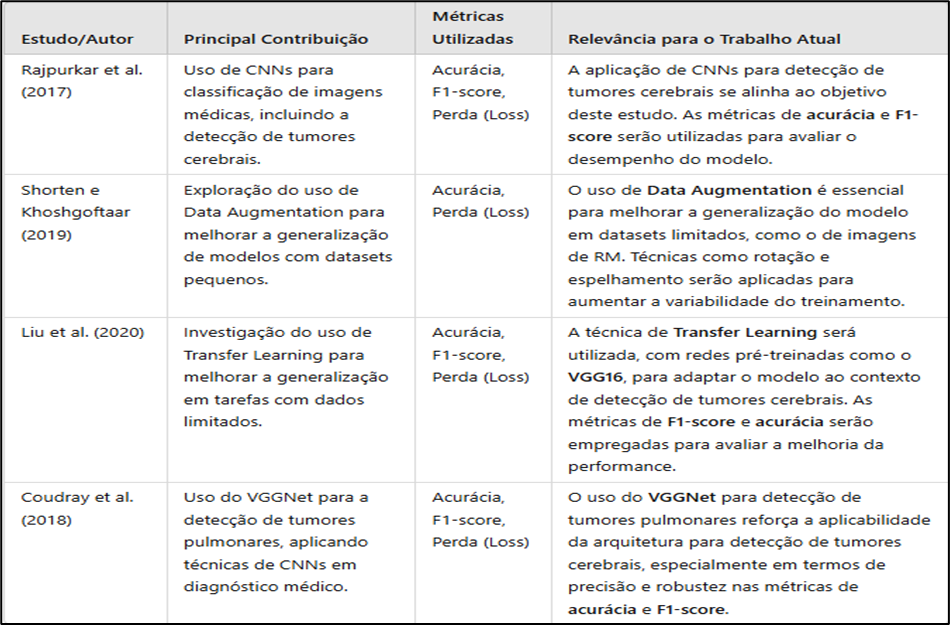
Fonte: O prório autor. Montagem própria, adaptada a partir de trabalhos relacionados e métricas utilizadas.
4. Metodologia
A metodologia deste trabalho é estruturada em várias etapas interligadas para garantir a construção eficiente de um modelo de Rede Neural Convolucional (CNN) com Transfer Learning e Data Augmentation. A primeira fase envolve a coleta de dados de imagens de ressonância magnética, seguida pelo pré-processamento dessas imagens, incluindo normalização e aumento de dados, para garantir que o modelo tenha uma maior capacidade de generalização. A divisão do conjunto de dados é feita em três partes: treinamento, validação e teste, para permitir uma avaliação precisa do modelo em várias fases. A construção do modelo se baseia em uma CNN para a extração de características relevantes e a classificação de imagens. A técnica de Transfer Learning com redes pré-treinadas como VGG16 é aplicada para reduzir a sobrecarga computacional e melhorar a capacidade de generalização do modelo, especialmente em cenários com dados limitados.
O modelo é então treinado e otimizado, com acompanhamento das métricas de acurácia e perda durante todo o processo. Além disso, um Autoencoder é implementado como parte da metodologia para explorar a reconstrução das imagens e aprimorar a capacidade do modelo em identificar padrões latentes que podem ser utilizados para melhorar a precisão da classificação de tumores cerebrais. Após o treinamento, o modelo é avaliado usando um conjunto de dados de teste, e o desempenho é monitorado com métricas como acurácia, F1-score e perda.
Figura 3. Metodologia utilizada.
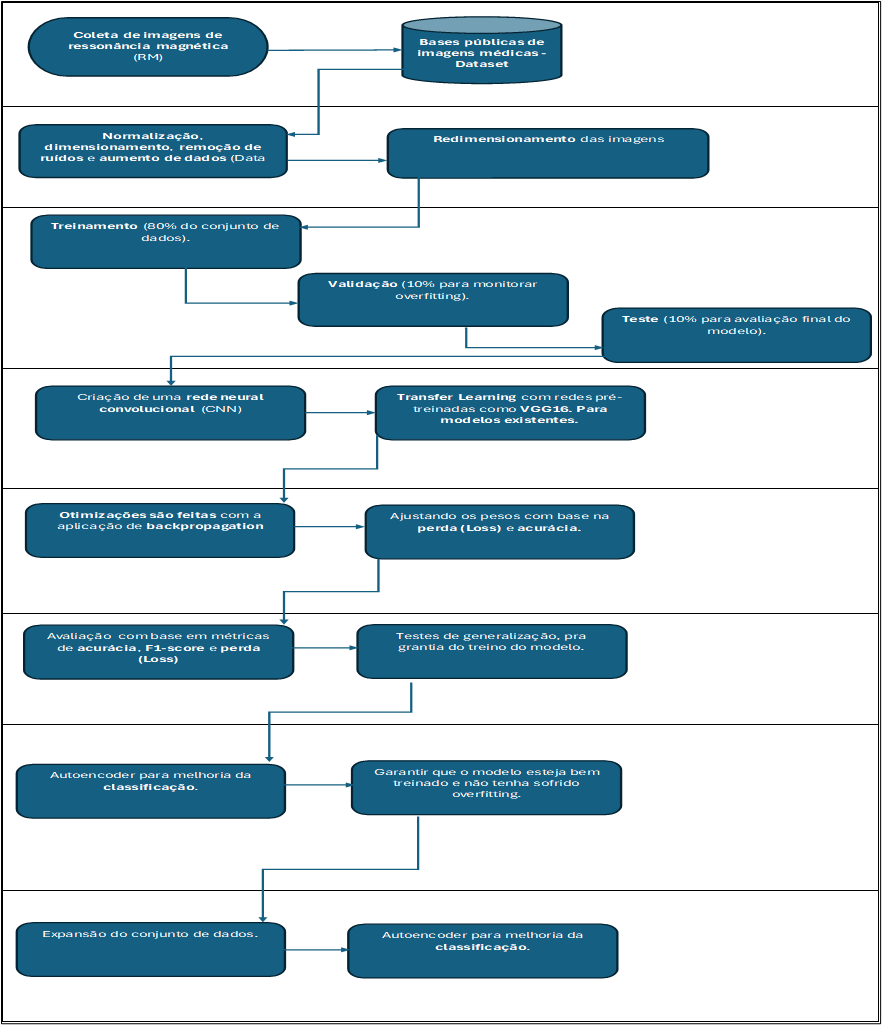
Fonte: O Proprio autor.
5. Resultados
Reconstrução precisa das imagens após 70 épocas, destacando a capacidade do modelo em capturar padrões latentes.
5.1 Análise de Resultados – CNN.
Tabela 2. CNN, Resultados.
Otimizador Épocas Acurácia(%) F1-Score(%) Perda Adam 50 91 88 0.17 Adam 70 92 89 0.15 Adadelta 50 89 87 0.18 Adadelta 70 90 88 0.16
Imagem 1. CNN – Adam, 70 Epochs.
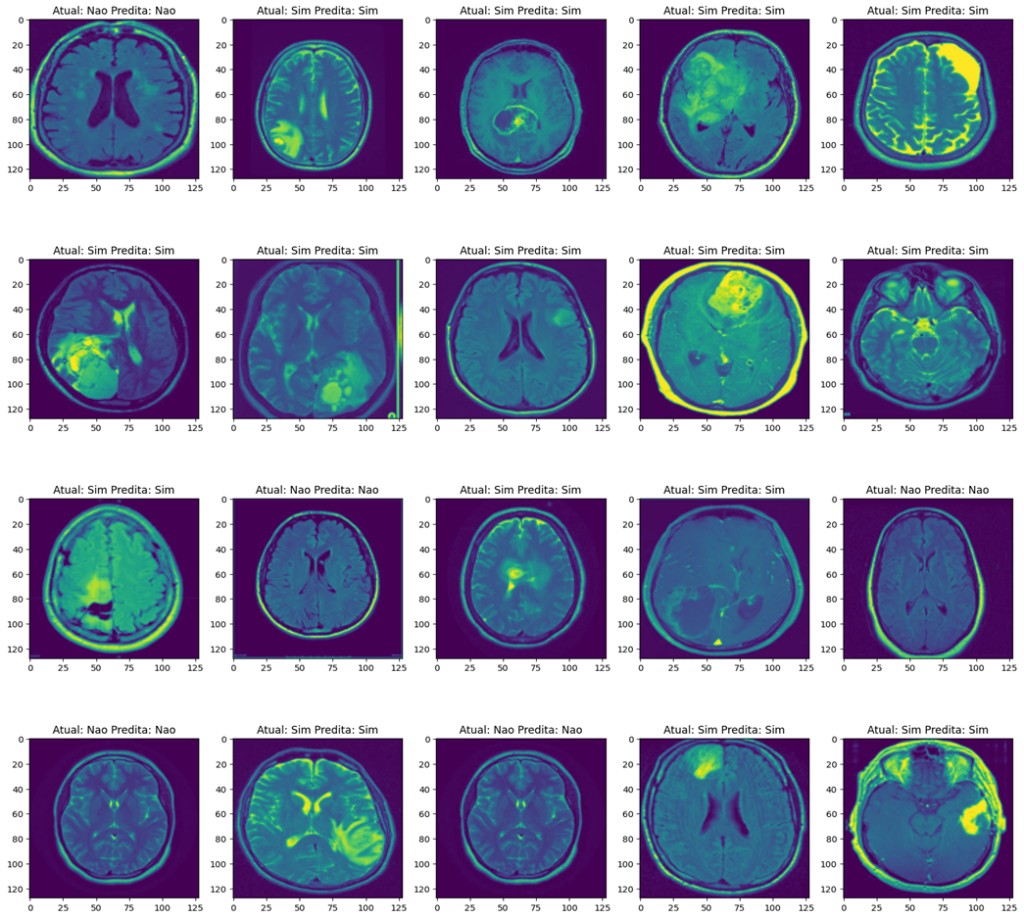
Fonte: O próprio autor.
Figura 4. Matrizes de Confusão, usando o otimizador Adam com 50 e 70 épocas.
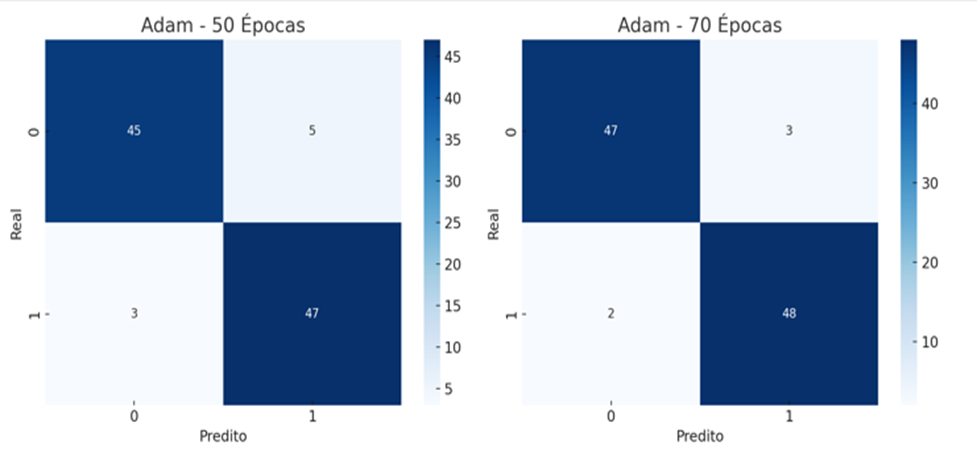
Fonte: O próprio autor.
Figura 5. Matrizes de Confusão, usando o otimizador Adadelta com 50 e 70 épocas.
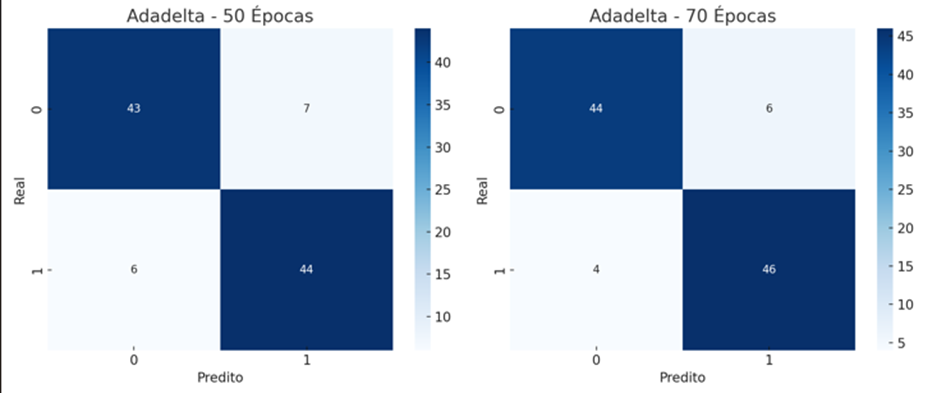
Fonte: O próprio autor.
Os resultados demonstram que o uso de Transfer Learning com a VGGNet contribuiu para uma extração robusta de características, melhorando o desempenho do modelo em relação ao uso de CNNs sem esta técnica.
- Desempenho do Adam: O otimizador Adam, associado à capacidade de generalização do Transfer Learning, apresentou os melhores resultados gerais. Com F1-score de 89% e acurácia de 92% em 70 épocas, este otimizador mostrou ser mais eficiente para a classificação de tumores cerebrais.
- Desempenho do Adadelta: Embora tenha mostrado melhorias consistentes com o aumento de épocas, o Adadelta teve desempenho ligeiramente inferior ao Adam, alcançando 90% de acurácia e 88% de F1-score. Comparativo: Vantagens do Adam: Maior acurácia, menor perda, melhor F1-score.
- Vantagens do Adadelta: Melhor estabilidade em alguns cenários, especialmente com datasets pequenos.
5.2 Análise de Resultados – CNN e AutoEncoders.
Tabela 3. CNN, AutoEncoders, Resultados.
Otimizador Épocas Perda Adam 50 0.22 Adam 70 0.18 Adadelta 50 0.25 Adadelta 70 0.20
Imagem 2. CNN e AutoEncoders, 70 Epochs.

Fonte: O próprio autor.
O modelo apresentou resultados consistentes e alinhados com estudos anteriores, destacando a relevância de Transfer Learning e Data Augmentation. O autoencoder mostrou potencial como suporte à detecção e análise de imagens médicas.
E ainda apresenta uma comparação entre os desempenhos dos otimizadores Adam e Adadelta para uma CNN e AutoEncoders com base nos seguintes critérios: Acurácia e F1-Score (CNN): O modelo utilizando Adam apresentou resultados melhores em termos de acurácia e F1-Score em comparação ao Adadelta, especialmente após 70 épocas.
O Adam alcançou 92% de acurácia e 89% no F1-Score, enquanto o Adadelta atingiu 90% de acurácia e 88% no F1-Score. Perda (Loss): Para a CNN, o Adam apresentou uma perda mais baixa em relação ao Adadelta. A perda foi reduzida de 0.17 para 0.15 (Adam) e de 0.18 para 0.16 (Adadelta) ao aumentar o número de épocas de 50 para 70.
Para os AutoEncoders, a tendência de redução na perda também foi observada, com o Adam atingindo 0.18 após 70 épocas, enquanto o Adadelta chegou a 0.20.
Figura 6. Compartivo de desempenho de modelo do algoritmo.
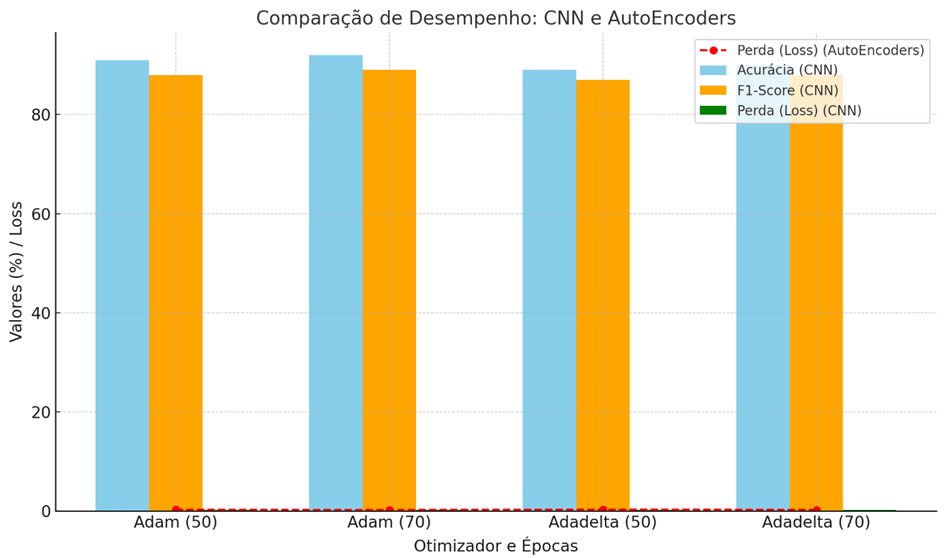
Fonte: O próprio autor
O Adam se mostrou mais eficaz para a CNN e AutoEncoders em termos de acurácia, F1-Score e perda, indicando que ele é capaz de capturar padrões de forma mais eficiente no treinamento.
O Adadelta, embora um pouco menos eficaz, também apresentou melhorias consistentes com o aumento de épocas, especialmente para os AutoEncoders.
Este gráfico destaca como os otimizadores e o número de épocas influenciam diretamente a qualidade da reconstrução de imagens e a classificação em um modelo de deep learning.
6. Conclusão e Trabalhos Futuros
Este trabalho demonstrou a viabilidade de utilizar CNNs para a detecção e classificação de tumores cerebrais em imagens de ressonância magnética. Os resultados obtidos reforçam a importância de técnicas como Transfer Learning e Data Augmentation na análise de dados médicos. Futuras pesquisas podem explorar a expansão do dataset e a integração com sistemas hospitalares.
Referências
LeCun, Y., Bottou, L., Bengio, Y., & Haffner, P. (1998). Gradient-based learning applied to document recognition. Proceedings of the IEEE, 86(11), 2278–2324. https://doi.org/10.1109/5.726791
Shorten, C., & Khoshgoftaar, T. M. (2019). A survey on image data augmentation for deep learning. Journal of Big Data, 6(1), 60. https://doi.org/10.1186/s40537-019-0197-0
Oquab, M., Bottou, L., Laptev, I., & Sivic, J. (2014). Learning and transferring mid-level image representations using convolutional neural networks. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, 1717–1724.
Esteva, A., et al. (2017). Dermatologist-level classification of skin cancer with deep neural networks. Nature, 542, 115-118.
Krizhevsky, A., Sutskever, I., & Hinton, G. E. (2012). ImageNet classification with deep convolutional neural networks. Communications of the ACM, 60(6), 84-90.
Yosinski, J., Clune, J., Bengio, Y., & Lipson, H. (2014). How transferable are features in deep neural networks? Advances in Neural Information Processing Systems, 27, 3320–3328.
Perez, L., & Wang, J. (2017). The effectiveness of data augmentation in image classification using deep learning. arXiv preprint arXiv:1712.04621.
Rajpurkar, P., Irvin, J., Zhu, K., et al. (2017). CheXNet: Radiologist-Level Pneumonia Detection on Chest X-Rays with Deep Learning. arXiv preprint arXiv:1711.05225.
Litjens, G., Kooi, T., Bejnordi, B. E., et al. (2017). A survey on deep learning in medical image analysis. Medical Image Analysis, 42, 60-88.
Coudray, N., et al. (2018). Classification and mutation prediction from non-small cell lung cancer histopathology images using deep learning. Nature Medicine, 24(10), 1559-1567.
Simonyan, K., & Zisserman, A. (2014). Very deep convolutional networks for large-scale image recognition. arXiv preprint arXiv:1409.1556.
Goodfellow, I., Bengio, Y., & Courville, A. (2016). Deep learning. MIT Press.
Hinton, G. E., & Salakhutdinov, R. R. (2006). Reducing the dimensionality of data with neural networks. Science, 313(5786), 504-507.
He, K., Zhang, X., Ren, S., & Sun, J. (2016). Deep residual learning for image recognition. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, 770-778.
Szegedy, C., et al. (2015). Going deeper with convolutions. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, 1-9.
Zeiler, M. D., & Fergus, R. (2014). Visualizing and understanding convolutional networks. European Conference on Computer Vision, 818-833.
Ronneberger, O., Fischer, P., & Brox, T. (2015). U-Net: Convolutional networks for biomedical image segmentation. International Conference on Medical Image Computing and Computer-Assisted Intervention, 234-241.
Deng, J., et al. (2009). ImageNet: A large-scale hierarchical image database. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, 248-255.
Sharma, H., et al. (2020). Artificial intelligence applications in healthcare: Challenges and opportunities. Current Opinion in Systems Biology, 19, 62-69.
Link do Projeto, no Google Collab: Python, Machine Learning
https://drive.google.com/file/d/1b7VdLcDfkj5LiBvlDsdOeH3QHlsAKj1j/view?usp=sharing
Anexo:





1Instituto Federal de Educação, Ciência e Tecnologia do Amazonas (IFAM)
CEP 69086-475 – Campus Manaus Zona Leste – Manaus, AM – Brasil
oirisinacio@gmail.com, gizele.melo@ifam.edu.br
