GASTROINTESTINAL TRACT CANCER AND EPIGENETIC SIGNATURE: A NEW SCENE IN DISEASE TREATMENT LINKED TO GENE EXPRESSION MODULATION
CÁNCER DEL TRACTO GASTROINTESTINAL Y FIRMA EPIGENÉTICA: UN NUEVO ESCENARIO EN EL TRATAMIENTO DE ENFERMEDADES VINCULADO A LA MODULACIÓN DE LA EXPRESIÓN GÉNICA
REGISTRO DOI: 10.5281/zenodo.7878923
Thiago Torres Terto da Silva1
Beatriz Modesto Silva Magalhães2
Bruna Modesto Silva Magalhães3
Jaim Simões de Oliveira4
RESUMO
INTRODUÇÃO: O câncer é uma doença complexa que envolve alterações de múltiplos processos genéticos e epigenéticos, contribuindo estes como fatores centrais para a evolução da doença neoplásica. abraçar a epigenética e conceituá-la como modificações potencialmente hereditárias na expressão genética que não envolvem, necessariamente, mudanças na sequência de DNA, há a inclusão, no escopo dessas alterações, de processos como a metilação de DNA, modificações de histona, remodeladores de cromatina e RNAs não codificantes. Essas alterações, em última análise, mediam o comportamento clínico dos pacientes com ou sem o diagnóstico oncológico e podem ainda ser utilizadas como biomarcadores para determinação do risco de desenvolvimento neoplásico, em que pese sua prévia detecção e consolidação de um prognóstico a seguir, à luz de uma previsão vivaz de resposta à terapia oncológica. OBJETIVO: Qual a relevância terapêutica da assinatura epigenética para os pacientes oncológicos, em que pese o contexto de detecção, acompanhamento e prognóstico voltados às neoplasias do trato gastrointestinal, ao envolver, em termos comparativos, os atuais sistemas de gerenciamento cancerígeno? METODOLOGIA: O fomento desta revisão integrativa, elaborada a partir de uma vertente quantitativa e descritiva, perpassou pela realização de buscas até março de 2022 nas plataformas de dados PubMed e Biblioteca Virtual em Saúde (BVS). Para tanto, foram utilizados os seguintes descritores: Gastrointestinal Neoplasms; Epigenomics; Therapeutics; Gene Expression. Estratégias de busca foram construídas a partir da combinação desses descritores com o operador booleano AND. Ao fim, 15 artigos foram selecionados para a efetiva discussão. RESULTADOS E DISCUSSÃO: Após completo balanço dos estudos, a maioria deles estabeleceu que a relevância da assinatura epigenética para os pacientes oncológicos é centrada na possibilidade do desenvolvimento de vias terapêuticas melhoradas, edificando a surgência de potenciais biomarcadores de neoplasias, o que aprofunda o limiar de acompanhamento clínico. CONCLUSÃO: O conjunto de trabalhos analisados demonstrou que alterações na metilação de alguns genes podem provocar importantes mudanças na expressão gênica, por meio de expansão do sinal, ativação ou repressão da transcrição a proposta de novas estratégias terapêuticas eficazes para o âmbito epigenético e neoplasias pode utilizar informações advindas de co ativadores transcricionais específicos. Por fim, novos estudos são necessários para aprofundar o entendimento entre delimitações prévias da relação do contexto epigenético com o cenário cancerígeno apresentado.
Palavras-chave: Neoplasias Gastrointestinais; Epigenética; Terapêutica; Expressão Gênica.
ABSTRACT
INTRODUCTION: Cancer is a complex disease involving alterations in multiple genetic and epigenetic processes, which contribute as central factors to the evolution of neoplastic disease. Embracing epigenetics and conceptualizing it as potentially heritable modifications in gene expression that do not necessarily involve changes in the DNA sequence, includes processes such as DNA methylation, histone modifications, chromatin remodelers, and non-coding RNAs. Ultimately, these alterations mediate the clinical behavior of patients with or without an oncologic diagnosis and can be used as biomarkers for determining the risk of neoplastic development, despite their prior detection and consolidation of a prognosis to follow, in light of a vivid prediction of response to oncologic therapy. OBJECTIVE: What is the therapeutic relevance of epigenetic signatures for oncology patients, in terms of detection, monitoring, and prognosis focused on gastrointestinal tract neoplasms, compared to current cancer management systems? METHODOLOGY: This integrative review, developed from a quantitative and descriptive approach, included searches up to March 2022 on the PubMed and Virtual Health Library (BVS) databases. For this purpose, the following descriptors were used: Gastrointestinal Neoplasms; Epigenomics; Therapeutics; Gene Expression. Search strategies were constructed from the combination of these descriptors with the Boolean operator AND. In the end, 15 articles were selected for the effective discussion. RESULTS AND DISCUSSION: After a complete balance of the studies, most of them established that the relevance of epigenetic signatures for oncology patients is centered on the possibility of developing improved therapeutic pathways, building the emergence of potential biomarkers for neoplasms, which deepens the threshold of clinical monitoring. CONCLUSION: The set of analyzed studies demonstrated that changes in the methylation of some genes can cause important changes in gene expression, through signal expansion, activation, or transcriptional repression, and that the proposal of new effective therapeutic strategies for the epigenetic and neoplasms context can use information from specific transcriptional coactivators. Finally, new studies are needed to deepen the understanding between previous delimitations of the relationship between the epigenetic context and the presented cancer scenario.
Keywords: Gastrointestinal Neoplasms; Epigenomics; Therapeutics; Gene Expression.
1.INTRODUÇÃO
O câncer é uma doença complexa que envolve alterações de múltiplos processos genéticos e epigenéticos, contribuindo estes como fatores centrais para a evolução da doença neoplásica. (PAJARES et al., 2021).
É importante frisar que o câncer colorretal, o câncer de fígado, o câncer de estômago, o câncer de pâncreas e o câncer de esôfago são as principais causas de mortes relacionadas aos subtipos dessa moléstia em todo o mundo (GRADY; YU; MARKOWITZ, 2021). O câncer de estômago, por exemplo, é uma malignidade mortal que aflige cerca de um milhão de pessoas no globo e a sobrevida do paciente, em grande parte, é pobre devido ao diagnóstico tardio e a terapias aquém das ideais (PADMANABHAN; USHIJIMA; TAN, 2017).
Nesse contexto, ao abraçar a epigenética e conceituá-la como modificações potencialmente hereditárias na expressão genética que não envolvem, necessariamente, mudanças na sequência de DNA, há a inclusão, no escopo dessas alterações, de processos como a metilação de DNA, modificações de histona, remodeladores de cromatina e RNAs não codificantes (BARETTI; AZAD, 2018).
Essas alterações, em última análise, mediam o comportamento clínico dos pacientes com ou sem o diagnóstico oncológico e podem ainda ser utilizadas como biomarcadores para determinação do risco de desenvolvimento neoplásico, em que pese sua prévia detecção e consolidação de um prognóstico a seguir, à luz de uma previsão vivaz de resposta à terapia oncológica (GRADY; YU; MARKOWITZ, 2021).
No cenário de cânceres gastrointestinais, diversos tipos de tecidos são adequados para as análises do DNA metilado, como tecidos tumorais, sangue, plasma e fezes, e alguns desses marcadores já são utilizados no cenário clínico. Os recentes avanços nas abordagens epigenômicas de todo o genoma estão permitindo o mapeamento abrangente do metiloma do câncer, fornecendo novos caminhos para tais biomarcadores numa abordagem eficaz de prognóstico e diagnóstico dessas doenças neoplásicas (WONG et al., 2018).
Nesse cenário, direcionar mecanismos epigenéticos pode ser uma estratégia terapêutica interessante para superar a heterogeneidade dos cânceres das vias gastrointestinais, já que os esforços realizados até o momento entregam sólidos resultados promissores (CANALE et al., 2020).
Dessa forma, a partir de uma revisão sistemática integrativa, o presente artigo buscou responder a seguinte pergunta-guia: qual a relevância terapêutica da assinatura epigenética para os pacientes oncológicos, em que pese o contexto de detecção, acompanhamento e prognóstico voltados às neoplasias do trato gastrointestinal, ao envolver, em termos comparativos, os atuais sistemas de gerenciamento cancerígeno?
2. METODOLOGIA
O fomento desta revisão integrativa, elaborada a partir de uma vertente quantitativa e descritiva perpassou pela realização de buscas até março de 2022 nas plataformas de dados PubMed e Biblioteca Virtual em Saúde (BVS). Para tanto, foram utilizados os seguintes descritores: Gastrointestinal Neoplasms; Epigenomics; Therapeutics; Gene Expression. Estratégias de busca foram construídas a partir da combinação desses descritores com o operador booleano AND. Tal análise contemplou, dentro dos últimos 5 anos, artigos em inglês, português e espanhol.
Os fatores de inclusão foram pautados em trabalhos que avaliaram a relação entre cânceres do trato gastrointestinal (CTG) e epigenética ou entre terapia epigenômica e expressão gênica relacionada a neoplasias gastrointestinais.
Foram descartadas publicações que abordavam os CTGs relacionados estritamente às questões genéticas, excluindo a vertente epigenética do processo cancerígeno. Situações cujas patologias associadas eram o centro do debate ou, ainda, cujos enfoques direcionaram a testes clínicos em não-humanos também foram descartadas. Além disso, foram descartados, também, os artigos de opinião, cartas ao editor, monografias, erratas, entrevistas, editoriais, revisões narrativas, revisões integrativas e revisões sistemáticas sem metanálise.
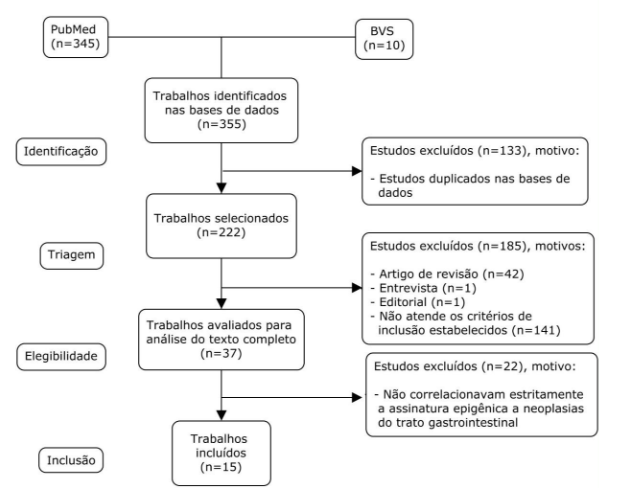
A seleção dos artigos foi realizada pela aplicação dos critérios de inclusão e exclusão a partir das seguintes etapas consecutivas: foram lidos inicialmente os títulos, depois os resumos e, por fim, os artigos selecionados após a leitura dos resumos, então, tiveram o seu texto completo analisado. Publicações duplicadas foram excluídas (133), quando identificadas após a leitura dos títulos, resultando em 222 trabalhos para análise nas posteriores etapas de seleção. Títulos que não abordavam a situação epigênica dos CTGs também foram excluídos (120). Ao fim da análise dos resumos, restaram 37 artigos para a realização da leitura do texto completo, posto que foram retirados os que englobavam um enfoque temático distante da noção de terapia epigênica (65).
Durante a leitura do texto completo, foram excluídos 22 artigos que também não correlacionaram, estritamente, a assinatura epigênica a neoplasias do trato gastrointestinal como eixo principal, restando 15 para serem discutidos e utilizados como evidências científicas para a escrita da presente revisão. Os resultados das estratégias de busca e das etapas de seleção são apresentados na figura 1.
FIGURA 1. Resultados das estratégias de busca e seleção dos artigos
3. RESULTADOS E DISCUSSÃO
QUADRO 1. Principais informações dos artigos selecionados para a escrita da revisão
Artigo Metodologia Principais Informações Objetivo Resultados Desfecho/Conclusão DELLA CHIARA et al., 2020. Estudo coorte prospectivo. Caracterizar epigeneticamente o câncer colorretal humano usando a descoberta do estado da cromatina de novo em uma biblioteca de diferentes organóides derivados de pacientes. Os coativadores transcricionais YAP/TAZ atuam como reguladores chave dos potenciadores ganhos de câncer colorretal conservados. Os mesmos intensificadores ligados a YAP/TAZ exibem perfis ativos de cromatina em diversos tumores humanos, destacando uma religação epigenética pan-câncer que, no nível de uma única célula, distingue as populações de células malignas das normais. A inibição de YAP/TAZ em organóides tumorais estabelecidos causa extensa morte celular, revelando seu papel essencial na manutenção do tumor. Uma camada comum de reprogramação potenciadora alimentada por YAP/TAZ é fundamental para o estado da célula cancerígena e pode ser explorada para o desenvolvimento de vias terapêuticas melhoradas. DÍEZ-VILLANUEVA et al., 2020. Estudo coorte prognóstico. Obter informações sobre o papel da metilação do DNA no crescimento maligno do câncer de cólon. Não foram encontradas diferenças na metilação entre tecidos adjacentes e saudáveis, mas diferenças claras foram encontradas entre amostras adjacentes e tumorais. Identificamos ilhas CpG hipermetiladas localizadas em regiões promotoras que direcionam a expressão gênica diferencial de fatores de transcrição e seus genes-alvo. Alterações na metilação de alguns genes provocam alterações importantes na expressão gênica, por meio da expansão do sinal por meio da ativação/repressão da transcrição. FARHANA et al., 2021. Estudo observacional transversal. Examinar se os genes envolvidos no controle epigenético dos mecanismos de reparo do DNA foram afetados durante o tratamento com camptotecina encapsulada em nanopartículas de β-ciclodextrina–EDTA–Fe3 O4. Os genes que controlam o reparo incompatível, o reparo por excisão de base e a recombinação de homólogos foram regulados negativamente nessas células cancerígenas. O tratamento com CPT-CEF aliviou essa repressão. Observamos que o CPT-CEF exerce seu efeito possivelmente afetando o mecanismo de reparo do DNA através da modulação epigenética envolvendo os genes HMGB1, APEX1 e POLE3. O tratamento com CPT-CEF poderia inibir a proliferação de células HT29, provavelmente revertendo essa desregulação. FANG et al., 2019. Estudo clínico não-randomizado. Analisar a expressão de KMT2A em tecidos de pacientes. O KMT2A foi superexpresso em tecidos de câncer colorretal em comparação com tecidos normais adjacentes e sua expressão foi positivamente correlacionada com os estágios do câncer. Em células HCT116 e DLD1 KMT2A-knockdown, a invasão celular e a migração foram consequentemente suprimidas. Além disso, a depleção de KMT2A suprimiu efetivamente a metástase do câncer in vivo. O KMT2A promove epigeneticamente a progressão do câncer, visando CTSZ, que tem funções específicas na invasão e metástase do câncer. KEL et al., 2019. Estudo de coorte prospectivo. Encontrar possíveis relações causais entre alterações epigenéticas (metilações do DNA) em regiões reguladoras de genes que afetam os locais de ligação do fator de transcrição (TFBS) e alterações na expressão gênica A identificação de potenciais biomarcadores epigenéticos de metilação do DNA foi realizada usando o serviço web de análise multi-ômica totalmente automático “My Genome Enhancer”s por meio da análise de uma extensa coleção de dados completos de expressão gênica do genoma (RNA-seq) e dados de metilação de DNA de ilhas CpG genômicas. Foi selecionado um conjunto mínimo de 6 biomarcadores, que juntos alcançam o melhor potencial de detecção de câncer. Os marcadores incluem posições hipermetiladas em regiões reguladoras dos seguintes genes: CALCA, ENO1, MYC, PDX1, TCF7, ZNF43. NAIR et al., 2020. Estudo clínico não-randomizado. Elucidar as alterações epigenéticas por trás da regulação positiva de pontos de controle imunológico e marcadores de exaustão de células T em pacientes com câncer colorretal. As expressões de mRNA de PD-1, TIM-3, CTLA-4, PD-L1 e TOX2 foram significativamente aumentadas em CD4 + e CD8 +linfócitos infiltrantes de tumor e tecidos tumorais de CRC em massa. A trimetilação da histona 3 lisina 9 foi regulada negativamente e a trimetilação da histona 3 lisina 4 foi regulada positivamente nos promotores PD-L1 e TOX2 em tecidos tumorais, sugerindo que a regulação positiva de PD-L1 e TOX2 em tumores de câncer colorretal poderia ser mediada pela ativação da histona 3 lisina 4 trimetilação. Modificações epigenéticas em promotores de pontos de checagem imune e genes de esgotamento de células T podem induzir sua regulação positiva e potencialmente implicar o uso de modificadores epigenéticos para aumentar a imunidade antitumoral em pacientes com câncer colorretal. NAJGEBAUER et al., 2019. Estudo observacional descritivo. Analisar alterações relacionadas ao câncer na expressão gênica e metilação do DNA em miofibroblastos primários derivados de tumores gástricos e esofágicos. Novas assinaturas de metilação de DNA específicas de CAM foram identificadas, que não são observadas em miofibroblastos derivados de tecidos adjacentes compatíveis com pacientes ou miofibroblastos derivados de tecidos normais correspondentes. A análise das alterações correlacionadas na metilação do DNA e na expressão gênica mostra que diferentes padrões de metilação específica do gene têm o potencial de conferir alterações pró-tumorais no metabolismo, sinalização celular e respostas diferenciais à hipóxia. NIINUMA et al., 2019. Estudo coorte prospectivo. Mostrar que a depleção de UHRF1 induz rapidamente a desmetilação do DNA em todo o genoma em células cancerígenas A depleção de UHRF1 mais a inibição de HDAC efetivamente restaura a expressão de genes epigeneticamente silenciados em células CRC. A inibição de HDAC suprime fortemente a proliferação de células CRC com depleção de UHRF1. Um estado de cromatina fechada persiste após a desmetilação induzida pela depleção de UHRF1 em células cancerígenas e que o direcionamento duplo de UHRF1 e modificadores de histona pode restaurar a expressão de genes silenciados epigeneticamente. A manutenção da metilação do DNA em células CRC é altamente dependente de UHRF1; a depleção de UHRF1 induz rapidamente a desmetilação do DNA, embora seja insuficiente para reativar totalmente os genes silenciados; e o direcionamento duplo de UHRF1 e HDAC pode ser uma nova estratégia terapêutica eficaz. NIINUMA et al., 2021. Estudo observacional transversal. Obter mais informações sobre o epigenoma de tumores estromais gastrointestinais, analisando a modificação de histonas em todo o genoma e a metilação do DNA em células GIST. A análise ChIP-seq revelou que o tratamento medicamentoso aumentou os níveis de H3K4me3 em retrotransposons, incluindo retrovírus endógenos (ERV). A hipermetilação de MEG3 é um evento frequente e um indicador de pior prognóstico em pacientes com GIST. Os inibidores epigenéticos podem ativar a sinalização do interferon via mimetismo viral nas células de tumores estromais gastrointestinais. Além disso, os dados do epigenoma podem ser um recurso útil para identificar novos genes relacionados ao de tumores estromais gastrointestinais. QU et al., 2020. Estudo clínico não-randomizado. Avaliar a expressão diferencial de microRNAs em carcinoma gástrico com status de microssatélite diferente, analisando os dados de microRNA de alto rendimento. 56 e 67 amostras de adenocarcinoma gástrico foram positivas para instabilidade de microssatélites baixo e alto. Nas 67 amostras com alto status de instabilidade de microssatélites, os níveis de expressão de 14 microRNAs foram regulados positivamente, mas cinco microRNAs foram regulados negativamente. MiR-30a-3p e miR-105-5p são potenciais biomarcadores para o adenocarcinoma gástrico MSI-H, possivelmente por alterar a expressão de genes reparadores de danos no DNA. SONG et al., 2020. Estudo observacional transversal. Desenvolver uma assinatura baseada em múltiplos genes para prever a sobrevida global em adenocarcinomas gastrointestinais. Um total de 7.621 genes significativamente expressos diferencialmente foram identificados entre adenocarcinomas gastrointestinais e tecidos normais. A análise de sobrevivência conjunta demonstrou que a combinação da expressão e nível de metilação de 28 genes foi significativamente associada com sobrevida global em pacientes com adenocarcinomas gastrointestinais. Uma nova assinatura epigenética foi estabelecida para pan-adenocarcinomas gastrointestinais através dos limites anatômicos para prever com precisão a sobrevida de pacientes afetados por essas malignidades. TENG et al., 2020. Estudo coorte prospectivo. Identificar a heterogeneidade da metilação do DNA (DNAm) inter e intratumoral, alterações do DNAm em todo o epigenoma junto com os reguladores funcionais envolvidos nas regiões hiper ou hipometiladas e sua associação com características clínicas. ESCC manifestou substancial heterogeneidade de DNAm inter e intratumoral. A alta heterogeneidade do DNAm intratumoral foi associada à metástase linfonodal e pior sobrevida global. Regiões hipermetiladas em ESCC foram enriquecidas em promotores de numerosos fatores de transcrição, e regiões não codificantes desmetiladas relacionadas à ligação do fator de transcrição RXR pareceram contribuir para o desenvolvimento de ESCC. Variações de DNAm em nível populacional de alta resolução em ESCC foram apresentadas, bem como evidências de ampla heterogeneidade de DNAm intratumoral entre pacientes com ESCC e inúmeras alterações de DNAm associadas à carcinogênese e progressão de ESCC. WANG et al., 2019. Estudo de rastreamento. Identificar um painel de marcadores de metilação do DNA para o diagnóstico precoce do câncer colorretal. Os modelos produziram alta sensibilidade e especificidade para distinguir câncer colorretal e seus precursores (adenoma colorretal e tumor colorretal de disseminação lateral) de controles normais em oito conjuntos de dados independentes e nossas amostras recém-coletadas. O modelo baseado em dois CpG mostrou alta especificidade na exclusão de doenças inflamatórias intestinais e outros 13 tipos de câncer. Um modelo de diagnóstico baseado em dois CpGs (cg09239744 e cg12587766) pode ser uma ferramenta poderosa para triagem de câncer colorretal. YOSHIDA et al., 2017. Estudo comparativo. Demonstrar que o FAT4, um novo supressor de tumor identificado pelo sequenciamento do exoma de GC, é silenciado por metilação, a qual está envolvida na cancerização do campo epigenético para câncer gástrico. A metilação do FAT4 foi associada à presença do fenótipo metilador da ilha CpG, mas não ao prognóstico, invasão tumoral, metástase em linfonodos ou tipos histológicos. Em mucosas gástricas não cancerosas, FAT4 alto níveis de metilação foram associados com a presença de GC e infecção por Helicobacter pylori O FAT4 foi silenciado por metilação em GCs. Sua metilação na mucosa gástrica foi associada à infecção por H. pylori e provavelmente contribuiu para a cancerização do campo epigenético. ŽLAJPAH, BOŠTJANČIČ, ZIDAR, 2020. Estudo coorte prospectivo.. Identificar reguladores (epi)genéticos da osteopontina (OPN, codificado pelo gene SPP1) da mucosa do cólon normal ao adenoma, adenoma com carcinoma inicial e carcinoma avançado. A expressão de hsa-miR-146a-5p é inversamente proporcional ao nível de expressão de SPP1 e que a expressão também pode ser controlada pelo número de cópias e metilação. A expressão de SPP1 e seu número de cópias, estado de metilação e expressão de seus reguladores podem ser usados como um potencial biomarcador de câncer colorretal.
O Quadro 1 integra os artigos selecionados para a escrita da presente discussão e elenca uma síntese das respectivas metodologias, objetivos, resultados e conclusões dos trabalhos.
Após completo balanço dos estudos, a maioria deles estabeleceu que a relevância da assinatura epigenética para os pacientes oncológicos é centrada na possibilidade do desenvolvimento de vias terapêuticas melhoradas, edificando a surgência de potenciais biomarcadores de neoplasias, o que aprofunda o limiar de acompanhamento clínico. Houve, para isso, ponderações lógicas e sequenciais dentro do escopo dos seguintes tipos de pesquisa: estudo coorte prospectivo, estudo observacional descritivo, estudo observacional transversal, estudo clínico não-randomizado, estudo de rastreamento e estudo comparativo.
3 trabalhos distintos versam a respeito do câncer colorretal, são eles: Della Chiara e outros colaboradores (2020), Nair e outros colaboradores (2020) e Wang e outros colaboradores (2019). Outros três se aprofundaram nos ditames do câncer gástrico: Najgebauer e outros colaboradores (2020), Qu e outros colaboradores (2020) e Yoshida e outros colaboradores (2017). Outros dois se concentraram em estudos cujas abordagens de cânceres ao trato gastrointestinal como um todo, quais foram: Niinuma e outros colaboradores (2021), Song e outros colaboradores (2020).
Ao adentrar numa observação mais acurada dos trabalhos, três deles, que são Della Chiara e outros colaboradores (2020), Farhana e outros colaboradores (2021) e Niinuma e outros colaboradores (2019) propõem evidenciar novas estratégias terapêuticas eficazes para esse contexto entre vínculo epigenético e neoplasias. O primeiro trabalho aponta que alguns co ativadores transcricionais específicos, tais como o YAP/TAZ, são fundamentais para o estado da célula cancerígena e isso, invariavelmente, pode ser explorado para o desenvolvimento de vias terapêuticas em neoplasias colorretais, por exemplo. O segundo, por sua vez, demonstra que a Camptotecina encapsulada em nanopartículas de β-ciclodextrina–EDTA–Fe3 O4 poderia inibir a proliferação de células HT29, que são de adenocarcinoma de colo humano, o que possibilita a reversão da desregulação cancerígena. Já o terceiro trabalho, ao explicitar que a manutenção da metilação do DNA em células cancerígenas é altamente dependente do gene UHRF1 e a depleção desse mesmo gene impulsiona a desmetilação do DNA, há uma indução à conclusão de que o direcionamento duplo de controle à metilação ou desmetilação de alguns genes pode ser uma nova estratégia terapêutica eficaz para tratamento da enfermidade.
É importante frisar que alguns potenciais marcadores cancerígenos colocam em sintonia, ao envolver a metilação aberrante ou hidroximetilação de determinados genes, os trabalhos de Žlajpah, Boštjančič e Zidar (2020), Zhu e outros colaboradores (2020), Niinuma e outros colaboradores (2019), Najgebauer e outros colaboradores (2019), Kel e outros colaboradores (2019) e Díez-Villanueva e outros colaboradores (2020). Sob essa perspectiva, há que se enfatizar que a profundidade alcançada pelos estudos pavimenta noções de entendimento mais amplo às demandas epigenéticas do processo carcinogênico e, dessa forma, com a manipulação desse conhecimento, é possível aplicar clinicamente novas estratégias para o manejo à qualidade e prolongamento de vida ou, a nível preventivo, afastar probabilidades de desenvolvimento maligno.
Nesse sentido, os trabalhos de Žlajpah, Boštjančič e Zidar (2020), ao exemplificar que a expressão do microRNA hsa-miR-146a-5p é inversamente proporcional ao nível de expressão do gene SPP1, o de Niinuma e outros colaboradores (2019), ao demonstrar que a depleção do gene UHRF1 induz rapidamente à desmetilação do DNA em todo o genoma de células cancerígenas, ou, propriamente, o trabalho de Fang e outros colaboradores (2019) que, ao delimitar o que gene KMT2A promove epigeneticamente a progressão de um tipo cancerígeno, condizem justificativas para todas as assertivas de que esse esboço de conhecimento torna factível melhores condições e orientações à prática médico-genética.
É importante frisar que Qu e outros colaboradores (2020), assim como Žlajpah, Boštjančič e Zidar (2020), também explicita certos microRNAs como potenciais biomarcadores para o adenocarcinoma gástrico. No caso de Que outros colaboradores, houve, a nível de evidência científica, descrições do MiR-30a-3p e do miR-105-5p como tais potenciais.
Com esse enfoque de demarcação, o trabalho de Song e outros colaboradores (2020) encontrou em seus resultados uma média de 7.621 genes expressos diferencialmente entre adenocarcinomas gastrointestinais e tecidos normais. Com essa margem, é possível até ampliar os horizontes de Yoshida e outros colaboradores (2017), posto que o gene FAT4, um supressor de tumor, é silenciado por metilação, e, com novas perspectivas de estudo escancaradas por Song e outros colaboradores (2020), deve-se pesar as condições de interação gênica no esboço de novos resultados no processo a cancerização gastrointestinal.
Dos seis estudos de coorte analisados, quais foram Della Chiara e outros colaboradores (2020), Díez-Villanueva e outros colaboradores (2020), Kel e outros colaboradores (2019), Teng e outros colaboradores (2020), Niinuma e outros colaboradores (2019) e Žlajpah, Boštjančič e Zidar (2020), o foco principal em identificar reguladores epigenéticos assenta resultados específicos para novos horizontes de pesquisa genômica. Ao pautar variações, interações, metilação ou hipermetilação como variáveis passíveis de condicionar uma qualidade de vida mais plural à população, há que se pensar que o estágio prospectivo dos estudos inter relacionam observação, comparação e condições probabilísticas.
4. CONCLUSÃO
Com a evidência de informações epigenéticas acerca do câncer, o conjunto de trabalhos analisados demonstrou que alterações na metilação de alguns genes podem provocar importantes mudanças na expressão gênica, por meio de expansão do sinal, ativação ou repressão da transcrição. Sob essa perspectiva, ainda, é possível que vias terapêuticas sejam melhoradas por meio de reprogramação do estado de células cancerígenas, o que propõe a melhoria de cenários clínicos, bem como a sobrevida de pacientes acometidos por processos oncológicos.
Nesse viés, é perceptível que outras intervenções podem apresentar-se como capazes de reverter a desregulação, a exemplo do tratamento com CPT-CEF, o qual alivia a repressão e afeta o mecanismo de reparo do DNA. Dessa forma, é notório também que conjunto de biomarcadores podem alcançar um potencial de detecção de câncer aumentado, o que facilitaria o tratamento precoce e ofereceria a melhoria de bem-estar dos pacientes a longo prazo.
Notou-se, ainda, que a proposta de novas estratégias terapêuticas eficazes para o âmbito epigenético e neoplasias pode utilizar informações advindas de co ativadores transcricionais específicos. Por fim, novos estudos são necessários para aprofundar o entendimento entre delimitações prévias da relação do contexto epigenético com o cenário cancerígeno apresentado.
5. CONTRIBUIÇÕES
Todos os autores contribuíram substancialmente na concepção e/ou no planejamento do estudo; na obtenção, na análise e/ou interpretação dos dados; assim como na redação e/ou revisão crítica; e aprovaram a versão final a ser publicada.
6. DECLARAÇÃO DE CONFLITO DE INTERESSE
Nada a declarar.
7. FONTES DE FINANCIAMENTO
Não há.
8. REFERÊNCIAS
BARETTI, M.; AZAD, N. S. The role of epigenetic therapies in colorectal cancer. Current Problems in Cancer, v. 42, n. 6, p. 530–547, 1 nov. 2018. Disponível em: https://pubmed.ncbi.nlm.nih.gov/29625794/. Acesso em: 26 mar 2022.
CANALE, M. et al. Epigenetic Mechanisms in Gastric Cancer: Potential New Therapeutic Opportunities. International Journal of Molecular Sciences, v. 21, n. 15, p. 5500, 31 jul. 2020. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7432799/. Acesso em: 26 mar 2022.
DELLA CHIARA, G. et al. Epigenomic landscape of human colorectal cancer unveils an aberrant core of pan-cancer enhancers orchestrated by YAP/TAZ. Nature Communications, v. 12, n. 1, 20 abr. 2021. Disponível em: https://www.nature.com/articles/s41467-021-22544-y. Acesso em: 26 mar 2022.
DÍEZ-VILLANUEVA, A. et al. DNA methylation events in transcription factors and gene expression changes in colon cancer. Epigenomics, v. 12, n. 18, p. 1593–1610, 1 set. 2020. Disponível em: https://pubmed.ncbi.nlm.nih.gov/32957849/. Acesso em: 26 mar 2022.
FANG, Y. et al. KMT2A histone methyltransferase contributes to colorectal cancer development by promoting cathepsin Z transcriptional activation. Cancer Medicine, v. 8, n. 7, p. 3544–3552, 15 maio de 2019. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6601586/. Acesso em: 26 mar 2022.
FARHANA, A. et al. Nanoparticle-Encapsulated Camptothecin: Epigenetic Modulation in DNA Repair Mechanisms in Colon Cancer Cells. Molecules, v. 26, n. 17, p. 5414, 6 set. 2021. Disponível em: https://pubmed.ncbi.nlm.nih.gov/34500845/. Acesso em: 26 mar 2022.
GRADY, W. M.; YU, M.; MARKOWITZ, S. D. Epigenetic Alterations in the Gastrointestinal Tract: Current and Emerging Use for Biomarkers of Cancer. Gastroenterology, v. 160, n. 3, p. 690–709, 1 fev. 2021. Disponível em: https://pubmed.ncbi.nlm.nih.gov/33279516/. Acesso em: 26 mar 2022.
KEL, A. et al. Walking pathways with positive feedback loops reveal DNA methylation biomarkers of colorectal cancer. BMC Bioinformatics, v. 20, n. S4, abr. 2019. Disponível em: https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-019-2687-7. Acesso em: 26 mar 2022.
NAIR, V. S. et al. Epigenetic regulation of immune checkpoints and T cell exhaustion markers in tumor-infiltrating T cells of colorectal cancer patients. Epigenomics, v. 12, n. 21, p. 1871–1882, nov. 2020. Disponível em: https://pubmed.ncbi.nlm.nih.gov/33169618/. Acesso em: 26 mar 2022.
NAJGEBAUER, H. et al. Integrated omics profiling reveals novel patterns of epigenetic programming in cancer-associated myofibroblasts. Carcinogenesis, v. 40, n. 4, p. 500–512, 10 jun. 2019. Disponível em: https://pubmed.ncbi.nlm.nih.gov/30624614/. Acesso em: 26 mar 2022.
NIINUMA, T. et al. UHRF1 depletion and HDAC inhibition reactivate epigenetically silenced genes in colorectal cancer cells. Clinical Epigenetics, v. 11, n. 1, 7 maio de 2019. Disponível em: https://pubmed.ncbi.nlm.nih.gov/31064417/. Acesso em: 26 mar 2022.
NIINUMA, T. et al. An Integrated Epigenome and Transcriptome Analysis to Clarify the Effect of Epigenetic Inhibitors on GIST. Anticancer Research, v. 41, n. 6, p. 2817–2828, 1 jun. 2021. Disponível em: https://pubmed.ncbi.nlm.nih.gov/34083271/. Acesso em: 26 mar 2022.
PADMANABHAN, N.; USHIJIMA, T.; TAN, P. How to stomach an epigenetic insult: the gastric cancer epigenome. Nature Reviews Gastroenterology & Hepatology, v. 14, n. 8, p. 467–478, 17 maio de 2017. Disponível em: https://pubmed.ncbi.nlm.nih.gov/28513632/. Acesso em: 26 mar 2022.
PAJARES, M. J. et al. Epigenetic Regulation of microRNAs in Cancer: Shortening the Distance from Bench to Bedside. International Journal of Molecular Sciences, v. 22, n. 14, p. 7350, 1 jan. 2021. Disponível em: https://pubmed.ncbi.nlm.nih.gov/34298969/. Acesso em: 26 mar 2022.
QU, X. et al. Differential microRNA expression profiles associated with microsatellite status reveal possible epigenetic regulation of microsatellite instability in gastric adenocarcinoma. Annals of Translational Medicine, v. 8, n. 7, p. 484–484, abr. 2020. Disponível em: https://pubmed.ncbi.nlm.nih.gov/32395528/. Acesso em: 26 mar 2022.
SONG, W. et al. Genome-wide methylation and expression profiling identify a novel epigenetic signature in gastrointestinal pan-adenocarcinomas. Epigenomics, v. 12, n. 11, p. 907–920, jun. 2020. Disponível em: https://pubmed.ncbi.nlm.nih.gov/32166971/. Acesso em: 26 mar 2022.
TENG, H. et al. Inter- and intratumor DNA methylation heterogeneity associated with lymph node metastasis and prognosis of esophageal squamous cell carcinoma. Theranostics, v. 10, n. 7, p. 3035–3048, 10 fev. 2020. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7053185/. Acesso em: 26 mar 2022.
WANG, X. et al. Genome-wide analysis of DNA methylation identifies two CpG sites for the early screening of colorectal cancer. Epigenomics, v. 12, n. 1, p. 37–52, jan. 2020. Disponível em: https://pubmed.ncbi.nlm.nih.gov/31762318/. Acesso em: 26 mar 2022.
WONG, C. C. et al. Epigenomic biomarkers for prognostication and diagnosis of gastrointestinal cancers. Seminars in Cancer Biology, v. 55, p. 90–105, 1 abr. 2019. Disponível em: https://pubmed.ncbi.nlm.nih.gov/29665409/. Acesso em: 26 mar 2022.
YOSHIDA, S. et al. Epigenetic inactivation of FAT4 contributes to gastric field cancerization. Gastric Cancer, v. 20, n. 1, p. 136–145, 20 jan. 2016. Disponível em: https://pubmed.ncbi.nlm.nih.gov/26792292/. Acesso em: 26 mar 2022.
ŽLAJPAH, M.; BOŠTJANČIČ, E.; ZIDAR, N. (Epi) genetic regulation of osteopontin in colorectal cancerogenesis. Epigenomics, v. 12, n. 16, p. 1389–1403, ago. 2020. Disponível em: https://pubmed.ncbi.nlm.nih.gov/32921164/. Acesso em: 26 mar 2022.
1Acadêmico do Curso de Medicina do Centro Universitário Tiradentes (UNIT-AL). Rua Dr. Zeferino Rodrigues, 315. Apto 405. CEP 57025-043. Maceió – AL, Brasil. thiago.terto@souunit.com.br. (82) 999820954.
2Acadêmica do Curso de Medicina do Centro Universitário Tiradentes (UNIT-AL). Rua Dr. José Castro de Azevedo, Edf. Alisson, 586, Bairro Pitanguinha. CEP: 57052-240. Maceió – AL, Brasil. beatriz.modesto@souunit.com.br. (82) 988026462.
3Acadêmica do Curso de Medicina do Centro Universitário Tiradentes (UNIT-AL). Rua Dr. José Castro de Azevedo, Edf. Alisson, 586, Bairro Pitanguinha. CEP: 57052-240. Maceió – AL, Brasil. bruna.modesto@souunit.com.br. (82) 988026461.
4Professor Titular do Curso de Medicina do Centro Universitário Tiradentes (UNIT-AL). Rua Dr. Antônio Cansanção, 575. Apto 709. CEP 57035-190. Maceió – AL, Brasil. jaim.simoes@souunit.com.br. (82) 996557334.
