ORAL CANCER BIOMARKERS, COMPARATIVE ANALYSIS OF PROTEIN PROFILES EXPRESSED IN ORAL MUCOSA AND SALIVA USING SDS-PAGE AND MASS SPECTROMETRY TECHNIQUES
REGISTRO DOI: 10.69849/revistaft/ma10202401311454
Cicera Peixoto de Azevedo1
Álvaro Arthur do Nascimento Soares2
Vinícius Eduardo Fernandes da Silva3
Maria de Fatima Ferreira da Silva4
Carla Louise Alves de Azevedo5
Pollyanna Almeida dos Santos Abu Hana6
Carlos Arthur Cardoso Almeida7
RESUMO
Este artigo analisa a utilização das técnicas de SDS-PAGE e espectrometria de massas na identificação de potenciais candidatos a biomarcadores proteicos em saliva e mucosa oral para o diagnóstico precoce do câncer oral, identificando e comparando os perfis proteicos das amostras, buscando alternativas menos invasivas para o diagnóstico. A pesquisa se fundamenta na relevância de métodos menos invasivos e de fácil aplicação, para reduzir a mortalidade associada ao carcinoma de células escamosas, que é predominante entre os tumores da cavidade oral. Por meio de uma revisão da literatura, foram selecionados estudos publicados entre 2004 e 2024, que empregaram ambas as técnicas para análise proteômica em amostras de saliva e tecido. Os resultados revelaram a identificação de 385 proteínas, sendo 24 compartilhadas entre as amostras analisadas, destacando diferenças fundamentais nos perfis proteicos comuns às duas matrizes biológicas, com destaque para proteínas como Anexina A1, Hemopexina, Histona 1 Peroxirredoxinas 1 e 2, e Queratinas 1, 4 e 10, Tubulina, foram apontadas como candidatas promissoras a biomarcadores devido ao seu papel associadas a processos tumorais. A saliva, apesar de apresentar menor diversidade proteica, mostrou-se promissora como fonte de biomarcadores. As descobertas reforçam o papel da “proteômica” no aprimoramento do diagnóstico do câncer oral e destacam a necessidade de padronização metodológica para aumentar a sensibilidade e a especificidade das análises. Conclui-se que o estudo pode contribuir para a identificação de potenciais candidatos a biomarcadores no CCE, podendo auxiliar no desenvolvimento de ferramentas diagnósticas e prognósticas, transformar as práticas clínicas e promover maior sobrevida aos pacientes.
Palavras-chave:Biomarcadores. Câncer oral. Células escamosas. SDS-PAGE. Espectrometria de Massas. Diagnóstico precoce.
ABSTRACT
This article analyzes the use of SDS-PAGE and mass spectrometry techniques in the identification of potential candidates for protein biomarkers in saliva and oral mucosa for the early diagnosis of oral cancer, identifying and comparing the protein profiles of the samples, seeking less invasive alternatives for diagnosis. The research is based on the relevance of less invasive and easy-to-apply methods to reduce mortality associated with squamous cell carcinoma, which is predominantly among tumors of the oral cavity. Through a literature review, studies published between 2004 and 2024 were selected, which used both techniques for proteomic analysis in saliva and tissue samples. The results revealed the identification of 385 proteins, 24 of which were shared between the samples demonstrated, highlighting fundamental differences in the protein profiles common to both biological matrices, with emphasis on proteins such as Annexin A1, Hemopexin, Histone 1, Peroxiredoxins 1 and 2, and Keratins 1, 4 and 10, Tubulin, were identified as promising candidates for biomarkers due to their role associated with tumor processes. Saliva, despite having less protein diversity, showed promise as a source of biomarkers. The findings reinforce the role of “proteomics” in improving the diagnosis of oral cancer and highlight the need for methodological standardization to increase the sensitivity and specificity of analyses. It is concluded that the study can contribute to the identification of potential candidates for biomarkers in SCC, which can assist in the development of diagnostic and prognostic tools, transform clinical practices and promote greater patient survival.
Keywords: Biomarkers. Oral cancer. Squamous cells.SDS-PAGE. Mass Spectrometry. Early diagnosis.
1 INTRODUÇÃO
As neoplasias malignas da cavidade oral constituem um sério problema de saúde pública no Brasil e no mundo. Nas últimas décadas, o câncer oral tem se tornado uma doença que preocupa as entidades de saúde pública, devido à sua alta incidência e mortalidade, especialmente em países em desenvolvimento (INCA, 2022). O tipo histológico de tumor maligno mais comum em cavidade oral, presente entre 90 a 95% dos casos, é o carcinoma de células escamosas (CCE), também chamado carcinoma epidermóide ou carcinoma espinocelular (BRIZENO, 2018).
Os fatores de risco para o desenvolvimento do CCE intraoral são, principalmente, fumo associado ao consumo de bebidas alcoólicas, segundo Petti (2009). Atualmente, têm-se relacionado também o vírus do papiloma humano (HPV), principalmente para indivíduos que não fazem uso de fumo e álcool (NOGUEIRA, 2022).
Na busca de marcadores moleculares que auxiliem no diagnóstico precoce e no tratamento de várias doenças humanas, dentre elas o câncer, vários estudos têm focado em alterações nos genes, seus transcritos e produtos proteicos envolvidos em processos celulares importantes.
A combinação de técnicas avançadas, como a eletroforese em gel de poliacrilamida com dodecilsulfato de sódio (SDS-PAGE) e a espectrometria de massa, permite a identificação e comparação de proteínas na mucosa oral e saliva, com base no seu peso molecular permitindo a visualização dos perfis proteicos. Com isso, tornando-se ferramentas relevantes na pesquisa de biomarcadores para o câncer oral. (HU et al., 2005).
Assim, o objetivo dessa pesquisa é identificar potenciais candidatos a biomarcadores proteicos presentes na mucosa oral e na saliva para o diagnóstico precoce do câncer oral, utilizando as técnicas de eletroforese SDS-PAGE e espectrometria de massa. De forma mais específica, buscou-se identificar as proteínas expressas nesses dois materiais biológicos e comparar o perfil da expressão dessas proteínas na mucosa oral e na saliva de pacientes com câncer oral, utilizando as técnicas de SDS-PAGE e espectrometria de massas.
Visando abordar essa problemática, a pesquisa justifica-se pela relevância social da possibilidade de apresentar alternativa menos invasiva para o diagnóstico do câncer oral, surgindo então, a necessidade de métodos de diagnósticos rápidos, seguros, de baixo custo e ampliar o conhecimento científico na área (HA et al., 2009).
2 REVISÃO DA LITERATURA
2.1 Lesões Potencialmente Malignas e Câncer Oral
Eventualmente pode ocorrer o aparecimento de lesões que precedem o surgimento de neoplasias malignas, denominadas de lesões potencialmente malignas. Estas podem permanecer silenciosas por um considerável período de tempo antes de evoluir, ou não, para malignidade e invasão. Cerca de 80% das neoplasias de cavidade oral são precedidas por lesões potencialmente malignas (LPM). O percentual de LPM que sofrem transformação maligna pode variar de 0,13 a 36,4% (Arduino et al., 2009; Ho et al., 2009; Warnakulasuriya et al., 2016). É essencial identificar alterações no padrão dessas lesões que possam sinalizar sua progressão para malignidade, permitindo a intervenção antes que essa transformação aconteça.
As principais lesões potencialmente malignas que acometem a região oral são a eritroplasia, queilose actínica e a leucoplasia oral (LO). A eritroplasia apresenta-se como uma placa eritematosa que não pode ser caracterizada como qualquer outra lesão e histologicamente apresenta a presença de atrofia epitelial e displasia. A queilose actínica é uma alteração causada em lábio associada à exposição solar que apresenta frequente transformação maligna e caracteriza-se, histologicamente, por hiperceratose, acantose, atrofia epitelial, infiltrado inflamatório crônico, elastose solar e displasia (CASTRO E LIMA, 2020.).
Sendo a principal lesão potencialmente maligna que acomete a cavidade oral, a LO. A prevalência global de leucoplasia oral é estimada em 2,60% (intervalo de confiança de 95% 1,72-2,74%) (Brizeno, 2018). Leucoplasia é definida como placa esbranquiçada que não pode ser caracterizada como qualquer outra lesão e apresenta-se, histologicamente, como área hiperceratótica, com acantose, podendo ou não haver displasia, sendo considerada potencialmente maligna quando esta última alteração está presente (NEVILLE, 2011).
As causas da maioria das leucoplasias são o tabaco associado ao álcool e, mais recentemente, a infecção pelo HPV (Syrjanen et al., 2011). De fato, taxas de transformação maligna de leucoplasia em pacientes não fumantes e etilistas são extremamente raras, entretanto sua história natural e o potencial de transformação maligna ainda permanecem incertos (Andrade, 2015). Um dos maiores desafios enfrentados por especialistas em medicina oral é avaliar o status de risco e a real probabilidade dessa lesão evoluir para um CCE de cavidade oral.
A etiopatogenia do CCE oral está ligada, além da predisposição genética, a presença de múltiplos fatores carcinogênicos atuando sobre o epitélio normal, o que resulta em um processo de displasia, a qual se caracteriza pela presença de atipias celulares e perda da estratificação normal do tecido. Dentre os agentes carcinogênicos o fumo apresenta importante função na etiologia de tal patologia, visto que em sua composição há presença de substâncias carcinogênicas (alcatrão e nicotina), aumentando em duas vezes o risco para desenvolver tal neoplasia. Além disso, a associação entre fumo e etanol eleva em 141 vezes a possibilidade de desenvolvimento do CCE oral. Estudos recentes associam o HPV como fator de risco para CCE oral principalmente em pacientes mais jovens não etilistas/tabagistas (SYRJANEN et al., 2011).
O quadro clínico do CCE oral é variado podendo manifestar-se como lesões exofíticas, vegetantes, entretanto a forma mais clássica é uma lesão ulcerada persistente, normalmente com endurecimento e infiltração periférica e durante os estágios iniciais geralmente indolor. A língua é o sítio mais comum para o CCE bucal, correspondendo a 44% dos casos, seguido pelo assoalho bucal com 16%5. O diagnóstico do CCE oral é realizado através de exame clínico, seguido por uma biópsia da lesão com exame anatomopatológico. A tomografia computadorizada é realizada para avaliação da extensão do tumor e da presença de metástases em nódulos cervicais (SASSI et al., 2010).
2.2 A Saliva como Ferramenta Diagnóstica
A saliva é um fluído claro, ligeiramente ácido (pH = 6,0-7,0) e complexo composto por secreções das glândulas salivares maiores: parótidas, submandibulares e sublinguais, bem como milhares de glândulas menores (labiais, bucais, linguais e palatais), fluído crevicular gengival, debris celulares, secreções brônquicas e nasais, células de revestimento, sangue e substâncias exógenas. Em geral, as glândulas salivares humanas produzem cerca de 1-1,5 litros de saliva por dia (Lee: Wong, 2009). Sua composição é uma mistura de componentes e é provavelmente o fluido mais acessível do nosso organismo. Possui um importante papel na manutenção da saúde oral participando de processos como remineralização do esmalte dentário, defesa contra micro-organismos, lubrificação, digestão, modulação de pH e paladar, mantendo assim o equilíbrio do ecossistema da cavidade oral (DaweS, 2008). Esses atributos são decorrentes das características de seus componentes, que incluem proteínas, hormônios, pequenas moléculas como ureia, e eletrólitos como cálcio, bicarbonato, fosfato e fluoreto (Rosa, 2012). As proteínas salivares têm sido estudadas por técnicas bioquímicas tradicionais e proteômicas e centenas delas já foram identificadas tanto na saliva total como em secreções de glândulas individuais, embora aquelas expressas em baixos níveis certamente não foram ainda detectadas (BARBOSA et al, 2012).
Há muitas vantagens no uso da saliva como um fluido biológico. Sua coleta é rápida, fácil, barata e não invasiva. É adequada para coleta domiciliar (sem a necessidade de pessoal qualificado), bem como para pesquisas epidemiológicas. Assim, é muito importante determinar os perfis de expressão destas proteínas e as alterações de um estágio não patológico para um estágio específico da doença (RIVERSOS, 2009).
Há um interesse crescente em todo o mundo pela análise da saliva através de genômica, transcriptômica e proteômica, já que essa é uma fonte não invasiva de rica informação genética. No caso da saliva, dois aspectos principais do diagnóstico do câncer devem ser diferenciados – sendo um o diagnóstico do câncer de boca (que tem contato direto com a saliva) e outro o diagnóstico do câncer em outras localizações. O câncer de boca em estádio avançado geralmente pode ser detectado pela inspeção da cavidade oral. Por outro lado, os carcinomas orais iniciais não são visíveis facilmente e podem não ser diagnosticados precocemente e tratados a tempo. O proteoma salivar também pode ser usado para a detecção do tumor (BRIZENO, 2018).
Os tecidos afetados na maioria das doenças humanas não são de fácil acesso e dificilmente serão utilizados para análise de rotina. Uma de suas principais limitações é a heterogeneidade celular, que pode levar a resultados imprecisos se uma avaliação histopatológica detalhada não for realizada. Ao contrário, os fluidos corporais apresentam características que superam essas limitações e são adequados para o desenvolvimento de ferramentas diagnósticas e prognósticas pouco ou menos invasivas. Além disso, são especialmente apropriados quando um monitoramento longitudinal é necessário (HU et al., 2006).
A saliva é um material biológico bem estudado por abordagens proteômicas. Composta de uma mistura de componentes secretados pelas glândulas salivares e derivados do sangue, ela é provavelmente o fluido mais acessível do nosso organismo (Dawes, 2008). É composta por 99% de água, 0,3% de proteínas e 0,2% de substâncias inorgânicas e orgânicas. Os componentes inorgânicos mais prevalentes incluem: sódio, potássio, cálcio, magnésio, cloreto e carbonatos, enquanto que os componentes orgânicos compreendem amilases, peroxidase, lipase, mucinas, lisozimas, lactoferrinas, calicreínas, cistatinas, hormônios e fatores de crescimento (CHIAPPIN et al., 2007).
No indivíduo saudável, a secreção salivar diária é entre 0,5 e 1,5 L (De Aquino et al., 2020). Possui um importante papel na manutenção da saúde oral participando de processos como remineralização do esmalte dentário, defesa contra microorganismos, lubrificação, digestão, modulação de pH e paladar. O grande interesse na saliva como fluido para diagnóstico tem levado à padronização de processos de coleta e armazenamento principalmente porque vários fatores afetam seu fluxo e composição. Entre esses fatores estão status fisiológico, medicação, alimentação, odores, ritmo circadiano, sexo, idade, a própria constituição do sangue e o grau de atividade das glândulas salivares (HARDT et al., 2005).
A análise proteômica salivar pode ser dificultada pela presença de proteínas de alta abundância que mascaram ou reduzem a sensibilidade de separação. Na saliva, essas proteínas incluem principalmente a alfa-amilase, mucina, albumina e prolina (75% do proteoma da saliva total). Essas proteínas podem impedir a detecção de proteínas de baixa abundância que possam servir como biomarcadores para determinadas doenças. Existem diversos métodos de remoção de proteínas de alta abundância como método de absorção enzima-substrato usado para remoção de alfa-amilase, imunodepleção e diversos tipos de colunas cromatográficas de afinidade, mas em geral são metodologias extremamente caras (KRIEF et al., 2012).
Como alternativa a essa questão, vários pesquisadores têm investido esforços no desenvolvimento e aplicação de técnicas e metodologias para enriquecer amostras de proteínas em diversos tipos de fluídos corporais. As lectinas têm sido amplamente utilizadas em vários estudos glicoproteômicos, como a cromatografia, microarranjos (Zhao et al., 2007) e técnicas de cromatografia de afinidade com múltiplas lectinas (M-LAC). Nesses estudos são usadas colunas cromatográficas de afinidade para capturar proteínas de baixa abundância ou remover glicoproteínas de alta abundância que possam eventualmente prejudicar a análise do proteoma (WANG et al, 2016).
Dessa forma os parâmetros de fluxo e composição salivar têm sido explorados no monitoramento do câncer oral. O sítio anatômico oferece à saliva uma importante vantagem em relação a outros fluidos, além da característica não invasiva e da compatibilidade com abordagens proteômicas, pelo fato de estar em contato com o tecido afetado e, portanto, de estar em contato com proteínas secretadas ou derivadas de células mortas, seu potencial vai desde a utilização para detecção em estágios iniciais até predição de agressividade e prognóstico (BARBOSA et al, 2012).
Proteínas detectadas na saliva como o antígeno carcinoembriogênico (CEA), defensina-1, fator de necrose tumoral (TNF-), interleucina – 1(IL-1), 6, 8 e o antígeno CD44 demonstraram aumento em sua detecção em pacientes com câncer de boca. A maioria desses estudos se baseou em ensaios imunológicos de produtos gênicos individuais (Arellano-Garcia et al., 2008; John et al., 2004). Espera-se que os biomarcadores proteômicos, quando combinados, aumentem a sensibilidade e especificidade da detecção do câncer humano.
Níveis salivares aumentados de defensina-1, antígeno cancerígeno CA 15-3, proteínas marcadoras tumorais, como c-erb-2 ou CA-125, anticorpos contra a proteína supressora de tumor p53, -1-antitripsina (AAT), hepatoglobina (HAP), C3 do complemento, hemopexina e transtirretina parecem marcadores promissores do câncer de boca e de outros tumores malignos. No futuro, um perfil proteômico global da saliva com métodos de análise do proteoma recém-desenvolvidos provavelmente resultará em outras sequências de peptídeos candidatos para detecção com alta sensibilidade (HU; LOO; WONG, 2006; FABIAN; FEJERDY; CSERMELY, 2008; JESSIE et al., 2013).
Alguns estudos de proteômica salivar como ferramenta diagnóstica pontuam como uma das principais dificuldades o fato que muitos analitos informativos estão geralmente presentes em quantidades menores nos fluidos orais, e que o perfil proteico da saliva é dominado por um subgrupo de proteínas abundantes, principalmente α-amilases salivares, albuminas, mucinas e imunoglobulinas que compõem aproximadamente 75% do proteoma total e mascaram potenciais biomarcadores de baixa abundância, esses autores defendem que a remoção dessas proteínas melhora a detecção de potenciais biomarcadores expressos em pequena quantidade (KRIEF et al., 2012).
As lectinas têm sido amplamente utilizadas em vários estudos glicoproteômicos, como a cromatografia microarranjos (ZHAO et al., 2007) e técnicas de cromatografia de afinidade com múltiplas lectinas (M-LAC). Nesses estudos são usadas colunas cromatográficas de afinidade para capturar proteínas de baixa abundância ou remover glicoproteínas de alta abundância que possam eventualmente prejudicar a análise do proteoma (Wang; Wu; Hancock, 2006). As lectinas são uma classe estrutural diversa de proteínas, possuindo como característica comum a capacidade de se ligar especificamente e reversivelmente a carboidratos e, eventualmente, aglutinar células (Sharon; Lis, 2004; Nascimento et al., 2016). São isoladas de fontes distintas, como vírus, bactérias, fungos, algas, animais e plantas, elas mostram especificidade para carboidratos distintos, como manose, ácido siálico, fucose, N-acetilglucosamina, galactose / N Acetilgalactosamina, glicanos complexos e glicoproteínas.
As lectinas podem reconhecer carboidratos e glicoconjugados em células, seções de tecidos e fluidos biológicos, sendo ferramentas valiosas em biotecnologia, incluindo diagnóstico e aplicações farmacológicas e terapêuticas (HASSAN et al., 2015).
Quando comparado ao sangue e outros fluidos biológicos, a saliva pode expressar marcadores mais sensíveis e específicos para certas doenças da cavidade oral. A saliva contém proteínas expressas localmente distintas do soro que podem ser melhores indicadores de alterações bucais. Há razões convincentes para usar a saliva como fluido diagnóstico para monitorar o início e a progressão do câncer de boca. A saliva é o fluido que está em contato direto com as lesões e observa-se nela o aumento do RNA e das proteínas associadas ao câncer de cavidade oral (WU et al., 2010). É fácil de armazenar e transportar, não coagula e pode refletir o estado fisiológico e/ou patológico atual de um indivíduo (ZORZETTO, 2017).
2.3 Proteômica e Câncer de Boca
A importância de se analisar as proteínas reside no fato de que elas desempenham funções finais tanto fisiológicas quanto patológicas nas células, além disso, estão sujeitas às modificações pós-traducionais, as quais vão influenciar o enovelamento correto para formação da estrutura tridimensional, a localização, a interação com outras proteínas, formando a estrutura quaternária extremamente importante para se tornarem funcionais. A taxa de transcrição, medida através de RNA mensageiro, nem sempre tem uma relação direta com a atividade biológica da proteína ou das proteínas (no caso de haver clivagem), sua função ou quantidade (BISCH, 2004).
A proteômica é uma das metodologias mais significativas para compreender a função do gene, embora seja muito mais complexa do que a genômica. Flutuações no nível de expressão gênica podem ser determinadas pela análise do transcriptoma ou proteoma para discriminar entre dois ou mais estados biológicos da célula. Assim, a proteômica seria considerada o conjunto de dados mais relevante para caracterizar um sistema biológico (COX; MANN, 2007).
Os biomarcadores foram usados primariamente como indicadores prognósticos para os pacientes com tumores de cabeça e pescoço. Mais recentemente, o papel dos biomarcadores tem sido grandemente expandido para abranger todos os aspectos do cuidado ao paciente, desde o diagnóstico precoce do câncer até o estadiamento mais preciso do tumor e mesmo a seleção daqueles pacientes mais propensos a se beneficiar de terapias específicas, até o monitoramento tumoral pós-tratamento (KACZOR-URBANOWICZ et al., 2017).
As técnicas convencionais para purificação de proteínas são as cromatografias, tais como cromatografia de troca iônica (IEC), cromatografia de exclusão (SEC) e cromatografia de afinidade. Para análise de proteínas seletivas, podem ser utilizados ensaios imunoabsorventes ligados a enzimas (ELISA) e western blotting. De acordo com Aslam et al. (2017), as técnicas de eletroforese em gel de poliacrilamida com dodecil sulfato de sódio (SDS-PAGE), eletroforese em gel bidimensional (2D) e eletroforese em gel diferencial bidimensional (2D-DIGE) são usadas para separação de amostras de proteínas complexas.
2.4 Eletroforese em Gel SDS-PAGE e Espectrometria de Massas
Dentre as abordagens mais utilizadas de separação de proteínas estão as separações em gel, tanto unidimensional (SDS-PAGE) quanto bidimensional (2D). Como citado por Brizeno (2018), na separação bidimensional a primeira dimensão separa as proteínas de acordo com seus pontos isoelétricos (focalização isoelétrica); e na segunda dimensão as proteínas são separadas com base em seus pesos moleculares. Os géis podem ser corados de várias maneiras, sendo as mais comuns a coloração com azul de Coomassie ou a impregnação com prata. A quantificação pode ser feita através da medida de intensidade da mancha no gel com programas computacionais específicos.
A eletroforese em gel SDS-PAGE é amplamente reconhecida como uma ferramenta eficaz para a separação de proteínas com base em seu peso molecular. Esse método utiliza o detergente SDS (dodecil sulfato de sódio), que desnatura as proteínas e atribui a elas uma carga negativa uniforme, facilitando sua migração através de um gel de poliacrilamida quando submetidas a um campo elétrico (ASLAM et al., 2017).
A eletroforese é uma técnica capaz de identificar diferentes pesos moleculares, sendo frequentemente utilizada como ferramenta inicial de triagem para orientar métodos mais específicos. Assim, é viável empregar a eletroforese para analisar o perfil de expressão proteica na saliva, como demonstrado no estudo de Lima (2021), permitindo selecionar e ajustar a técnica mais apropriada conforme o proteoma identificado. No entanto, há técnicas específicas para a identificação de proteínas, como (ELISA), western blotting, cromatografias, espectrometria de massa, entre outras.
Por sua vez, a espectrometria de massas (MS) é uma técnica analítica sofisticada, capaz de identificar e caracterizar proteínas com elevada precisão, fornecendo dados detalhados sobre suas massas moleculares e modificações pós-traducionais (Aebersold & Mann, 2016). Essa técnica é particularmente valiosa na caracterização de proteínas envolvidas nos estágios iniciais de transformação neoplásica no epitélio oral (Mang et al., 2016). A integração da SDS-PAGE com a MS permite traçar um perfil proteômico mais completo, evidenciando alterações proteicas relacionadas ao desenvolvimento do câncer (LIMA,2021).
A aplicação combinada dessas metodologias se destaca pela complementaridade: Enquanto a SDS-PAGE realiza uma separação das proteínas presentes na amostra, numa triagem inicial a MS identifica e analisa essas moléculas de forma precisa e aprofundada. Esse conjunto de técnicas tem se mostrado indispensável em pesquisas proteômicas, especialmente para a descoberta de biomarcadores relacionados a doenças como o câncer oral (WANG et al., 2016).
Outra técnica que é amplamente utilizada em pesquisas, Western Blotting, que investiga a expressão de proteínas, alterações pós-traducionais e a validação de biomarcadores descobertos em estudos proteômicos. Sua alta especificidade, proporcionada pelo uso de anticorpos, aliada à sensibilidade, torna essa técnica uma ferramenta essencial em diversas áreas de estudo, especialmente na pesquisa de doenças como o câncer (Mahmood & Yang, 2012). Podendo ser utilizada associada com as técnicas de SDS-PAGE e MS.
3. METODOLOGIA
3.1. Metodologia de pesquisa da literatura
Trata-se de uma revisão de literatura, referente às produções científicas sobre trabalhos que utilizam as técnicas de SDS-PAGE e Espectrometria de massas para a identificação de biomarcadores de câncer oral.
Para tanto foram estabelecidos a seleção da temática, os critérios de inclusão e exclusão dos artigos para a pesquisa. Sendo realizada a análise das referências encontradas nas bases de dados Pubmed, Scopus, Spring Nature, Biblioteca Virtual em Saúde (BVS) e Google Acadêmico. Em todas as bases dados, foram usados os termos “Oral cancer”, “Oral squamous cells carcinoma”, “Mouth neoplasms”, “Head and neck cancer”, “proteins”, “Proteomic”, “SDS-PAGE”, “sodium dodecyl sulphate-polyacrylamide gel electrophoresis” e “Mass spectrometry”.
Os critérios de inclusão dos artigos foram:
- Artigos publicados no espaço cronológico de 2004 a 2024;
- Artigos que apresentassem as técnicas de SDS-PAGE e Espectrometria de massas aplicada ao câncer oral;
- Artigos em que as amostras utilizadas eram saliva e/ou tecido;
- Identificar proteínas para diagnóstico e/ou prognóstico; ● Lista das proteínas identificadas.
Os critérios de exclusão foram:
- Artigos científicos que não apresentavam arquivo completo ou que estão disponíveis apenas em resumos;
- Artigos que se repetiam nas bases de dados.
- Artigos que só apresentavam uma das técnicas;
- Artigos que não apontavam uma relação direta de dependência metodológica entre
SDS-PAGE e espectrometria de massas;
- Artigos que utilizaram amostras de soro e/ou plasma; ● Artigos sobre secretoma.
É importante destacar que adaptações específicas foram implementadas em cada base de dados, considerando as particularidades e funcionalidades dos filtros disponíveis, com o objetivo de assegurar uma seleção mais precisa dos artigos pertinentes ao estudo. Adicionalmente, o operador lógico ‘NOT’, ou equivalentes análogos, foi empregado para excluir artigos que, ao apresentarem em seus títulos termos alheios ao escopo da pesquisa, comprometeriam a relevância dos resultados obtidos (Quadro 1).
Quadro 1. Metodologia de pesquisa utilizada em cada base de dados
Base de dados Termos utilizados na barra de pesquisa Operador “NOT” ou similar Filtros Especiais Pubmed ((“Oral Cancer” OR “Oral Squamous Cells Carcinoma” OR “Mouth Neoplasms” OR “Head and neck cancer”) AND (proteins OR proteomic) AND (SDS-PAGE OR “sodium dodecyl sulphate-polyacrylamide gel electrophoresis”) AND “mass spectrometry”) Scopus (( “Oral Cancer” OR “Oral Squamous Cells Carcinoma” OR “Mouth Neoplasms” OR “Head and neck cancer” ) AND ( proteins OR proteomic ) AND ( sds-page OR “sodium dodecyl sulphate-polyacrylamide gel electrophoresis” ) AND “mass spectrometry” ) Spring Nature ((“Oral Cancer” OR “Oral Squamous Cells Carcinoma” OR “Mouth Neoplasms” OR “HEAD AND NECK CANCER”) AND (proteins OR proteomic) AND (SDS-PAGE OR “sodium dodecyl sulphate-polyacrylamide gel electrophoresis”) AND “mass spectrometry”) NOT review NOT canine NOT lung X Content Type (Article; Research article); Subjects (Oral cancer, Head and neck cancer, oral cancer detection, tumour biomarkers, oncogene proteins, Squamous cell carcinoma. Biblioteca Virtual em Saúde ((“Oral Cancer” OR “Oral Squamous Cells Carcinoma” OR “Mouth Neoplasms” OR “HEAD AND NECK CANCER”) AND (proteins OR proteomic) AND (SDS-PAGE OR “sodium dodecyl sulphate-polyacrylamide gel electrophoresis”) AND “mass spectrometry”) Google Acadêmico ((“Oral Cancer” OR “Oral Squamous Cells Carcinoma” OR “Mouth Neoplasms” OR “Head and neck cancer”) AND (proteins OR proteomic) AND (oral mucosa AND saliva) AND (SDS-PAGE AND “mass spectrometry”) -two-dimensional gel electrophoresis -canine -review -blood X
Fonte: Autores, 2024.
Este procedimento visou otimizar a recuperação de informações científicas alinhadas aos objetivos do trabalho por meio da redução do ruído informacional.
3.2. Etapas da triagem de artigos
Na etapa inicial, os artigos foram tabulados e seus títulos foram examinados para a identificação de duplicidades. Em seguida, procedeu-se à leitura preliminar dos artigos restantes com o objetivo de extrair informações como: doença abordada, tipo de amostra, aplicação de SDS-PAGE e Espectrometria de Massas. Posteriormente, foi realizada uma análise da seção ‘Metodologia’ para verificar a existência de uma relação metodológica direta entre SDS-PAGE e Espectrometria de Massas na identificação de proteínas.
Por fim, os artigos foram analisados em profundidade, considerando os objetivos propostos, os dados levantados e a presença de biomarcadores identificados, com a elaboração de uma lista enumerativa desses biomarcadores.
3.3. Identificação das proteínas no uniprot e organização dos dados
As proteínas mencionadas nos artigos foram catalogadas, sendo aquelas que possuíam identificadores de entrada prontamente tabuladas em planilhas eletrônicas no programa Excel. No caso de proteínas desprovidas de identificadores, procedeu-se a uma investigação de sua identidade e respectivo identificador, mediante pesquisa no banco de dados Universal Protein Resource (Uniprot).
Convém ressaltar que os dados foram estratificados em dois grupos: um grupo compreendia proteínas identificadas em artigos que utilizavam saliva como amostra, enquanto o outro consistia em proteínas identificadas em artigos que utilizavam tecido como amostra. Essa categorização visou facilitar a análise comparativa entre as diferentes origens amostrais no Excel.
3.4. Identificação de proteínas redundantes
Realizou-se uma análise intragrupo com o objetivo de identificar e eliminar redundâncias oriundas de proteínas repetidas, evitando a inclusão de resultados duplicados no estudo. Essa etapa foi crucial, considerando que a posterior comparação entre os conjuntos de proteínas salivares e teciduais pode ser comprometida pela presença de duplicidades, as quais tendem a amplificar desnecessariamente o número de interseções durante as análises cruzadas. A execução desse cruzamento intragrupo foi realizado utilizando o comando “=CONT.SE” no software Microsoft Excel. As duplicidades identificadas eram excluídas até que restasse somente a proteína de referência, que seguia para a etapa seguinte.
3.5. Comparação entre proteínas da mucosa oral e saliva
Procedeu-se à comparação intergrupal entre as proteínas identificadas na saliva e aquelas oriundas do tecido da mucosa oral, com o intuito de identificar proteínas compartilhadas entre os dois grupos. Esse cruzamento foi igualmente realizado utilizando o comando “=CONT.SE” no Excel, fundamentando-se nos dados obtidos a partir de artigos científicos que descreveram a identificação proteica por meio de saliva e tecido.
4. RESULTADOS
4.1. Artigos selecionados
A busca por estudos relevantes realizada nas bases de dados científicas, foram: PubMed (21 artigos), Biblioteca Virtual em Saúde (21 artigos), Google Acadêmico 20 artigos), Springer Nature (12 artigos) e Scopus (10 artigos). Essa busca inicial resultou em um total de 84 artigos potencialmente elegíveis entre os anos de 2004-2024. No entanto, após a aplicação dos critérios de inclusão e exclusão, apenas oito estudos atenderam a todos os requisitos para serem incluídos nesta revisão (Tabela 1).
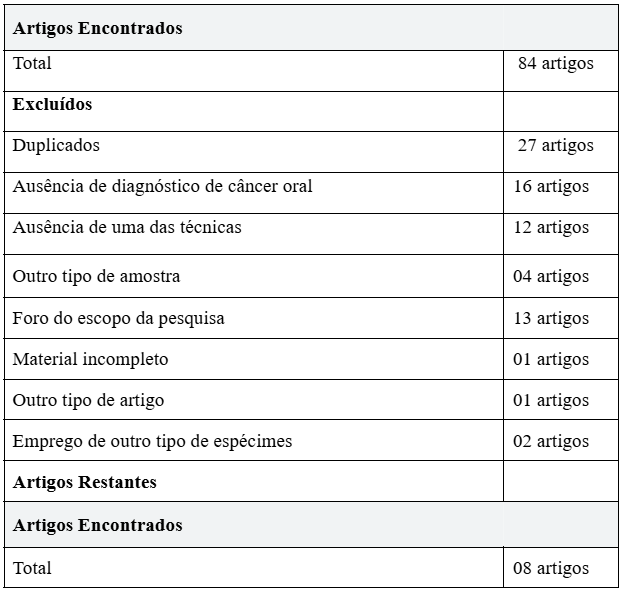
Fonte: Autores, 2024.
A análise dos oito estudos incluídos nesta revisão revelou uma divisão equilibrada entre aqueles que utilizaram a saliva (n=4) e aqueles que empregaram tecido ou culturas celulares derivadas de tecido de câncer oral (n=4) como Dfontes para a identificação de biomarcadores. A análise conjunta desses estudos permitiu a identificação de um total de 404 proteínas distribuídas entre os 08 artigos. A tabela 2 aponta a quantidade de proteínas identificadas por artigo.
Tabela 2: Características dos artigos selecionados.
Título Amostra Objetivos Marcadores Saliva proteome profiling reveals potential salivary biomarkers for detection of oral cavity squamous cell carcinoma. Saliva Descobrir biomarcadores salivares de CEC por meio do perfil comparativo dos proteomas da saliva dos voluntários saudáveis, dos indivíduos OPMD e dos pacientes com CEC usando SDS-PAGE acoplado a LC-MS/MS. 33 proteínas High-throughput screening of saliva for early detection of oral cancer: a pilot study Saliva Desenvolver um método amplamente disponível, barato, não invasivo e de alto rendimento, adequado para rastrear populações de alto risco, pelo menos, para sinais precoces de transformação maligna na cavidade oral. 09 proteínas Mass spectrometry-based salivary proteomics for the discovery of head and neck squamous cell carcinoma. Saliva Desenvolver um método espectrométrico de massa eficiente, rápido e sensível para a detecção de proteínas expressas diferencialmente como biomarcadores específicos de tumor na saliva de pacientes com CECP. 10 proteínas Pre-analytic saliva processing affect proteomic results and biomarker screening of head and neck squamous carcinoma. Saliva Avaliar o efeito do processamento pré-analítico na análise proteômica da saliva e identificar biomarcadores salivares para potenciais aplicações clínicas. 31 proteínas Proteomic approaches identify members of cofilin pathway involved in oral tumorigenesis. Tecido Enriquecer a caracterização molecular de subgrupos de CEC com diferentes prognósticos, especialmente aqueles com risco de disseminação tumoral. 213 proteínas Enrichment and nlc-ms/ms analysis of head and neck cancer mucinome glycoproteins. Cultura de células neoplásicas O objetivo deste estudo foi pesquisar o espaço proteômico de proteínas contendo domínio de mucina no câncer de células escamosas de cabeça e pescoço. 16 proteínas Nuclear heterogeneous nuclear ribonucleoprotein d is associated with poor prognosis and interactome analysis reveals its novel binding partners in oral cancer. Cultura de células neoplásicas Determinar as redes de proteínas associadas ao hnRNPD e seu significado clínico no carcinoma espinocelular oral (CEC). 90 proteínas Extracellular heat shock protein a9 is a novel interaction partner of podoplanin in oral squamous cell carcinoma cells. Cultura de células neoplásicas Procurar moléculas que interagem com PDPN por meio de análise baseada em proteômica e confirmar a interação ECM usando o mesmo sistema de células CEC oral que foi usado em nosso estudo recente 02 proteínas
Ao se avaliar a proporção de proteínas identificadas nos diferentes tipos de amostras, observou-se um predomínio de proteínas em experimentos realizados com tecido em comparação com saliva. A análise da literatura revelou a identificação de 83 proteínas em estudos envolvendo saliva, enquanto em pesquisas com tecido e/ou culturas celulares, esse número ascendeu a 321 proteínas. Nesse sentido, as proteínas de tecido/cultura representam 80% do total de proteínas identificadas nos estudos analisados.
4.2. Proteínas redundantes
Vale salientar que das 83 proteínas encontradas em artigos de saliva, 02 eram proteínas repetidas. Enquanto que das 321 proteínas de tecido/cultura, 17 eram repetidas. Isso se deve ao fato de que mais de um artigo daquele grupo amostral, seja saliva ou tecido/cultura, apresentavam as mesmas proteínas.
Nas proteínas de saliva, a proteína Annexin A1 (ANXA1_HUMAN) estava presente em 3 três artigos e a proteína Keratin, type II cytoskeletal 1 (K2C1_HUMAN) estava presente em 2 artigos (Tabela 3).
Nas proteínas de tecido/cultura, as proteínas Immunoglobulin heavy constant alpha 1 (IGHA1_HUMAN), Histone H2A type 1-B/E (H2A1B_HUMAN), Protein S100-A9 (S10A9_HUMAN), Heterogeneous nuclear ribonucleoproteins C1/C2 (HNRPC_HUMAN), Heterogeneous nuclear ribonucleoprotein A1 (ROA1_HUMAN), Lysosome-associated membrane glycoprotein 2 (LAMP2_HUMAN), Histone H1.5 (H15_HUMAN), Histone H1.2 (H12_HUMAN), Heterogeneous nuclear ribonucleoproteins A2/B1 (ROA2_HUMAN), Histone H2B type 2-F (H2B2F_HUMAN), Heterogeneous nuclear ribonucleoprotein K (HNRPK_HUMAN), Large ribosomal subunit protein uL23 (RL23A_HUMAN), Histone H4 (H4_HUMAN), Small ribosomal subunit protein RACK1 (RACK1_HUMAN), Heterogeneous nuclear ribonucleoprotein U (HNRPU_HUMAN), Histone H1.10 (H1X_HUMAN) e SWI/SNF-related matrix-associated actin-dependent regulator of chromatin subfamily D member 2 (SMRD2_HUMAN) estavam presentes em dois artigos cada (Tabela 3).
Tabela 3: Distribuição das proteínas redundantes.
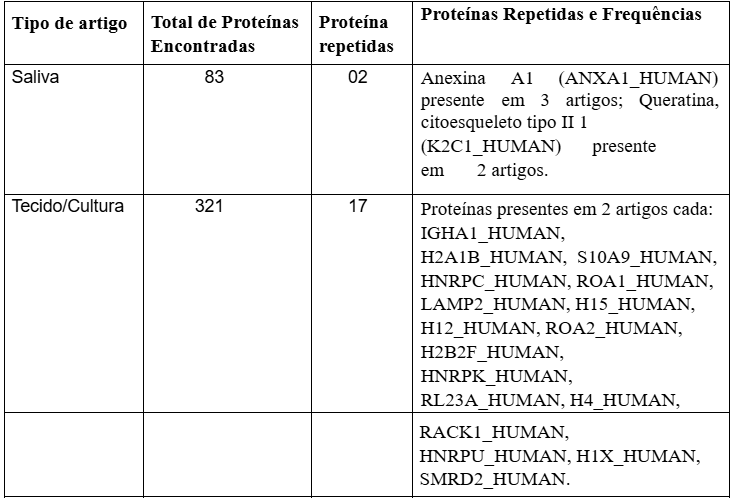
Fonte: Autores, 2024.
Dessa forma, o quantitativo final de proteínas identificadas, após a exclusão de duplicidades, foi de 81 proteínas em amostras de saliva e 304 proteínas em amostras de tecido ou cultura, resultando em um total consolidado de 385 proteínas.
4.3. Proteínas compartilhadas
Identificou-se um total de 24 proteínas comuns às amostras de saliva e às de tecido ou cultura de câncer oral. Essas proteínas correspondem a 42,85% do total de proteínas detectadas em tecido ou cultura e a 8,6% do total de proteínas identificadas na saliva. A maior prevalência de proteínas exclusivas no tecido sugere diferenças fundamentais na expressão proteica entre as amostras.
Figura 1. Diagrama de Venn – Quantidade de proteínas compartilhadas (na interseção) entre saliva e tecido/cultura de câncer oral.
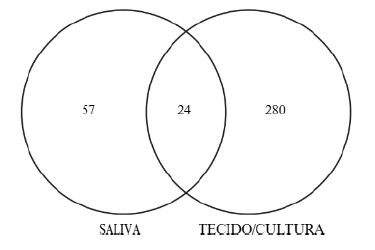
Imagem: Autores, 2024
As proteínas compartilhadas entre as duas amostras foram: Annexin A1 (ANXA1_HUMAN), Keratin, type II cytoskeletal 1 (K2C1_HUMAN), Tubulin alpha-1C chain (TBA1C_HUMAN), Histone H2A type 1-A (H2A1A_HUMAN), Annexin A2 (ANXA2_HUMAN), Peroxiredoxin-1 (PRDX1_HUMAN), Peroxiredoxin-2 (PRDX2_HUMAN), Albumin (ALBU_HUMAN), Keratin, type II cytoskeletal 4 (K2C4_HUMAN), Keratin, type I cytoskeletal 13 (K1C13_HUMAN), Collagen alpha-3(VI) chain (CO6A3_HUMAN), Fructose-bisphosphate aldolase A (ALDOA_HUMAN), Alpha-enolase (ENOA_HUMAN), Actin, cytoplasmic 1 (ACTB_HUMAN), Glucose-6-phosphate isomerase (G6PI_HUMAN), Heat shock cognate 71 kDa protein (HSP7C_HUMAN), Hemopexin (HEMO_HUMAN), Keratin, type I cytoskeletal 10 (K1C10_HUMAN), Neutrophil gelatinase-associated lipocalin (NGAL_HUMAN), Peptidyl-prolyl cis-trans isomerase A (PPIA_HUMAN), Apolipoprotein A-I (APOA1_HUMAN), Protein S100-A8 (S10A8_HUMAN), Protein S100-A9 (S10A9_HUMAN) e Transaldolase (TALDO_HUMAN).
5. DISCUSSÃO
A identificação de biomarcadores em fluidos biológicos consolidou-se como uma das principais áreas de pesquisas neste campo, principalmente devido à facilidade de coleta e ao menor impacto no conforto dos pacientes. Ressalta-se que essa abordagem é viabilizada pela presença de biomarcadores detectáveis em diferentes matrizes biológicas, incluindo tecidos e fluidos, como a saliva. Nesta investigação, foram identificadas 24 proteínas presentes tanto na saliva quanto em tecidos ou culturas celulares de pacientes diagnosticados com câncer de boca, por meio da análise comparativa das proteínas detectadas em ambas as amostras.
Uma observação de significativa relevância foi a identificação de 23 proteínas compartilhadas entre os estudos que analisaram saliva e o único estudo que empregou tecido excisado (Polachini et al., 2012). Em contraposição, apenas uma proteína foi detectada simultaneamente na saliva e nas culturas de células neoplásicas derivadas de carcinoma oral (Wu et al., 2015; Kumar et al., 2015). A divergência entre os perfis proteômicos de células cultivadas in vitro e tecidos frescos in vivo têm sido amplamente discutida na literatura ao longo dos anos. Evidências experimentais sugerem que essa discrepância decorre de diferenças nas demandas metabólicas e biológicas impostas às células em cultura. Nesse contexto, a abundância de nutrientes disponíveis in vitro induz uma priorização de recursos para vias relacionadas à proliferação celular e sinalização, frequentemente em detrimento de funções especializadas características do tecido de origem (Pan et al., 2008).
Estudos têm demonstrado variações significativas na expressão gênica entre linhagens celulares e seus respectivos tecidos de origem. De forma notável, genes com regulação positiva em linhagens celulares frequentemente exibem regulação negativa em tecidos, e o inverso também é observado. Genes cuja expressão está aumentada em linhagens celulares estão predominantemente associados a processos como o ciclo celular, metabolismo energético, biossíntese e processamento de biomoléculas. Em contrapartida, nos tecidos, os genes regulados positivamente estão majoritariamente implicados em funções relacionadas à adesão celular, sinalização mediada por receptores proteicos e comunicação intercelular, refletindo a complexidade e especialização do ambiente tecidual (Sandberg et al., 2005).
É importante destacar que nem todas as 24 proteínas identificadas foram detalhadamente discutidas nos estudos em que apresentaram alterações. Entre as proteínas compartilhadas, apenas Annexin A1, Keratin 1, Tubulin alpha-1C chain, Histone H2A type 1-A, Peroxiredoxin-1, Peroxiredoxin-2, Keratin 4, Hemopexin e Keratin 10 receberam análise mais aprofundada, devido a suas alterações expressivas em pacientes quando comparados aos controles (Wu et al., 2015; Szanto et al., 2012; Jarai et al., 2012; Oshiro et al., 2007; Kumar et al., 2025; Polachini et al., 2012). As demais proteínas foram apenas mencionadas como integrantes de um conjunto mais amplo de proteínas alteradas, sem discussões detalhadas ou específicas sobre suas funções ou relevância nos estudos.
Em relação à Anexina A1 (ANXA1), os dados apresentados por Szanto et al. (2012) indicaram superexpressão da proteína em cinco amostras de saliva de pacientes com câncer, enquanto esteve ausente em todos os controles. Por sua vez, Oshiro et al. (2007) relataram a presença de ANXA1 em todas as amostras de pacientes, embora também fosse detectada em amostras de indivíduos saudáveis, porém em menor frequência. Corroborando esses achados, Jarai et al. (2012) observaram superexpressão da proteína na maioria das amostras salivares de pacientes. Em tecidos, ANXA1 foi predominantemente associada a tumores menos agressivos, com redução de sua expressão em tumores de maior agressividade (Polachini et al., 2012). Atualmente, reconhece-se que a Anexina A1 desempenha papeis cruciais na regulação de processos inflamatórios e imunológicos, além de estar envolvida na proliferação celular, invasão e metástase quando desregulada, o que a torna um biomarcador relevante em diversos tipos de câncer (Rahimy et al., 2022).
Na presente pesquisa, foram encontradas três tipos de queratinas: Keratin 1 (KRT1), Keratin 4 (KRT4) e Keratin 10 (KRT10). Fazendo parte da família das queratinas, a Keratin 1 é uma proteína estrutural essencial para a integridade do epitélio, apresenta mudanças em sua expressão que estão ligadas à diferenciação celular e ao comportamento invasivo em carcinomas de células escamosas (RAHIMY, et al.2022). A proteína foi relatada por Ohshiro et. al (2007) e foi identificada em todas as amostras de pacientes com câncer, e só estava presente em 1 ou 2 amostras saudáveis. Entretanto, no tecido, sua expressão diminui conforme o grau de agressividade do tumor aumenta, onde foi observado que margens cirúrgicas e tumores menos agressivos apresentaram maior expressão da proteína, se comparado a tumores mais agressivos (POLACHINI, et al. 2012). A Keratin 4, por sua vez, está ligada à estabilidade estrutural de células epiteliais não queratinizadas e também desempenha um papel na diferenciação celular em tumores (ZIMTA, et al. 2019). A supracitada proteína foi classificada como potencial marcador tumoral por Jarai et. al (2012), uma vez que foi identificada em mais de 85% de amostras de saliva de pacientes com câncer oral. No tecido, por sua vez, foi apontada a ausência ou subexpressão da proteína em tumores por Polachini, et al (2012). Os autores ainda destacam que pacientes que apresentavam expressão negativa da dita proteína possuíam maiores taxas de apresentarem recorrência local e metástase. Além disso, se comparado às outras queratinas identificadas no estudo, a keratin 4 era a que apresentava menor expressão em tecidos tumorais se comparado ao tecido de margem cirúrgica. Quanto a Keratin 10, também estava presente em todas as amostras de saliva maligna (OHSHIRO et. al. 2007), entretanto seus dados com relação ao tecido se expressaram diferentemente das outras queratinas, uma vez que houve um aumento da sua expressão nos tumores se comparado a margem cirúrgica, que era livre de tumor (POLACHINI, et al. 2012).
A Tubulina alfa-1C (TUBA1C) é um componente essencial dos microtúbulos, desempenhando um papel crítico no processo de divisão celular. Sua superexpressão tem sido correlacionada à progressão tumoral e à resistência a agentes quimioterápicos que afetam a dinâmica dos microtúbulos. No estudo de Wu et al. (2015), TUBA1C foi detectada exclusivamente na saliva de pacientes com câncer, estando ausente tanto nas amostras de indivíduos saudáveis quanto nas de pacientes com lesões orais potencialmente malignas. Em tecidos tumorais, sua expressão foi associada a tumores de maior agressividade (Polachini et al., 2012).
De forma semelhante, a Histona H2A tipo 1-A (H2A1A), responsável por compactar o DNA e regular a expressão gênica, sofre modificações que podem alterar a expressão de genes relacionados ao câncer e favorecer a progressão tumoral (BINAROVÁ; TUSZYNSKI, 2019). Na saliva, a proteína foi encontrada exclusivamente nas amostras de pacientes (WU et. al. 2015), além disso, como já relatado, foi a única proteína do artigo de cultura de tecido cruzado com saliva, e na análise das proteínas derivadas da cultura, ela se encontrava superexpressa, juntamente de sua classe (KUMAR, et al. 2015).
As Peroxirredoxinas 1 e 2 (PRDX1 e PRDX2) são proteínas antioxidantes que desempenham um papel crucial na proteção celular contra danos induzidos por espécies reativas de oxigênio. No estudo conduzido por Szanto et al. (2012), ambas as proteínas foram detectadas em todas as amostras salivares de pacientes com câncer, enquanto estiveram ausentes em todas as amostras de indivíduos saudáveis. Contudo, em tecidos, PRDX1 e PRDX2 apresentaram um padrão de subexpressão, com níveis de expressão inversamente proporcionais ao grau de agressividade tumoral, conforme relatado por Polachini et al. (2012).
A Hemopexina (HPX) é uma proteína responsável pela regulação dos níveis de heme livre no plasma, desempenhando um papel essencial na modulação do ambiente oxidativo tumoral, com potencial para influenciar processos como proliferação e sobrevivência celular (Chen et al., 2019). No estudo de Oshiro et al. (2007), HPX foi detectada em quatro das cinco amostras salivares de pacientes com câncer, enquanto esteve ausente em todas as amostras de indivíduos saudáveis. Em contrapartida, na análise de tecidos, não foram observadas variações significativas na expressão de HPX entre os tecidos tumorais e aqueles livres de tumor (Polachini et al., 2012). Esses resultados sugerem que a detecção de HPX pode ser mais relevante em fluidos biológicos, como a saliva, do que em tecidos sólidos.
Juntas, essas proteínas não apenas impactam diretamente o desenvolvimento e a progressão do câncer, mas também podem interferir na resposta dos tumores aos tratamentos disponíveis.
6 CONSIDERAÇÕES FINAIS
A pesquisa alcançou os objetivos propostos ao identificar e comparar proteínas expressas na mucosa oral e saliva de pacientes com câncer oral, destacando seu provável potencial como biomarcadores para diagnóstico precoce. A utilização combinada das técnicas de SDS-PAGE e espectrometria de massas confirma a eficácia dessas técnicas na identificação das proteínas compartilhadas entre as amostras analisadas.
Com isso, foi possível constatar que o problema inicial proposto, foi respondido revelando diferenças fundamentais no perfil proteico entre saliva e tecido. Constatou-se que a saliva, apesar de apresentar menor diversidade proteica, mostra-se uma alternativa viável e não invasiva para monitoramento clínico. A metodologia utilizada, baseada na análise da literatura existente, foi adequada para os fins deste estudo.
A pesquisa contribui para o avanço da identificação de biomarcadores menos invasivos, apontando novos caminhos para o aprimoramento das práticas diagnósticas, podendo diminuir o número de biópsias e com isso aumentar a sobrevida do paciente.
No entanto, limitações relacionadas à padronização metodológica e à heterogeneidade das amostras destacam a necessidade de estudos futuros que explorem abordagens de enriquecimento proteico e ferramentas analíticas avançadas para aprimorar a sensibilidade dos testes. Bem como, o uso da bioinformática com papel essencial no avanço dessas abordagens, o que permite a análise integrada de grandes volumes de dados proteômicos. Visto que a combinação de bioinformática e tecnologias avançadas pode representar um salto significativo na pesquisa e no manejo clínico de doenças complexas como o câncer.
REFERÊNCIAS
AEBERSOLD, Ruedi; MANN, Matthias. Mass-spectrometric exploration of proteome structure and function. Nature, v. 537, n. 7620, p. 347-355, 2016.
AFFONSO, Vivian Regina et al. Infiltrado peritumoral no prognóstico do carcinoma epidermóide da cavidade oral. Revista Brasileira de Otorrinolaringologia , v. 81, n. 4, p. 416-421, 2015.
ANDRADE, M. A. C. Prevalência de lesões malignas e potencialmente malignas do complexo buco-maxilo-facial em pacientes de Odontoclínica Central da Marinha do Brasil. Faculdade de Odontologia de Piracicaba-Universidade Estadual de Campinas, Piracicaba, SP, Brasil, 2015.
ARDUINO, Paolo G. et al. Outcome of oral dysplasia: a retrospective hospital‐based study of 207 patients with a long follow‐up. Journal of oral pathology & medicine, v. 38, n. 6, p. 540-544, 2009.
ARELLANO‐GARCIA, M. E. et al. Multiplexed immunobead‐based assay for detection of oral cancer protein biomarkers in saliva. Oral diseases, v. 14, n. 8, p. 705-712, 2008.ASLAM, Bilal et al. Proteômica: Tecnologias e suas aplicações. Journal of Chromatographic Science , v. 55, n. 2, 2017.
BARBOSA, Eduardo Buzolin et al. Proteômica: metodologias e aplicações no estudo de doenças humanas. Revista da Associação Médica Brasileira, v. 58, p. 366-375, 2012.
BINAROVÁ, Pavla; TUSZYNSKI, Jack. Tubulin: structure, functions and roles in disease. Cells, v. 8, n. 10, p. 1294, 2019.
BISCH, Paulo Mascarello. Genômica funcional: proteômica. Genômica. São Paulo: Atheneu, p. 139I162, 2004.
BRIZENO, Luiz André Cavalcante. Análise proteômica de saliva de pacientes com lesões potencialmente malignas e carcinoma de células escamosas intraorais. 2018.
CASTRO, Ana Julia Porto de; LIMA, Julia da Costa. Lesões orais potencialmente cancerizáveis: revisão de literatura. 2020.
CHIAPPIN, Silvia et al. Saliva specimen: a new laboratory tool for diagnostic and basic investigation. Clinica chimica acta, v. 383, n. 1-2, p. 30-40, 2007.
COX, Jürgen; MANN, Matthias. Is proteomics the new genomics?. Cell, v. 130, n. 3, p. 395-398, 2007.
CHANDLER, Kevin Brown et al. Enrichment and nLC-MS/MS Analysis of Head and Neck Cancer Mucinome Glycoproteins. Journal of Proteome Research, v. 22, n. 4, p. 1231-1244, 2023.
CHEN, Ying et al. “Iron metabolism and its contribution to cancer (Review).” International journal of oncology vol. 54,4 (2019): 1143-1154. doi:10.3892/ijo.2019.4720
DAWES, C. Salivary flow patterns and the health of hard and soft oral tissues. The Journal of the American Dental Association, Chicago, v. 139, p. 18S-24S, 2008.
DE AQUINO, José Milton et al. A saliva como sendo um meio de diagnósticos: uma revisão de literatura. Revista Eletrônica Acervo Saúde, n. 41, p. e2506-e2506, 2020.
FÁBIÁN, T. K.; FEJÉRDY, P.; CSERMELY, P. Salivary genomics, transcriptomics and proteomics: the emerging concept of the oral ecosystem and their use in the early diagnosis of cancer and other diseases. Current genomics, v. 9, n. 1, p. 11-21, 2008.
FARINHA, F. I. A saliva como meio de diagnóstico. 2015. 87 f. Dissertação (Mestrado em Medicina Dentária)- Instituto Superior de Ciências da Saúde Egas Moniz. http://hdl.handle.net/10400.26/11761
GARFIN, D.E. Two-dimensional gel electrophoresis: an overview. Trends in Analytical Chemistry, v. 22, n. 5, p. 263-272, 2023.
HA, Patrick K. et al. Técnicas moleculares e alterações genéticas no câncer de cabeça e pescoço. Oncologia oral , v. 45, n. 4-5, p. 335-339, 2009.
HARDT, Markus et al. Avaliando os efeitos da variação diurna na composição da saliva da parótida humana: análise quantitativa de peptídeos nativos usando reagentes iTRAQ. Analytical Chemistry , v. 77, n. 15, p. 4947-4954, 2005.
HASSAN, Mohamed Ali Abol et al. Lectinas de cogumelo: especificidade, estrutura e bioatividade relevantes para doenças humanas. International journal of molecular sciences , v. 16, n. 4, p. 7802-7838, 2015.
HU, S.; LOO, J. A.; WONG, D. T. Human body fluid proteome analysis. Proteomics, Weinheim, v. 6, n. 23, p. 6326-6353, 2006.
HU, Shen et al. Large‐scale identification of proteins in human salivary proteome by liquid chromatography/mass spectrometry and two‐dimensional gel electrophoresis‐mass spectrometry. Proteomics, v. 5, n. 6, p. 1714-1728, 2005.
INSTITUTO NACIONAL DE CÂNCER (INCA). Tipos de câncer de boca. 2022. Disponível em: https://www.inca.gov.br/tipos-de-cancer/cancer-de-boca. Acesso em: 19 dez. 2024
JARAI, Tamas et al. Mass spectrometry-based salivary proteomics for the discovery of head and neck squamous cell carcinoma. Pathology & Oncology Research, v. 18, p. 623-628, 2012.
JESSIE, Kala et al. Aberrant proteins in the saliva of patients with oral squamous cell carcinoma. Electrophoresis, v. 34, n. 17, p. 2495-2502, 2013.
JOHN, Maie AR St et al. Interleukin 6 and interleukin 8 as potential biomarkers for oral cavity and oropharyngeal squamous cell carcinoma. Archives of Otolaryngology–Head & Neck Surgery, v. 130, n. 8, p. 929-935, 2004.
KACZOR-URBANOWICZ, Karolina Elżbieta et al. Saliva diagnostics–Current views and directions. Experimental Biology and Medicine, v. 242, n. 5, p. 459-472, 2017.
KUMAR, Manish et al. Nuclear heterogeneous nuclear ribonucleoprotein D is associated with poor prognosis and interactome analysis reveals its novel binding partners in oral cancer. Journal of translational medicine, v. 13, p. 1-15, 2015.
KRIEF, Guy et al. Comparison of diverse affinity based high-abundance protein depletion strategies for improved bio-marker discovery in oral fluids. Journal of proteomics, v. 75, n. 13, p. 4165-4175, 2012.
LEE, Yu-Hsiang; WONG, David T. Saliva: an emerging biofluid for early detection of diseases. American journal of dentistry, v. 22, n. 4, p. 241, 2009.
LIMA, Tayná Ferreira de. Perfil de proteínas expressas em amostras de saliva de indivíduos com câncer de boca através da técnica de SDS-PAGE eletroforese. 2021.
MAHMOOD, Tahrin; YANG, Ping-Chang. Western blot: técnica, teoria e resolução de problemas. Revista norte-americana de ciências médicas , v. 4, n. 9, p. 429, 2012.
NASCIMENTO, Kyria Santiago et al. Purification of a thermostable antinociceptive lectin isolated from Andira anthelmia. Journal of Molecular Recognition, v. 29, n. 6, p. 248-252, 2016.
NEVILLE, Brad. Patologia Oral e Maxilofacial. Elsevier. Brasil, 2011.
NOGUEIRA JÚNIOR, Jocélio Augusto. Análise do impacto de uma ação extensionista sobre a frequência de desordens orais malignas e potencialmente malignas em um Serviço de Estomatologia do Rio Grande do Norte. 2022. Trabalho de Conclusão de Curso. Universidade Federal do Rio Grande do Norte.
OHSHIRO, Kazufumi et al. Pre-analytic saliva processing affect proteomic results and biomarker screening of head and neck squamous carcinoma. International journal of oncology, v. 30, n. 3, p. 743-749, 2007.
PAN, Cuiping et al. Comparative proteomic phenotyping of cell lines and primary cells to assess preservation of cell type-specific functions. Molecular & Cellular Proteomics, v. 8, n. 3, p. 443-450, 2009.
PETTI, S. Lifestyle risk factors for oral cancer. Oral oncology, Oxford, v. 45, n. 4, p. 340- 350, 2009.
POLACHINI, Giovana M. et al. Proteomic approaches identify members of cofilin pathway involved in oral tumorigenesis. PloS one, v. 7, n. 12, p. e50517, 2012.
RAHIMY, Rifkath Marie Laurence et al. Rastreamento de moléculas diferencialmente expressas em neoplasias mieloproliferativas. 2022. Tese de Doutorado.
RIVERSOS, A.C.G. Análise proteômica do saco vitelino de bovinos. 2009. 111 f. Dissertação de Mestrado. Universidade de São Paulo.
ROSA, Nuno et al. Do proteoma salivar ao oraloma: Biologia molecular oral abrangente. Revista Arquivos de biologia oral, 7 ed.,vol. 57, p 853-864. Universidade Católica Portuguesa, 2012.
SANDBERG, Rickard; ERNBERG, Ingemar. The molecular portrait of in vitro growth by meta-analysis of gene-expression profiles. Genome biology, v. 6, p. 1-15, 2005.
SASSI, Laurindo Moacir et al. Carcinoma espinocelular de boca em paciente jovem: relato de caso e avaliação dos fatores de risco. RSBO, v. 7, n. 1, p. 105-9, 2010.
SHARON, Nathan; LIS, Halina. History of lectins: from hemagglutinins to biological recognition molecules. Glycobiology, v. 14, n. 11, p. 53R-62R, 2004.
SYRJÄNEN, S. et al. Human papillomaviruses in oral carcinoma and oral potentially malignant disorders: a systematic review. Oral diseases, v. 17, p. 58-72, 2011.
SZANTO, I. et al. High-throughput screening of saliva for early detection of oral cancer: a pilot study. Technology in cancer research & treatment, v. 11, n. 2, p. 181-188, 2012.
TSUNEKI, Masayuki et al. Extracellular heat shock protein A9 is a novel interaction partner of podoplanin in oral squamous cell carcinoma cells. Biochemical and biophysical research communications, v. 434, n. 1, p. 124-130, 2013.
WANG, Kevin et al. Identification of novel O-linked glycosylated Toxoplasma proteins by Vicia villosa lectin chromatography. PloS one, v. 11, n. 3, p. e0150561, 2016.
WARNAKULASURIYA, S.; ARIYAWARDANA, A. Malignant transformation of oral leukoplakia: a systematic review of observational studies. Journal of Oral Pathology & Medicine, Copenhagen, v. 45, n. 3, p. 155-166, 2016.
WU, Chih‐Ching et al. Saliva proteome profiling reveals potential salivary biomarkers for detection of oral cavity squamous cell carcinoma. Proteomics, v. 15, n. 19, p. 3394-3404, 2015.
WU, Jia-Yo et al. Potential biomarkers in saliva for oral squamous cell carcinoma. Oral oncology, v. 46, n. 4, p. 226-231, 2010.
ZHAO, Jia et al. N-linked glycosylation profiling of pancreatic cancer serum using capillary liquid phase separation coupled with mass spectrometric analysis. Journal of proteome research, v. 6, n. 3, p. 1126-1138, 2007.
ZIMTA, Alina-Andreea et al. The role of Nrf2 activity in cancer development and progression. Cancers, v. 11, n. 11, p. 1755, 2019.
ZORZETTO, Ricardo. No sangue, mais pistas sobre o câncer: Biópsia líquida começa a ser usada para avaliar a evolução de tumores e a necessidade de mudar o tratamento. Pesquisa Fapesp, p. 51-55, 2017.
1Graduanda do Curso de Farmácia pela Universidade Federal de Alagoas, Campus A. C. Simões. http://lattes.cnpq.br/3698234033227022
2Mestrando do Curso de Farmácia pela Universidade Federal de Alagoas, Campus A. C. Simões. http://lattes.cnpq.br/7030250280204326
3Graduando do Curso de Farmácia pela Universidade Federal de Alagoas, Campus A. C. Simões. http://lattes.cnpq.br/0640720694788276
4Graduanda do Curso de Farmácia pela Universidade Federal de Alagoas, Campus A. C. Simões. https://lattes.cnpq.br/2214274218287781
5Graduanda do Curso de Medicina pela AFYA, Campus Cruz das Almas. http://lattes.cnpq.br/0284684652776409
6Professora Adjunta da Universidade Estadual de Ciências da Saúde de Alagoas. http://lattes.cnpq.br/1520259176683500
7Professor Associado do Curso de Farmácia, do Instituto de Ciências Farmacêuticas da Universidade Federal de Alagoas, Campus A.C. Simões. http://lattes.cnpq.br/7010351122476225
