APPLICATIONS OF NEXT-GENERATION SEQUENCING TECHNOLOGY AND THE USE OF BIOINFORMATICS TOOLS FOR DATA ANALYSIS
REGISTRO DOI: 10.5281/zenodo.11519236
Isabella Sanchez Morim1
Michelle Buscarilli de Moraes2
RESUMO
Neste estudo, investigou-se as aplicações da tecnologia de sequenciamento de nova geração e o uso da ferramenta de bioinformática com a finalidade de atingir a contribuição da tecnologia de sequenciamento de nova geração para avanços na ciência e prática clínica, bem como a implementação de ferramentas de bioinformática para análise dos dados gerados pelo sequenciamento de nova geração. O método consiste em uma revisão narrativa da literatura, abrangendo o período de 2003 a 2024 e utilizando artigos científicos e teses. Os resultados evidenciam que o NGS permite sequenciar genomas inteiros ou áreas específicas, identificar variantes genéticas e realizar análises em larga escala. O fluxo de trabalho do NGS inclui etapas como extração de ácidos nucleicos, preparação de bibliotecas e sequenciamento. As principais plataformas de NGS, como Roche/454, Ion Torrent, illumina MiSeq, SOLiD, PacBio e Oxford Nanopore MinION, são discutidas em termos de princípios e aplicações. A análise bioinformática dos dados gerados pelo NGS envolve etapas como verificação de qualidade, alinhamento, chamada de variantes e anotação funcional. As aplicações do NGS incluem medicina de precisão, farmacogenômica, microbiologia, epigenética, metagenômica e estudos de associação genômica ampla. A integração da automação laboratorial com inteligência artificial promete expandir ainda mais as capacidades do NGS. Por fim, as considerações finais destacam a importância do NGS e da bioinformática, bem como os desafios a serem superados, como qualidade de dados e custos.
Descritores: sequenciamento de nova geração, análise bioinformática, genoma.
ABSTRACT
The article discusses the contribution of Next-Generation Sequencing (NGS) technology to advancements in science and clinical practice, as well as the implementation of bioinformatics tools for data interpretation. The method consists of a narrative review of the literature, covering the period from 2000 to 2023 and utilizing scientific articles and theses. The results demonstrate that NGS enables whole genome or specific area sequencing, identification of genetic variants, and large-scale analyses. The workflow of NGS includes steps such as nucleic acid extraction, library preparation, and sequencing. Major NGS platforms such as Roche/454, Ion Torrent, Illumina MiSeq, SOLiD, PacBio, and Oxford Nanopore MinION are discussed in terms of principles and applications. Bioinformatic analysis of NGS-generated data involves steps like quality checking, alignment, variant calling, and functional annotation. NGS applications encompass precision medicine, pharmacogenomics, microbiology, epigenetics, metagenomics, and genome-wide association studies. Integration of laboratory automation with artificial intelligence promises to further expand NGS capabilities. Finally, concluding remarks highlight the importance of NGS and bioinformatics, as well as challenges to overcome such as data quality and costs.
Keywords: Next-Generation Sequencing, bioinformatic analysis, genome.
INTRODUÇÃO
No século XIX, Gregor Johann Mendel, um monge e biólogo austríaco, contribuiu para a descoberta das bases da genética moderna através de seus experimentos com cruzamentos de ervilhas. As leis de Mendel, publicadas em 1866, estabeleceram os princípios básicos da hereditariedade, demonstrando que características hereditárias são transmitidas através de unidades básicas de herança, hoje conhecidas como genes (1).
Em 1953, James Watson e Francis Crick descobriram a estrutura tridimensional do DNA – a dupla hélice – que foi primeiramente desvendada pelos dados de difração de raio-x por Rosalind Franklin. Essa estrutura elucidou como os genes são transmitidos ao longo das gerações. Através desse marco, abriu-se caminho para novas descobertas na ciência, impulsionando a biologia molecular até os dias de hoje (1).
Uma das grandes contribuições para a área científica, foi o desenvolvimento da técnica de sequenciamento de DNA por Frederick Sanger e Alan Coulson no ano de 1975. Essa técnica possibilitou a determinação precisa da ordem na qual os nucleotídeos estão em uma dada molécula de DNA (ácido desoxirribonucleico), permitindo sequenciar pequenos fragmentos de ácidos nucleicos, após fragmentação química, física ou enzimática (1). O método de Sanger, também conhecido como didesoxi, constitui a base da primeira geração de sequenciadores de DNA. Este método utiliza a enzima DNA polimerase para sintetizar cadeias de DNA de diferentes comprimentos (2,3). Com a automação foi possível realizar grandes projetos de sequenciamento, como o Projeto Genoma Humano. O projeto, que utilizou a tecnologia de sequenciamento de Sanger (terminação de cadeia), levou 13 anos e custou US$3 bilhões e foi concluído em 2003. Porém, apesar do sucesso no sequenciamento do genoma humano completo, o sequenciamento de Sanger é um método caro e ineficiente para projetos de larga escala. Para tarefas como esta, as novas técnicas de NGS são mais eficientes e possuem menor custo (3).
O progresso tecnológico e outras demandas impulsionaram o desenvolvimento de novas metodologias de sequenciamento, caracterizadas por maior qualidade, custo reduzido, maior rapidez e maior capacidade de geração de dados. Esses novos métodos são classificados como Sequenciamento de Nova Geração, abrangendo a segunda, terceira e quarta gerações de tecnologias. Entre esses métodos, destacam-se o 454 Roche, Ion Torrent, iIIumina, SOLiD, PacBio e o Nanopore (3,4).
A tecnologia de sequenciamento de nova geração (NGS, do inglês Next-Generation Sequencing), também conhecida como sequenciamento massivo em paralelo, revolucionou a pesquisa genômica, permitindo sequenciar milhões de fragmentos simultaneamente em uma única corrida. O sequenciamento de nova geração, também oferece maior sensibilidade para detectar variantes novas ou raras, permite fluxos de trabalho de alto rendimento e grandes conjuntos de dados. Esta tecnologia tem aplicações em pesquisas científicas, permitindo novos conhecimentos sobre a base genética das doenças, processos biológicos complexos, medicina personalizada, microbiologia, farmacologia, relações evolutivas entre as espécies, entre outros campos (1,4,5). De acordo com a BCC Research, atualmente o mercado NGS está em expansão, estima-se que o mercado global para aplicações clínicas de sequenciamento de próxima geração aumente de US$ 21,9 bilhões em 2023 para atingir US$ 52,9 bilhões em 2028, a uma taxa composta de crescimento anual de 19,3% de 2023 a 2028 (6,7).
Uma realização recente de grande relevância científica foi o sequenciamento completo do genoma do vírus SARS-CoV-2 no Brasil em apenas 48 horas após a confirmação do primeiro caso de Covid-19 no país, utilizando-se a tecnologia de sequenciamento Oxford MinION Nanopore, uma plataforma de terceira geração. Este avanço foi liderado pela cientista e biomédica Jaqueline Goes de Jesus.
Sob esse aspecto, a capacidade de gerar quantidades tão vastas de dados genômicos traz consigo o desafio de processar e interpretar essas informações de forma eficiente e precisa. É aqui que entra a bioinformática. A bioinformática se desenvolveu principalmente depois do surgimento dos sequenciadores de DNA. O sequenciamento de nova geração gera grandes quantidades de dados genômicos que requerem pipelines de bioinformática sofisticados para análise e interpretação, e oferece um conjunto de ferramentas e metodologias para o processamento, armazenamento e análise de dados biológicos. As ferramentas de alinhamento de sequência, por exemplo, permitem o mapeamento eficiente de leituras de sequência a um genoma de referência, facilitando a identificação de variantes genéticas e a detecção de novos genes (1,4,5,6,7).
Neste contexto, este artigo tem como objetivo aprofundar as aplicações do sequenciamento de nova geração e o uso da ferramenta de bioinformática, abrangendo aspectos técnicos e perspectivas futuras.
OBJETIVO
Este artigo visa elucidar a contribuição da tecnologia de sequenciamento de nova geração para os avanços na ciência e para a prática clínica. Além disso, pretende-se destacar as aplicações do sequenciamento de nova geração e a implementação da ferramentas de bioinformática para a interpretação eficiente e precisa dos dados gerados.
JUSTIFICATIVA
O presente estudo foi escolhido em razão de um interesse acadêmico, motivado pela curiosidade em explorar as tecnologias de sequenciamento genético e a aplicação de ferramentas de bioinformática. Esta pesquisa busca contribuir significativamente para o entendimento dessas avançadas metodologias, que desempenham um papel importante no avanço de várias áreas da ciência e para a medicina personalizada, tornando os tratamentos mais efetivos. A escolha do tema reflete a relevância contemporânea e o potencial inovador do uso dessas tecnologias, oferecendo uma análise aprofundada que visa enriquecer o conhecimento na área.
MATERIAIS E MÉTODOS
Trata-se de um estudo de revisão narrativa da literatura, com o objetivo de avaliar o potencial e aplicações do sequenciamento de nova geração e o uso da ferramenta de análise de bioinformática. Esta revisão explora as diversas aplicações do NGS e da utilização de ferramentas computacionais para a análise de dados genéticos, destacando suas contribuições para a pesquisa científica e diagnóstico clínico.
Para realizar o levantamento bibliográfico do presente trabalho, foram utilizados artigos científicos, dissertações e teses, disponibilizadas online e selecionadas através dos principais bancos de periódicos: Scielo, PubMed e Google Acadêmico entre os anos 2000 e 2023.
CONSIDERAÇÕES GERAIS
Sequenciamento de Nova Geração
O NGS refere-se a um conjunto de tecnologias de sequenciamento de ácidos nucleicos em larga escala que permite sequenciar o genoma inteiro ou apenas áreas específicas de interesse, incluindo todos os aproximadamente 20.000 genes codificadores ou um pequeno número de genes individuais, além de identificar variantes genéticas em grande escala, análises de metagenoma e identificação de mutações somáticas no câncer, entre outros [9].
Essa técnica permite observar o sequenciamento de dezenas a centenas de milhares de leituras de fragmentos de DNA por amostra em uma única corrida. A tecnologia NGS pode ser caracterizada como um sequenciamento de DNA e RNA automatizado, paralelo e de alto rendimento [10].
De uma maneira geral, o sequenciamento de nova geração funciona fragmentando o DNA em pedaços menores de forma aleatória e depois usando enzimas para sintetizar fitas complementares de DNA. As cadeias de DNA são então sequenciadas e os dados resultantes são usados para reconstruir a sequência do DNA original [11].
Um dos principais diferenciais do sequenciamento de nova geração é a possibilidade de obter de reads de forma significativamente mais rápida. No entanto, o comprimento dos reads é reduzido, variando de algumas dezenas a poucas centenas de pares de bases, conforme a metodologia utilizada. O seu processamento é mais rápido e seu custo é reduzido em comparação com o sequenciamento de Sanger [10, 12,13].
Em princípio, os conceitos por trás da tecnologia Sanger e da tecnologia de sequenciamento de nova geração são semelhantes. No sequenciamento de nova geração e no sequenciamento Sanger, a DNA polimerase adiciona nucleotídeos fluorescentes um por um a uma fita modelo de DNA em crescimento. Cada nucleotídeo incorporado é identificado por sua etiqueta fluorescente [10, 12,13].
A principal diferença entre as duas tecnologias é o volume de sequenciamento. O método Sanger sequencia apenas um único fragmento de DNA por vez, enquanto o NGS é massivamente paralelo, o que significa que sequência milhões de fragmentos simultaneamente por corrida. O sequenciamento de nova geração também oferece recursos aprimorados de descoberta para detectar variantes novas ou raras por meio de sequenciamento por profundidade de ácidos nucleicos [13,14,15].
Fluxo de trabalho do sequenciamento de nova geração
Os principais processos envolvidos no sequenciamento de nova geração incluem, extração dos ácidos nucleicos, preparação de biblioteca, amplificação, sequenciamento e análise de dados. No contexto do sequenciamento de nova geração, uma biblioteca é definida como um conjunto de fragmentos de DNA ou RNA que representa o genoma ou transcriptoma completo, ou uma região alvo específica. Cada plataforma de NGS possui características próprias, mas, de maneira geral, a preparação de uma biblioteca NGS inicia-se com a fragmentação do material genético. Em seguida, adaptadores de sequência são ligados aos fragmentos para permitir o enriquecimento desses fragmentos [5, 15].
Sabe-se que uma biblioteca de alta qualidade deve ter grande sensibilidade e especificidade. Isso implica que todos os fragmentos de interesse devem estar igualmente representados na biblioteca. No entanto, alcançar essa qualidade é desafiador, pois as regiões genômicas variam em sua propensão a serem sequenciadas, o que dificulta a construção de uma biblioteca que seja simultaneamente sensível e específica [5, 15, 16].
O primeiro estágio na preparação de bibliotecas em grande parte dos processos de sequenciamento de nova geração consiste na fragmentação do ácido nucleico. Esse processo pode ser realizado por meio de métodos físicos ou enzimáticos. Entre os métodos físicos destacam-se a sonificação, nebulização, fragmentação mecânica e cisalhamento hidrodinâmico. Já os métodos enzimáticos incluem a fragmentação com transposases e DNase [5,17,18]. De acordo com uma pesquisa conduzida por um estudo comparativo sistemático entre métodos de fragmentação enzimática e física, observou-se que ambos apresentam rendimentos similares [5, 19].
Após a fragmentação do DNA inicial, procede-se à ligação de adaptadores aos fragmentos gerados. A inclusão desses adaptadores visa estabelecer pontos de início e término conhecidos para as sequências aleatórias, facilitando assim o subsequente processo de sequenciamento [5,20]. Posteriormente, logo após a fragmentação do ácido nucleico, é realizada a seleção dos fragmentos com base no tamanho da biblioteca desejada, sendo este parâmetro condicionado pelo tipo de equipamento de sequenciamento de nova geração utilizado e pela aplicação específica do sequenciamento. Um exemplo prático, os sequenciadores de leitura curta, como os sistemas illumina e Ion Torrent, apresentam um desempenho superior quando as bibliotecas de DNA consistem em fragmentos relativamente curtos e de tamanhos uniformes. Os fragmentos sequenciados por dispositivos illumina podem atingir até 1.500 pares de bases, enquanto os gerados por sistemas Ion Torrent alcançam até 400 pares de bases [5,18,21].
A etapa de enriquecimento, onde a quantidade de material alvo é aumentada em uma biblioteca a ser sequenciada. Quando apenas uma parte do genoma precisa ser investigada tanto para pesquisa quanto para aplicações clínicas, isso é conhecido como bibliotecas-alvo. Basicamente, dois métodos são comumente usados para tais abordagens direcionadas, o sequenciamento baseado em hibridização de captura e sequenciamento baseado em amplicon. No método de captura híbrida, na etapa de fragmentação, as moléculas fragmentadas são hibridizadas especificamente com fragmentos de DNA complementares às regiões alvo de interesse. O sequenciamento baseado em amplicon, a amplificação é realizada usando primers (oligonucleotídeos) específicos que se ligam às extremidades da região de interesse. Uma vez que a amplificação é completa, os produtos amplificados são sequenciados usando técnicas como sequenciamento de nova geração ou sequenciamento de terceira geração [5, 22, 23, 24]. Os métodos de amplicon possuem algumas limitações, como duplicatas e amplificação não uniforme. Por outro lado, a captura híbrida oferece cobertura e profundidade mais uniformes, apesar de ser mais cara e demorada [25, 26].
Principais plataformas de Sequenciamento de Nova Geração
As principais plataformas de sequenciamento de nova geração são desenvolvidas por diversas empresas e possuem diferentes características e aplicações para o sequenciamento em massa.
A plataforma Roche/454 (segunda geração) representou um avanço significativo no sequenciamento de DNA de alto rendimento, marcando o início da era do sequenciamento de nova geração. Este sistema pioneiro foi o primeiro de sua categoria a ser comercializado. Baseado no método de pirosequenciamento, ele detecta o pirofosfato liberado durante a incorporação de nucleotídeos na cadeia de DNA em síntese. Em uma única corrida, a plataforma era capaz de produzir aproximadamente 1 milhão de sequências. No entanto, esta técnica gradualmente perdeu relevância após 2013, com a introdução de tecnologias subsequentes de sequenciamento que ofereciam maior eficiência e capacidade de geração de dados [27].
A tecnologia de segunda geração Ion Torrent é uma plataforma de sequenciamento de leitura curta a média (200-600 bp) que utiliza semicondutores e se destaca pelo método de variação de pH como indicador. Este método, conhecido como pHmetro, baseia-se na alteração do pH devido à liberação de prótons durante a adição de nucleotídeos no processo de sequenciamento. A mensuração precisa dessas variações de pH permite inferir a sequência de nucleotídeos do DNA analisado. Durante a adição de cada nucleotídeo, uma molécula de H + é liberada, alterando o pH da solução. A detecção dessa variação de pH é realizada por um sensor de íons que registra as mudanças no ambiente químico. Uma vantagem distintiva desta técnica é a ausência de necessidade de nucleotídeos marcados, ao contrário dos métodos de Sanger e illumina. No entanto, este método pode enfrentar desafios relacionados a erros de inserção ou deleção (indels) de nucleotídeos em regiões de sequências repetitivas [28, 29, 30, 31].Uma das plataformas de sequenciamento de nova geração mais utilizadas globalmente é a illumina MiSeq (segunda geração), que vem ganhando mercado devido à sua capacidade de realizar grandes sequenciamentos com alta qualidade de reads a um custo mais acessível. O processo de sequenciamento illumina inicia-se com a preparação da biblioteca de DNA, onde o DNA alvo é fragmentado em pequenos pedaços e adaptadores específicos são ligados às extremidades desses fragmentos [28]. O processo de sequenciamento por illumina começa com a preparação da biblioteca de DNA. O DNA alvo é fragmentado em pequenos pedaços e adaptadores específicos são ligados às extremidades desses fragmentos [28, 32]. Esses adaptadores funcionam como pontos de ligação durante as etapas subsequentes do sequenciamento. Após a preparação da biblioteca, os fragmentos de DNA adaptados são amplificados clonalmente em uma superfície sólida, como uma lâmina ou flow cell [28, 32, 33]. Nesse ambiente controlado, os fragmentos de DNA se multiplicam formando clusters de amplicons, cada um contendo várias cópias do mesmo fragmento original de DNA. O sequenciamento por síntese ocorre nos clusters de DNA amplificados. Durante esse processo, cada base do DNA é adicionada sequencialmente em uma reação química controlada [33, 34]. Cada base é marcada com uma fluorescência distinta, permitindo a identificação da base adicionada em cada ciclo de síntese. Após a adição de cada base, uma imagem é capturada para registrar a fluorescência em cada cluster, determinando assim a sequência de nucleotídeos. Uma vez concluído o sequenciamento, os dados brutos são processados para identificar as bases presentes em cada cluster [33, 34]. Esse processo envolve a análise das intensidades de fluorescência para determinar a sequência de nucleotídeos em cada posição. Os dados de sequenciamento são então montados em sequências de DNA, que podem ser utilizadas em análises subsequentes, como montagem de genoma, identificação de variantes genéticas, análise de expressão gênica, entre outros. O sequenciamento de nova geração por illumina oferece várias vantagens, incluindo alta precisão e custo relativamente baixo por base sequenciada. Pode ser usado para sequenciar genomas completos, exomas, transcriptomas, epigenomas e outras aplicações, proporcionando uma ampla gama de possibilidades de pesquisa. Embora a precisão do sequenciamento illumina seja elevada, ainda podem ocorrer erros sistemáticos [28, 32, 33, 34].
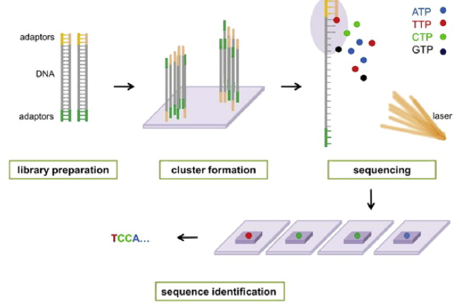
Figura 1. Princípio do sequenciamento por illumina [34].
A plataforma de sequenciamento SOLiD, ou sequenciamento por ligação e detecção de nucleotídeos, é uma tecnologia de segunda geração que utiliza uma enzima ligase. Diferente do sequenciamento por síntese (Illumina), que incorpora nucleotídeos fluorescentes, o SOLiD liga oligonucleotídeos fluorescentes específicos à sequência de DNA em cada etapa do ciclo de sequenciamento. A tecnologia SOLiD emprega amplificação em emulsão para gerar milhões de cópias de cada fragmento de DNA em compartimentos individuais, permitindo o sequenciamento em paralelo [35,36]. Embora essa plataforma tenha um alto rendimento, os curtos comprimentos de leitura tornam a montagem do genoma extremamente complexa. Portanto, é utilizada principalmente em estudos que exigem alta cobertura para análise de variantes e sequenciamento por profundidade [35,36].
O sequenciamento de terceira geração, conhecido como Pacific Biosciences (PacBio), é um método de sequenciamento de molécula única em tempo real. Esta tecnologia se destaca por gerar reads que podem ultrapassar 20 kb, superando as limitações de métodos tradicionais como o sequenciamento primeira geração e de segunda geração. Inicialmente, o DNA é fragmentado e as extremidades dos fragmentos são ligadas para formar uma estrutura circular, permitindo a leitura contínua de ambos os lados do fragmento em um único ciclo de sequenciamento. Adaptadores são incorporados entre as leituras para facilitar a identificação e montagem das sequências. O sequenciamento ocorre em nano-poços, cada um contendo uma molécula de DNA e uma enzima DNA polimerase. Bases são adicionadas ciclicamente, e a incorporação de uma base pela polimerase libera um fluoróforo, cujo sinal é detectado em tempo real [35]. Uma das maiores vantagens do PacBio é sua capacidade de gerar leituras de DNA muito longas, úteis para montar genomas complexos e identificar estruturas genômicas, como repetições e rearranjos [35,37,38]. No entanto, o custo por base do sequenciamento PacBio é geralmente mais alto do que outras tecnologias de sequenciamento, o que limita seu uso em alguns projetos de larga escala.
Outro sistema de sequenciamento que vem inovando o mercado é o Oxford Nanopore MinION (terceira geração), desenvolvido em 2013. Este dispositivo oferece um ótimo custo-benefício comparado a outros sequenciadores, além de ser rápido, portátil e fácil de usar. O fluxo de trabalho do MinION é distinto por não exigir a etapa intermediária de amplificação. As etapas envolvem três fases principais: preparação da biblioteca, sequenciamento e análise de dados. A tecnologia baseia-se na passagem da sequência de DNA através de um poro (α-hemolisina) em uma membrana artificial dentro de uma câmara de sequenciamento. Durante a passagem do DNA, um sinal elétrico é gerado, causando uma alteração de potencial elétrico detectada pelos sensores do aparelho, que identificam as bases nitrogenadas pela variação no potencial eletrostático [38]. As quatro bases do DNA – adenina (A), timina (T), citosina (C) e guanina (G) – apresentam diferentes conformações estruturais e dimensões moleculares, resultando em variações detectáveis na corrente elétrica. Essas variações são medidas por um dispositivo de chip eletrônico, e um algoritmo especializado processa os dados, convertendo-os em uma sequência de nucleotídeos [39]. O sequenciamento por nanoporos possui diversas aplicações, facilitando o estudo das modificações genéticas e epigenéticas e sua influência na expressão gênica [40]. Além disso, é eficaz na detecção de novos tipos de vírus e bactérias, proporcionando uma solução de alta capacidade de sequenciamento e uma plataforma portátil [39, 40, 41]. A NASA obteve sucesso ao usar a tecnologia Nanopore MinION no espaço, sequenciando material genético de microrganismos a bordo da Estação Espacial Internacional, demonstrando a viabilidade da técnica em condições de microgravidade [41, 42, 43]. Embora a tecnologia esteja em rápida evolução, os sequenciadores de nanoporos ainda podem ter limitações de throughput em comparação com outras plataformas, afetando a escalabilidade para projetos de alto volume.
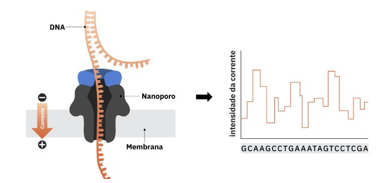
Figura 2. Princípio do sequenciamento por nanoporos [44].
Análise bioinformática dos dados gerados por sequenciamento de nova geração
Depois de concluir o sequenciamento, os dados gerados precisam ser processados e analisados para obter informações úteis sobre o genoma ou o conjunto de genes. Esse processo envolve uma série de etapas e softwares e algoritmos específicos, dependendo da tecnologia de sequenciamento utilizada. Cada plataforma de sequenciamento tem suas próprias características em termos de métodos de geração de dados, qualidade de leitura, comprimento das leituras e taxas de erro [45]. O resultado de um sequenciamento de nova geração, gera um arquivo de dados brutos que contém a leitura de até bilhões de fragmentos de DNA. As etapas envolvidas na análise dos dados é chamada de pipeline que incluem algoritmos específicos projetados para identificar características em diferentes tipos de sequenciamento, utilizando uma interface de linha de comando. O processo consiste em algumas etapas fundamentais: inicialmente, há a geração da sequência de interesse, seguida pelo alinhamento dessa sequência com o genoma de referência. Logo após, ocorre a chamada de variantes, anotação das variantes, seleção e, por fim, a elaboração do laudo clínico [45].
A primeira etapa da análise bioinformática é a verificação da qualidade dos dados. Ferramentas como o FASTQC são utilizadas para avaliar a qualidade das leituras de sequenciamento. Esta ferramenta identifica problemas como leituras de baixa qualidade, adaptadores remanescentes e regiões de baixa complexidade, que podem interferir na análise subsequente. Com base na avaliação de qualidade, é realizado o pré-processamento dos dados, que pode incluir a remoção de adaptadores, filtragem de leituras de baixa qualidade e corte das extremidades das leituras para melhorar a qualidade geral do conjunto de dados [46, 47].Os dados de sequenciamento são armazenados em arquivos no formato FASTQ, que contêm as leituras de DNA após o sequenciamento. Na etapa de alinhamento, as reads processadas são alinhadas com um genoma de referência ou um conjunto de sequências de referência relacionadas. Isso é importante para identificar a localização e a função de sequências específicas no genoma [47, 48].
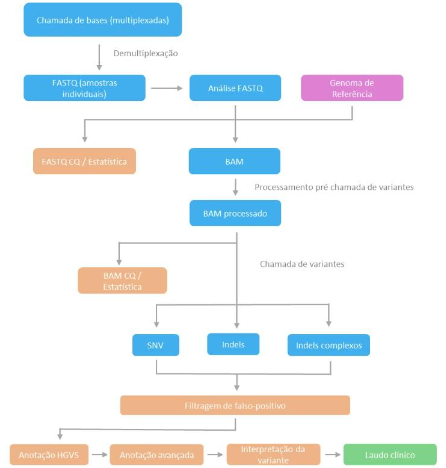
Figura 3. Esquema demonstrativo de um pipeline de análise de bioinformática para sequenciamento de nova geração [45].
Uma vez que as reads estão alinhadas, a próxima etapa é a chamada de variantes, que identifica como SNPs (Polimorfismos de Nucleotídeo Único) e indels (inserções e deleções), podem ser identificadas comparando-se as reads com o genoma de referência. Isso é útil para estudos de variabilidade genética e associação com fenótipos específicos. Analisar essas variações requer métodos avançados de bioinformática e genômica [49].
Em alguns casos, quando não há um genoma de referência disponível ou quando se deseja montar um genoma de um organismo não caracterizado, as reads são montadas em contigs ou scaffolds para reconstruir o genoma [49].
Para estudos de transcriptômica, as reads são mapeadas em transcritos conhecidos ou montadas em novos transcritos para quantificar a expressão gênica relativa entre diferentes condições experimentais [50].
Uma vez que os dados genômicos ou transcriptômicos são processados e as sequências de interesse são identificadas, elas podem ser anotadas funcionalmente para entender suas funções biológicas e possíveis interações [50].
Cada plataforma de sequenciamento (como illumina, PacBio, Oxford Nanopore, entre outros) tem suas próprias características e gera dados com diferentes perfis de erro e qualidade, o que pode afetar o processo de análise de dados. Portanto, as estratégias específicas de análise podem variar dependendo da plataforma utilizada e dos objetivos do estudo [28].
Nesse contexto, lidar com a complexidade dos dados tão vastos e heterogêneos demanda ferramentas computacionais e algoritmos de alta complexidade. A bioinformática é uma área em constante evolução. Para acompanhar essas mudanças, os pesquisadores precisam se manter atualizados com as últimas ferramentas, técnicas e métodos na área. [28, 32, 33].
Por fim, a segurança e a privacidade dos dados também são preocupações críticas, exigindo medidas especiais para garantir a conformidade com as regulamentações de proteção de dados.
Aplicações do sequenciamento de nova geração
As pesquisas sobre o genoma humano têm impulsionado significativamente o avanço das tecnologias de sequenciamento de nova geração. Atualmente, suas aplicações são de extrema relevância para diversas áreas da medicina e da ciência.
A aplicação do sequenciamento de nova geração na medicina de precisão permite prever como certas mutações podem levar ao desenvolvimento de doenças. Um exemplo importante é o sequenciamento dos genes BRCA1 e BRCA2 em casos familiares de câncer de mama e ovário hereditário [51]. A detecção de alterações patogênicas nesses genes pode indicar uma suscetibilidade aumentada à doença, orientando, assim, a conduta médica no tratamento preventivo. O NGS contribui significativamente para o diagnóstico e tratamento individualizado de doenças, possibilitando a identificação de mutações genéticas associadas a diversas condições, como doenças raras e distúrbios metabólicos [4,6].
A farmacogenômica investiga as variações no metabolismo entre diferentes indivíduos. Um exemplo proeminente da aplicação do NGS na farmacogenética, é o sequenciamento da enzima CYP2D6, uma das mais estudadas no campo da farmacogenética. Esta enzima desempenha um papel importante na orientação do tratamento para a depressão, metabolizando muitas drogas psiquiátricas. O perfil metabolizador do CYP2D6 para cada paciente tem um impacto significativo sobre os efeitos colaterais e a susceptibilidade à dependência química dessas drogas, influenciando, assim, o tempo e os custos de hospitalização [52].
Na área da microbiologia, o NGS permite a caracterização completa da microbiota, o conjunto de microrganismos que habitam nosso corpo e influenciam nossa saúde. Através do sequenciamento do DNA microbiano, é possível identificar as espécies presentes na microbiota, quantificar sua abundância e analisar suas funções metabólicas. Essa informação pode ser utilizada para o desenvolvimento de probióticos, antibióticos e outras terapias que visam modular a microbiota e tratar doenças [53].
No campo da epigenética, o NGS possibilita o estudo das modificações químicas no DNA que não alteram a sequência de bases, mas influenciam a expressão gênica. Através do sequenciamento da metilação do DNA, é possível investigar como fatores ambientais e comportamentais podem influenciar o desenvolvimento de doenças e outros processos biológicos [54].
Na área da metagenômica, a utilização do sequenciamento de nova geração vai muito além do estudo em seres humanos. A metagenômica permite, por exemplo, a identificação de microrganismos com elevado potencial biotecnológico, além de possibilitar o estudo de patologias e suas rotas de transmissão. A bioinformática é utilizada para a classificação taxonômica, resultando na identificação dos organismos presentes na amostra. Após a identificação de cada organismo na amostra, torna-se viável a montagem de seus genomas e a anotação de seus genes, o que engloba a caracterização de genes relacionados à virulência e à resistência a antibióticos [55].
Em estudos de associação genômica ampla (GWAS), o sequenciamento de nova geração é utilizado para identificar variantes genéticas associadas a doenças e outros traços fenotípicos. Através da análise de milhões de genomas individuais, é possível identificar variantes raras e comuns que aumentam o risco de desenvolver uma determinada doença. O GWAS é responsável pela identificação de diversos distúrbios de importante clínica além de traçar semelhanças genéticas entre populações [56].
O avanço contínuo das tecnologias de sequenciamento de nova geração possibilita estudos cada vez mais aprofundados do material genético de uma ampla gama de organismos. Esta evolução promete não apenas aprimorar gradualmente a qualidade de vida por meio da medicina de precisão, mas também novos produtos biotecnológicos, estudos sobre fatores epigenéticos, entre outros.
Para além desses avanços, a integração da automação laboratorial com a inteligência artificial (IA) e o aprendizado de máquina tem o potencial de expandir ainda mais as fronteiras do sequenciamento de nova geração, possibilitando o processamento e a análise de dados em uma escala significativamente maior [57].
CONSIDERAÇÕES FINAIS
A partir desta revisão, foi explorada a vasta gama de aplicações da tecnologia de sequenciamento de nova geração, destacando sua importância e seus desafios. Ademais, foi discutida a relevância da bioinformática como uma ferramenta essencial para a análise e interpretação dos enormes volumes de dados gerados pelo sequenciamento de nova geração. Através de algoritmos sofisticados e métodos computacionais avançados, a bioinformática desempenha um papel fundamental na extração de informações a partir desses dados, permitindo a identificação de padrões, a previsão de estruturas biológicas e a realização de estudos comparativos entre diferentes genomas. No entanto, é importante reconhecer que, apesar dos avanços significativos alcançados até o momento, ainda existem desafios a serem enfrentados. Questões relacionadas à qualidade dos dados, padronização de protocolos e custos associados ao sequenciamento e análise ainda representam obstáculos importantes a serem superados. Portanto, é fundamental que os pesquisadores continuem a investir em pesquisa e desenvolvimento nesta área, buscando soluções inovadoras e aprimoramentos tecnológicos que possam maximizar o potencial das ferramentas de sequenciamento de nova geração e bioinformática.
REFERÊNCIAS
- Moraes, Michelle Buscarilli de. Estudo clínico e molecular de pacientes com síndrome de Noonan e síndromes relacionadas à síndrome de Noonan pelo sequenciamento de nova geração [tese]. São Paulo: Faculdade de Medicina; 2019 [citado 2024-04-14]. doi:10.11606/T.5.2019.tde-25102019-163119.
- Stussi D Lage F, Brito F. Sequenciamento de primeira geração: método de Sanger. BIOINFO Portal Rev Bras Bioinformatica [Internet]. 2021 [citado 14 abr 2024];1(1):32-37. Disponível em: https://doi.org/10.51780/978-6-599-275326
- Shendure, J., Ji, H. Sequenciamento de DNA de próxima geração. Nat Biotechnol 26 , 1135–1145 (2008). https://doi.org/10.1038/nbt1486.
- Qin D. Next-generation sequencing and its clinical application. Cancer Biol Med. 2019 Feb;16(1):4-10. doi: 10.20892/j.issn.2095-3941.2018.0055. PMID: 31119042; PMCID: PMC6528456.
- Pereira R, Oliveira J, Sousa M. Bioinformatics and Computational Tools for Next-Generation Sequencing Analysis in Clinical Genetics. J Clin Med. 2020 Jan 3;9(1):132. doi: 10.3390/jcm9010132. PMID: 31947757; PMCID: PMC7019349.
- Carvalho MC da CG de, Silva DCG da. Sequenciamento de DNA de nova geração e suas aplicações na genômica de plantas. Cienc Rural [Internet]. 2010Mar;40(3):735–44. Available from: https://doi.org/10.1590/S0103-84782010000300040.
- Prosdocimi F. Curso online de introdução à bioinformática [Internet]. 2007 [citado 14 abr 2024].
- W. Ansorge et al. “Next-generation DNA sequencing techniques..” New biotechnology, 25 4 (2009): 195-203 . https://doi.org/10.1016/j.nbt.2008.12.009.
- B. Slatko et al. “Overview of Next‐Generation Sequencing Technologies.” Current Protocols in Molecular Biology, 122 (2018). https://doi.org/10.1002/cpmb.59.
- Verli H. Bioinformática da Biologia à flexibilidade molecular. 1ª ed. São Paulo: SBBq; 2014. 282 p. il. Disponível em: https://www.ufrgs.br/bioinfo/ebook/
- Mardis, Elaine R. “Next-generation sequencing platforms.” Annual review of analytical chemistry 6 (2013): 287-303.
- Schuster, Stephan C. “Next-generation sequencing transforms today’s biology.” Nature Methods 5.1 (2008): 16-18.
- Mardis, Elaine R. “A decade’s perspective on DNA sequencing technology.” Nature 470.7333 (2011): 198-203.
- Sarda S, Hannenhalli S. Next-generation sequencing and epigenomics research: a hammer in search of nails. Genomics Inform. 2014 Mar; 12(1):2-11. PMCID: PMC3990762.
- Aird, D.; Ross, MG; Chen, W.-S.; Danielsson, M.; Fennel, T.; Russo, C.; Jaffe, DB; Nusbaum, C.; Gnirke, A. Analisando e minimizando o viés de amplificação por PCR em bibliotecas de sequenciamento Illumina. Genoma Biol. 2011 , 12 , R18.
- International Human Genome Sequencing Consortium. Finishing the euchromatic sequence of the human genome. Nature 431, 931–945 (2004). https://doi.org/10.1038/nature0300.
- van Dijk EL, Jaszczyszyn Y, Thermes C. Library preparation methods for next-generation sequencing: tone down the bias. Exp Cell Res. 2014 Mar 10;322(1):12-20. doi: 10.1016/j.yexcr.2014.01.008. Epub 2014 Jan 15. PMID: 24440557.
- Head SR, Komori HK, LaMere SA, Whisenant T, Van Nieuwerburgh F, Salomon DR,
Ordoukhanian P. Library construction for next-generation sequencing: overviews and challenges. Biotechniques. 2014 Feb 1;56(2):61-4, 66, 68, passim. doi: 10.2144/000114133. PMID: 24502796; PMCID: PMC4351865.
- Knierim E, Lucke B, Schwarz JM, Schuelke M, Seelow D. Systematic comparison of three methods for fragmentation of long-range PCR products for next generation sequencing. PLoS One. 2011;6(11):e28240. doi: 10.1371/journal.pone.0028240. Epub 2011 Nov 30. PMID: 22140562; PMCID: PMC3227650.
- Ilumina. Preparação da biblioteca Nextera XT: dicas e solução de problemas.[Internet]. [acesso em: 26 abr 2024]. Disponível online: https://www.illumina.com/content/dam/illumina-marketing/documents/products/technotes/nextera-xt-tr oubleshooting-technical-note.pdf.
- Torrente de íons. Sequenciamento de genoma pequeno Ion PGM. [acesso em 20 mai 2024]. Disponível online: https://tools.thermofisher.com/content/sfs/brochures/Small-Genome-Ecoli-De-Novo-App-Note.pdf.
- Samorodnitsky E, Jewell BM, Hagopiano R, Miya J, Asa MR, Lyon E, Damodaran S, Bhatt D, Reeser JW, Datta J, et al. Evaluation of hybridization capture-based methods versus amplicon for whole exome sequencing. Mutat Res. 2015;36:903–914.
- Pendurado SS, Meissner B, Chávez EA, Ben-Neriah S, Ennishi D, Jones Sr, Shulha HP, Chan FC, Boyle M, Kridel R, et al. Evaluation of capture-based and amplicon approaches for developing a targeted next-generation sequencing pipeline to personalize lymphoma management. J Mol Diagn. 2018;20:203–214.
- Horn S. Target Enrichment via DNA Hybridization Capture. In: Shapiro B, Hofreiter M, editors. Ancient DNA: Methods and Protocols. Humana Press: Totowa, NJ, USA; 2012. p. 177-188.
- Kanagawa T. Bias and artifacts in multi-template polymerase chain reactions (PCR). J Biosci Bioeng. 2003;96:317–323.
- Sloan DB, Broz AK, Sharbrough J, Wu Z. Detecting rare mutations and DNA damage with sequencing-based methods. Trends Biotechnol. 2018;36:729–740.
- Harrington CT, Lin EI, Olson MT, Eshleman JR. Fundamentals of pyrosequencing. Arch Pathol Lab Med. 2013 Sep;137(9):1296-303. doi: 10.5858/arpa.2012-0463-RA. PMID: 23991743.
- MARIANO, DCB (org.) et al. BIOINFO #03 – Revista Brasileira de Bioinformática e Biologia Computacional. 3. Ed. Vol. 3. ISBN: 978-65-992753-8-8 Lagoa Santa: Alfahelix, 2023. doi: 10.51780/978-65-992753-8-8
- Rusk, N. Torrents of sequence. Nat Methods 8, 44 (2011). https://doi.org/10.1038/nmeth.f.330
- Liu L, Li Y, Li S, Hu N, He Y, Pong R, et al. Comparison of Next-Generation Sequencing Systems. J Biomed Biotechnol. 2012;2012:1–11.
- Turchetto-Zolet A, Turchetto C, Guzman F, Silva GA, Sperb Ludwig F, Vetö N. Capítulo 8 Polimorfismo de Nucleotídeo único (SNP): metodologias de identificação, análise e aplicações. 2017.
- Ravi RK, Walton K, Khosroheidari M. MiSeq: A Next Generation Sequencing Platform for Genomic Analysis. Methods Mol Biol. 2018;1706:223-232. doi: 10.1007/978-1-4939-7471-9_12. PMID: 29423801.
- Paszkiewicz K, Studholme DJ. De novo assembly of short sequence reads. Brief Bioinform. 2010 Sep;11(5):457-72. doi: 10.1093/bib/bbq020. Epub 2010 Aug 19. PMID: 20724458.
- Koutsoumanis KP, Angelidis AS. Evidence-Based Biosafety: A Review of the Principles and Effectiveness of Microbiological Decontamination Procedures for Foods and Food Contact Surfaces.
In: Schaffner DW, editor. HACCP and Hygiene Control in the Meat Industry. 1st ed. Elsevier; 2017. p. 17-50. doi:10.1016/B978-0-12-802234-4.00002-1.
- Kremer, F. S. (2019, julho 27). Entendendo o NGS (parte 1): plataformas de sequenciamento. Stussi D Lage F, Brito F. Sequenciamento de primeira geração: método de Sanger. BIOINFO Portal Rev Bras Bioinformatica [Internet]. 2021 [citado 14 abr 2024];1(1):32-37. Disponível em: https://doi.org/10.51780/978-6-599-275326
- Gupta N, Verma VK. Next-Generation Sequencing and Its Application: Empowering in Public Health Beyond Reality. Microbial Technology for the Welfare of Society. 2019 Sep 13;17:313–41. doi: 10.1007/978-981-13-8844-6_15. PMCID: PMC7122948.
- Rhoads A, Au KF. PacBio Sequencing and Its Applications. Genomics Proteomics Bioinformatics. 2015 Oct;13(5):278-89. doi: 10.1016/j.gpb.2015.08.002. Epub 2015 Nov 2. PMID: 26542840; PMCID: PMC4678779.
- Deamer D, Akeson M, Branton D. Three decades of nanopore sequencing. Nat Biotechnol. 2016 May 6;34(5):518-24. doi: 10.1038/nbt.3423. PMID: 27153285; PMCID: PMC6733523.
- YourGenome. What is Oxford Nanopore Technology (ONT) sequencing? YourGenome [Internet]. [acesso em 20 mai 2024]. Disponível em: https://www.yourgenome.org/theme/what-is-oxford-nanopore-technology-ont-sequencing
- Amarasinghe SL, Su S, Dong X, Zappia L, Ritchie ME, Gouil Q. Opportunities and challenges in long-read sequencing data analysis. Genome Biol. 2020 Feb 7;21(1):30. doi: 10.1186/s13059-020-1935-5. PMID: 32033565; PMCID: PMC7006217.
- Lin B, Hui J, Mao H. Tecnologia Nanopore e suas aplicações em sequenciamento de genes. Biossensores . 2021; 11(7):214. https://doi.org/10.3390/bios11070214.
- Carr CE, Bryan NC, Saboda KN, Bhattaru SA, Ruvkun G, Zuber MT. Nanopore sequencing at Mars, Europa, and microgravity conditions. NPJ Microgravity. 2020 Sep 7;6:24. doi: 10.1038/s41526-020-00113-9. PMID: 32964110; PMCID: PMC7477557.
- Wang, Y., Zhao, Y., Bollas, A. et al. Tecnologia de sequenciamento de nanoporos, bioinformática e aplicações. Nat Biotechnol 39 , 1348–1365 (2021). https://doi.org/10.1038/s41587-021-01108-x
- Mendelics. Nanopore: tecnologia de sequenciamento de long reads. Mendelics Blog [Internet]. [acesso em 20 mai 2024]. Disponível em: https://blog.mendelics.com.br/nanopore-tecnologia-de-sequenciamento-de-long-reads/.
- https://varstation.github.io/bioinfo-playbook/docs/get-started/
- Leggett RM, Ramirez-Gonzalez RH, Clavijo BJ, Waite D, Davey RP. Sequencing quality assessment tools to enable data-driven informatics for high throughput genomics. Front Genet. 2013 Dec 17;4:288. doi: 10.3389/fgene.2013.00288. PMID: 24381581; PMCID: PMC3865868.
- Andrews, S. (2010). FastQC: A quality control tool for high throughput sequence data. Retrieved from https://www.bioinformatics.babraham.ac.uk/projects/fastqc/.
- Silva RCC da, Alves MCS. O uso de ferramentas de bioinformática para análise de dados genéticos: uma revisão. Sci. Electronic Arch [Internet]. 24º de dezembro de 2023 [citado 26 de maio de 2024];17(1). Disponível em: https://sea.ufr.edu.br/index.php/SEA/article/view/1872
- Slatko BE, Gardner AF, Ausubel FM. Overview of Next-Generation Sequencing Technologies. Curr Protoc Mol Biol. 2018 Apr;122(1):e59. doi: 10.1002/cpmb.59. PMID: 29851291; PMCID: PMC6020069.
- Correa-Aguila R, Alonso-Pupo N, Hernández-Rodríguez EW. Multi-omics data integration approaches for precision oncology. Mol Omics. 2022 Jul 11;18(6):469-479. doi: 10.1039/d1mo00411e. PMID: 35470819.
- Petrucelli N, Daly MB, Feldman GL. Hereditary breast and ovarian cancer due to mutations in BRCA1 and BRCA2. Genet Med. 2010 May;12(5):245-59. doi: 10.1097/GIM.0b013e3181d38f2f. PMID: 20216074.
- Yang Y, Botton MR, Scott ER, Scott SA. Sequencing the CYP2D6 gene: from variant allele discovery to clinical pharmacogenetic testing. Pharmacogenomics. 2017 May;18(7):673-685. doi: 10.2217/pgs-2017-0033. Epub 2017 May 4. PMID: 28470112; PMCID: PMC5591463.
- Hernández M, Quijada NM, Rodríguez-Lázaro D, Eiros JM. Aplicación de la secuenciación masiva y la bioinformática al diagnóstico microbiológico clínico [Bioinformatics of next generation sequencing in clinical microbiology diagnosis]. Rev Argent Microbiol. 2020 Apr-Jun;52(2):150-161. Spanish. doi: 10.1016/j.ram.2019.06.003. Epub 2019 Nov 26. PMID: 31784184.
- Meaburn E, Schulz R. Next generation sequencing in epigenetics: insights and challenges. Semin Cell Dev Biol. 2012 Apr;23(2):192-9. doi: 10.1016/j.semcdb.2011.10.010. Epub 2011 Oct 19. PMID: 22027613.
- Biome Hub. Bioinformática e metagenômica. Biome Hub. https://www.biome-hub.com/post/bioinformatica-e-metagenomica#:~=A%20metagen%C3%B4mica%2 0permite%20o%20estudo,incluindo%20bact%C3%A9rias%2C%20v%C3%ADrus%20e%20fungos. Acesso em: 25 mai de 2024.
- Tam V, Patel N, Turcotte M, Bossé Y, Paré G, Meyre D. Benefits and limitations of genome-wide association studies. Nat Rev Genet. 2019 Aug;20(8):467-484. doi: 10.1038/s41576-019-0127-1. PMID: 31068683.
- Celesti F, Celesti A, Wan J, Villari M. Why Deep Learning Is Changing the Way to Approach NGS
Data Processing: A Review. IEEE Rev Biomed Eng. 2018;11:68-76. doi: 10.1109/RBME.2018.2825987. Epub 2018 Apr 12. PMID: 29993643.
1Graduanda do Curso de Biomedicina do Centro Universitário das Faculdades Metropolitanas Unidas-FMU, Brasil
2Biomédica, Profa. Dra. do Curso de Biomedicina do Centro Universitário das Faculdades Metropolitanas Unidas-FMU, Brasil
