REGISTRO DOI:10.5281/zenodo.11005365
Pedro Victor Da Silva
Ana Verônica Silva do Nascimento
RESUMO
A batata-doce (Ipomoea batatas (L) Lam.) é uma hortaliça de importância nacional, uma vez que é a quarta hortaliça mais consumida no Brasil com grande adaptação aos diversos sistemas de climas e solos, possuindo baixo custo de produção. Nacionalmente, as regiões Sul e Nordeste destacam-se por serem responsáveis por grande parte da produção do país. Contudo, perdas significativas na produção têm sido observadas decorrente da as infeções de patógenos fúngicos, em destaque a espécie Fusarium Oxysporum. Dessa forma,o presente trabalho teve por objetivo fazer um estudo in sílico do gene LYS2 Fusarium Oxysporum f. sp. rapae com o intuito de obter possível oligonucleotídeos especifico para kit de diagnóstico molecular. As buscas das sequências gênicas foram realizadas utilizando a plataforma da plataforma do NCBI e ferramenta blast foram selecionadas 15 sequencias para em seguida gerar a árvore filogenética com as sequencias selecionadas e depois desenhar os marcadores para qPCR. Os resultados obtidos da arvore filogenética, com o método de inferência bootstrap, apresentaram bons resultados de similaridades entre as espécies. Os oligonucleotídeos desenhados com a plataforma primer3Plus demonstraram serem bons marcadores para diagnosticar o F. oxysporum na batata doce, sendo um protocolo para técnica qPCR.
Palavras-chave: Batata-doce; Primers; kit diagnostico.
1- INTRODUÇÃO
A batata-doce é uma dicotiledônea pertencente à família Convolvulaceae, gênero Ipomoea e espécie Ipomoea batatas (L) Lam que agrupam aproximadamente 50 gêneros e mais de 1000 espécies, mas somente a batata-doce tem cultivo de expressão econômica. É uma planta de clima tropical ou subtropical, podendo ser cultiva em regiões temperadas. Esta espécie vem despertando grande interesse dos pesquisadores por conta da diversidade de material existente no Brasil, por sua rusticidade e alta produtividade, além de destaca-se por seu cultivo e por um baixo custo de implantação (Mota et al. 2011). A batata doce é um alimento fundamental na dieta brasileira, especialmente no Nordeste do país, onde desempenha um papel vital tanto na nutrição quanto na economia regional. Segundo IBGE, (2020) a batata doce é valorizada por sua resistência a condições climáticas adversas, sendo uma escolha robusta para os agricultores da região. Segundo Mota et al. (2011) destaca que, além de ser uma fonte rica em carboidratos, vitaminas A e C, a batata doce é também uma importante fonte de renda para pequenos produtores, devido à sua demanda constante nos mercados locais e nacionais. Essas características tornam a batata doce um componente essencial não apenas na dieta dos brasileiros, mas também em seus aspectos socioeconômicos, especialmente no Nordeste, onde a agricultura é uma parte crucial da identidade cultural e econômica.
As doenças da batata doce representam um desafio significativo para os agricultores, impactando diretamente a produtividade e a qualidade dos cultivos. Dentre as doenças, destaca-se o Fusarium oxysporum f. sp. rapae sendo um patógeno fúngico que representa uma ameaça significativa à produção de batata doce no estado da Paraíba. A infecção desse fungo ocorre principalmente através do solo, onde o fungo pode persistir por longos períodos mesmo na ausência de plantas hospedeiras, como mencionado por Martins et al. (2020), acarretando os sintomas de murcha, amarelecimento e necrose das folhas, comprometendo severamente a qualidade e o rendimento das culturas.
A compreensão do ciclo de vida e da biologia de Fusarium oxysporum f. sp. rapae é crucial para o desenvolvimento de métodos de controle mais eficazes e sustentáveis, destacando a importância da pesquisa contínua nesta área. A detecção molecular de Fusarium oxysporum em batata doce é uma área crucial para a prevenção e manejo eficaz da murcha de Fusarium, uma das doenças mais devastadoras dessa cultura. As técnicas de detecção molecular permitem identificar o patógeno com precisão e rapidez, mesmo em estágios iniciais da infecção, o que é fundamental para o controle eficiente da doença. A PCR (Reação em Cadeia da Polimerase) é uma das técnicas mais utilizadas para a detecção de Fusarium oxysporum, como destacado por Santos e Almeida (2019). Esta técnica permite a amplificação de segmentos específicos do DNA do patógeno, facilitando sua identificação rápida e precisa. Outra abordagem é a qPCR (PCR quantitativa), que, conforme descrito por Oliveira et al. (2020), não apenas detecta, mas também quantifica a carga do patógeno, proporcionando uma avaliação da severidade da infecção. Essas tecnologias não só melhoram a precisão na detecção de doenças, mas também contribuem para a implementação de estratégias de manejo integrado de doenças, reduzindo a dependência de fungicidas e apoiando práticas agrícolas mais sustentáveis. Dessa forma, O desenvolvimento de kits de diagnóstico para Fusarium, particularmente para patógenos como Fusarium oxysporum, é essencial para o manejo eficaz das doenças em culturas agrícolas. Estes kits oferecem métodos rápidos, precisos e acessíveis para a detecção do patógeno, contribuindo significativamente para a prevenção e controle de surtos de doenças. Portanto, considerando que o estado da Paraíba se destaca como produtor de batata doce (Soares et al., 2002), e que as técnicas moleculares são ótimas opções para diagnósticos de fitopatógenos, o presente trabalho teve como objetivo analisar in sílico a filogenia do gene LYS2 de espécie Fusarium Oxysporum f sp rapae e desenvolver oligonucleotideos para diagnóstico do fungo em batata doce.
2- METODOLOGIA
O presente trabalho foi realizado inteiramente in sílico, com uso de ferramentas de softwares e servidores para obter e manipular os dados referentes ao gene LYS2 (acesso GenBank – AB586946.1) relacionado ao patógeno fúngico Fusarium oxysporum em batata-doce (Ipomoea batatas).
TRIAGEM E OBTENÇÃO DAS SEQUÊNCIAS
Foram feitas pesquisas na plataforma do NCBI com a ferramenta PubMed (https://pubmed.ncbi.nlm.nih.gov/) com o intuito de encontrar o gene de conservação dentro do Fusarium oxysporum, e comoresultado foi escolhido o gene da LYS2, visto que é o mais promissor relacionado com conservação dentro da espécie de estudo e demais especeis do gênero. A sequência do gene foi obtida com a mesma plataforma, mas com a ferramenta do GenkBank (https://www.ncbi.nlm.nih.gov/genbank/). Para encontrar as sequencias similares com o gene da LYS2, foi utilizado a ferramenta do mesmo servidor, o BlastG (https://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE=gene), sendo coletado 15 sequências juntamente com a pesquisa. Nas sequências da pesquisa, foram adicionadas na triagem tanto espécies do Fusarium spp quanto de outros microrganismos, levando em consideração o grau de similaridade entre o gene escolhido (Santos, 2021).
ANÁLISE FILOGENÉTICA E AGRUPAMENTO DAS SEQUÊNCIAS
As sequências gênicas obtidas, foram primeiramente alinhadas pelo algoritmo ClustalW do software MEGA XI para visualização dos dados e em seguida, usando o mesmo software, a produção da árvore filogenética (Santos, 2021). A construção da árvore filogenética deu-se pelo método de máxima parcimônia com bootstrap de mil réplicas, modelo de substituição padrão do programa, realizado um total de 15 sequências para análises.
PRODUÇÃO E VALIDAÇÃO DE PRIMERS
Com o uso do servidor NCBI (https://www.ncbi.nlm.nih.gov/pmc/), foi coletado a sequência de DNA do gene da lisina (LYS2). Posterior, os primers foram desenhados nos servidores Primer-blast no NCBI (https://www.ncbi.nlm.nih.gov/tools/primer–blast) e Primer3Plus (https://www.bioinformatics.nl/cgi–bin/primer3plus/primer3plus.cgi) ( Vilhena, 2015). Para validação dos primers, foi usado o servidor Oligo Cal (http://biotools.nubic.northwestern.edu/OligoCalc.html) (Kibbe, 2007; Santos, 2021).
PROCOTOLO DE EXTRAÇÃO DE DNA E TÉCNICA RT-qPCR e LAMP
Foram feitas triagens na literatura de protocolos de extração de DNA de fitopatógenos fúngicos para a extração de DNA visando qual teria maior custo benefício e menor tempo de processo, e assim foi indicado para o kit de extração de DNA, DNeasy Plant Mini Kit (Qiagen Brasil).
3- RESULTADOS E DISCUSSÃO
A partir das buscas de dados feitas na plataforma no NCBI, foram coletadas sequências depositadas até o ano de 2022 que fosse similar com a sequência do gene de interesse, lisina – LYS2, usando a ferramenta Blast presente na plataforma. A Tabela 1 demonstra os fungos e o código de acesso das sequências depositadas no banco de dados.
Tabela 1 – Informações relacionadas com as sequências do Gene LYS2. Dados coletadas com a ferramenta Blast da plataforma NCBI
| Fungo | Código Acesso |
| Fusarium oxysporum f. sp. rapae | AB586946.1 |
| Fusarium subglutinans | AB586959.1 |
| Fusarium proliferatum | AB586957.1 |
| Fusarium fujikuroi | AB725608.1 |
| Fusarium phyllophilum | AB586956.1 |
| Fusarium verticillioides | AB586960.1 |
| Fusarium musae putative | XM_044820443.1 |
| Fusarium mangiferae putative | XM_041828816.1 |
| Fusarium tjaetaba | XM_037352590.1 |
| Fusarium redolens | XM_046191600.1 |
| Fusarium odoratissimum | XM_031212870.1 |
| Fusicolla violacea | AB586949.1 |
| Ilyonectria robusta | XM_046252900.1 |
| Cordyceps militaris | XM_006668541.1 |
| Metarhizium brunneum | XM_014686153.1 |
O alinhamento das sequencias com a ferramenta de algoritmo ClustalW do software MEGA XI. Logo após ao alinhamento com a ferramenta de algoritmo ClustalW do software MEGA XI, com a ferramenta de construção de arvores filogenéticas e com o método de máxima parcimônia e de inferência o bootstrap de mil réplicas, foi construída a árvore filogenética (Figura 1), para entender o grau de similaridade entres as espécies do gênero Fusarium spp, uma vez que há sequencias similares dentre e entre espécies.
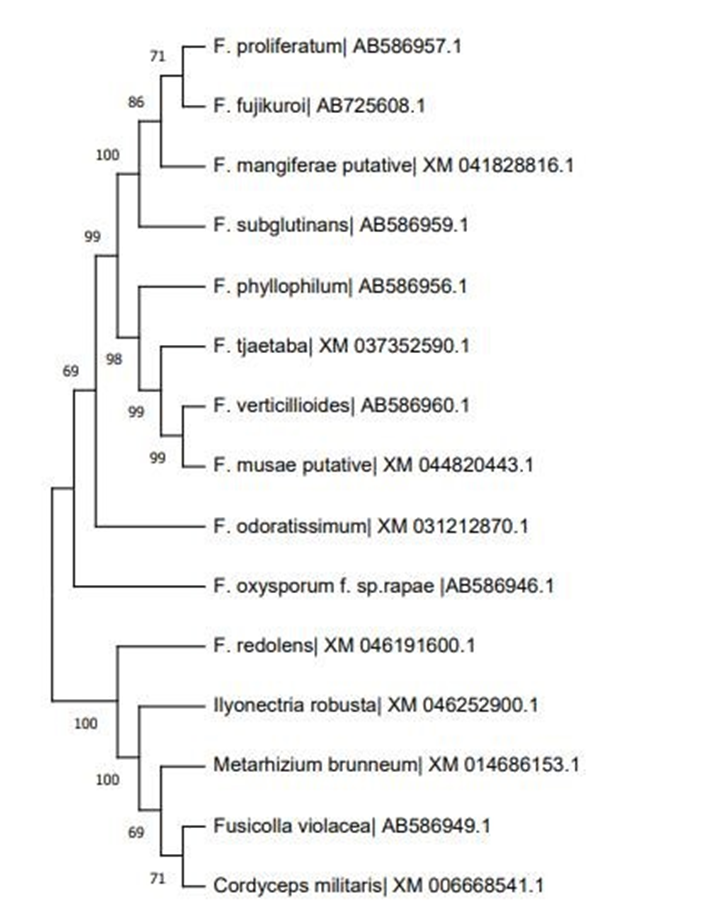
Analisando a árvore filogenética, Figura 1, pode-se observar uma homogeneidade grande, já que, dentre as 15 sequências, 11 são do gênero Fusarium spp. É possível notar que o grupo existente é muito espesso, possuindo 6 espécies: F. odoratissimum, F. oxysporum f.sp rapae, F. redolens, Ilynoctria robusta, F. phyllophilum, e F. subglutinans. Ademais, os Fusarium estão presentes também, formando grupos: F. proliferatum e F. fujikuroi, F. verticillioides e F. musae putative, e Fusicolla violácea e Cordyceps militaris. A análise filogenética demonstra que o grupo da espécie Fusarium oxysporum f. sp. rapae, formou um clado distinto das demais espécies, como ocorreu na arvore filogenética da Figura 1 e nos trabalhos realizados por Watanabe et al., (2011). Ainda, Watanabe et al., (2011) apresentam resultados de filogenia com as espécies do Fusarium com bastante similaridade usando o método da máxima verossimilhança envolvendo mais um gene, incluindo o gene da LYS2. Diante dos resultados obtidos destes autores, um clado na árvore filogenética apresentou uma relação de espécies que estão presente neste trabalho, com o teste de confiabilidade de mais de 90% em relação ao método utilizado pelos autores. Além disso, os autores enfatizam a importância do estudo filogenético com o Fusarium, visto que os conhecimentos taxonômicos possibilitam conhecer a evolução de um organismo de importância socioeconômica. Ocasionalmente, a filogenia molecular e a inferência da classificação taxonômicas das espécies é uma concepção plausível, visto que a árvore filogenética de gene atribui sua construção em um conjunto de homólogos, isso a partir de dados moleculares que podem ter representação de uma espécie (Santos, 2021).
Figura 1– Árvore filogenética bootstrap consensus do gene LYS2 do microrganismo Fusarium oxysporum f. sp. rapae obtida no MEGA-XI.
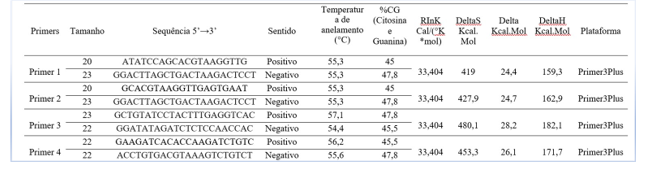
Após a análise da arvore filogenética, foi desenhado os oligonucleotídeos específicos das sequências estudadas para a viabilização de possível estudo experimental relacionado ao diagnóstico de Fusarium em estudo na espécie de batata-doce (Tabela 2). Na presente tabela 2 é possível observar a sua sequência, sentido, tamanho e outras informações de relevância para realização do trabalho em bancada (in situ) na parte de execução da qPCR, para que encontre a região de interesse do gene na sequência do
Fusarium que irá diagnostica a Ipomoea batatas como infetada ou não. Desta forma, a sequência do gene foi usada na construção dos oligonucleotídeos, uma vez que o gene LYS2 estar relacionado com a síntese de lisina e é altamente específico em fungos, e tem uma relação positiva dentro do gênero Fusarium (Watanabe et al., 2011), razão pela qual foi escolhido o referido gene. Com a sequência do DNA do gene, foi possível determinar os primers e quais deles se estabelecia dentro dos parâmetros estimados. Assim, após a construção dos devidos primers, pode-se analisar os três parâmetros relacionados a sua qualidade: parâmetro 1 – tamanho (desejável de 18 a 28 bases – específicos e estáveis); parâmetro 2 – temperatura de anelamento (dentre 50 a 60 °C); e parâmetro 3 – porcentagem de guaninas e citosinas (preferível entre 45 a 55%). Diante de todos esses parâmetros quanto mais próximo dos resultados desejáveis, mais especifico, eficiente e seguro será o primer.
Tabela 2 ¬ Oligonucleotideos do gene LYS2 de Fusarium oxysporum f. sp. rapae
A partir da determinação do melhor do oligonucleotideos, segundo os critérios estabelecidos, é indicado um protocolo de qPCR com as sequências do gene da lisina (LYS2). Para a extração de DNA em tecidos vegetais, é utilizado o kit de extração de DNA de fungos DNeasy® Plant Kit (Qiagen Brasil). Para realização da qPCR, é utilizado o DNA extraído das amostras, com o kit de extração de DNA, DNeasy Plant Mini Kit (Qiagen Brasil). O procedimento de detecção de qPCR consistente do método descrito por Chai et al. (2020). Assim, 20µL volume de reação contendo 10µL de SuperReal PreMix Plus, 0,2µL de Primers 3/Primers 4 (10µM) (Tabela 2), 1µL de DNA modelo (210 ng) e 0,4µL de 50×Corante de referência ROX. Para a amplificação é usado o sistema ABI 7500 Real-Time PCR (Applied Biosystems Brasil). A fluorescência é detectada após cada ciclo, em que será possível fazer o diagnóstico molecular.
4- CONSIDERAÇÕES FINAIS
- Foi possível observar uma identidade próxima do gene LYS2 no agrupamento filogenético;
- Os oligonucleotidoes obtidos na plataforma Primer3Plus (3 e 4) foram os mais indicados na análise in sílico;
- Foi possível propor um kit diagnóstico utilizando a técnica qPCR com os oligonucleotídeos obtidos in sílico diagnóstico do fungo em batata doce.
5 REFERÊNCIAS
IBGE. Produção Agrícola Municipal: Tabela 1612 – Área plantada, área colhida, quantidade produzida e valor da produção da lavoura temporária. Rio de Janeiro:
SIDRA, 2020.
KIBBE, W. A. OligoCalc: an online oligonucleotide properties calculator.
Nucleic AcidsResearch. v. 35, p. 43 – 46, 2007.
MOTA, J. H. et al. Qualidade de raízes de batata-doce comercializadas em Jataí-GO. Congresso Brasileiro de Olericultura. Voçosa, 2011. Disponível
em:https://www.alice.cnptia.embrapa.br/bitstream/doc/897495/1/JONYEISHIYURI120
11.pdf. Acesso em: 10 mar. 2023.
O’DONNELL, K. et al. Phylogenetic diversity of insecticolous fusaria inferred from multilocus DNA sequence data and their molecular identification via FUSARIUM-ID and Fusarium MLST. Mycologia, v.104, n. 2, p. 427-445, 2012.
SANTOS, D. S. MAPEAMENTO E ANÁLISE IN SILICO DAS PROTEÍNAS REFERENTESAO ESTRESSE SALINO EM ESPÉCIES DO GÊNERO DO ALGODO
EIRO (Gossypium spp). 2021. Monografia. Universidade Federal de Campina Grande.
SOARES, K.T.; MELO, A.S. de; MATIAS, E.C. A cultura da batata-doce (Ipomea batatas (L.) Lam). Joao Pessoa: EMEPA-PB, 2002. 26 p. il. (EMEPA-PB. Documentos, 41). SPERIMENTAZIONE. Sweetpotato. S/L,2021. Disponível em: http://sperimentazione.altervista.org/Sweetpotato.html. Acesso em: 26 março 2023.
VILHENA, A. P. Brimer: Um sistema web para gerenciamento de primers para o aboratório de tecnologia biomolecular da UFPA.2015. Monografia (Bacharel em Siste mas de Informação) Universidade Federal do Sul e Sudeste do Pará, Marabá, 2015, 70p. Disponível em: Acesso em: 09 de abril. de 20223.
WATANABE, Maiko et al. Molecular phylogeny of the higher and lower taxonomy of the Fusariumgenus and differences in the evolutionary histories of multiple genes. BMC Evolutionary Biology, v. 11, n. 1, p. 1-16, 2011.
