REGISTRO DOI: 10.5281/zenodo.11110692
Débora Souza Dos Santos1
Ana Verônica Silva do Nascimento1
Horace José Jimenez2
RESUMO
A cotonicultura é uma plantação muito importante no Brasil, detentora do quinto lugar de maior produtora e terceira maior exportadora mundial. Nacionalmente, o plantio de algodão herbáceo concentra-se no centro-oeste e nordeste, onde é majoritariamente cultivado por irrigação, seguido da técnica de sequeiro e sem rega. Os fatores climáticos, alta evapotranspiração e baixa pluviosidade, qualidade da água de irrigação, manejo, características do solo e espécie, são determinantes para existência e agravamento do estresse salino no plantio, sendo um dos mais importantes estresses abióticos, juntamente com o hídrico, a afetar o crescimento, produtividade e qualidade da fibra. Para isto, o melhoramento genético vegetal é a alternativa mais viável para redução das consequências geradas na planta pelo excesso de sais, como uso de produtos químicos na lavoura e perda quantitativa e econômica da produção. Com desenho de primers in silico que reconhecerão os domínios conservados no gene relacionados com a tolerância e/ou resistência, viabilizando a manipulação genética para criação de cultivares mais adaptadas a este fator. Assim, a partir da bioinformática, o presente trabalho, analisou taxonomicamente e físico-quimicamente três proteínas de interesse selecionadas, segundo triagem prévia, realizando desenho dos primers, validação destes e seleção dos dez primers mais estáveis, seis das proteínas e quatro degenerados, os quais são seguros, específicos e estão de acordo com padrões qualitativos de referência. De extrema relevância para área experimental, o conhecimento do perfil, ação das proteínas e desenho dos marcadores moleculares realizados, o caminho para se minimizar os problemas gerados pela salinidade ao cultivo é palpável.
Palavras-chave: Cotonicultura; Salinidade; Primers.
1. INTRODUÇÃO
A família Malvaceae, gênero Gossypium, é referente popularmente ao algodoeiro. Cultivo datado a mais de 4 mil anos (Saldanha; Belo, 2016; De Sousa, 2010) com a finalidade de utilização de sua fibra para o setor têxtil e sementes para alimentação animal e extração de óleos vegetais (Blanco, 2008), entre outras finalidades ligadas a bioenergia e outros setores. Apresenta nesse gênero, mais de 50 espécies, sendo quatro as de maior valor econômico e cultivo em grande escala, G. arboreum L. e G. herbaceum L. diplóides, G. hirsutum L. e G. barbadense L. tretraplóides, respectivamente em pares, nativas do Velho Mundo e Novo Mundo (De Sousa, 2010).
Nacionalmente, o algodoeiro é uma cultura de bastante importância, sendo o quinto maior produtor mundial na safra de 2017/2018, somando com a produção dos quatro países à sua frente 76,66% da produção mundial (Da Silva, 2020). Nacionalmente, pela última atualização do IBGE, a produção do algodão herbáceo em 2021 tem possibilidade de ultrapassar a do ano anterior, apresentando na primeira safra já quase 6 milhões de toneladas produzidas até abril de 2021 em relação ao total de 7 milhões do anterior, 2020 (IBGE, 2021).
Regionalmente, no Brasil, o centro-oeste concentra 73,3% da produção brasileira, seguido pelo Nordeste com 29,3%, sudeste com 3%, norte com 0,3, já o Sul não tem dados, de acordo com a produção do ano de 2020 (IBGE (2021). Tais estados, encontram-se no clima semiárido e vegetação caatinga, em sua maior parte, exceto o estado do Maranhão que possui majoritariamente o bioma cerrado, juntamente com os estados do centro-oeste.
Esta área se caracteriza pelas altas temperaturas, eventos de estiagem prolongados, baixa pluviosidade, solos com baixo potencial de retenção de água, alta infiltração e evapotranspiração que faz com que haja uma maior concentração de sais e nutrientes neste solo (Hoffmann Neto, 1999; De Sena, 2011; Correia, et al., 2011). E consequente uma presença maior de poços artesianos e amazonas, nestes locais, pois a acumulação aquífera se dá subterraneamente, principalmente na caatinga, já que o cerrado é o “berço das águas brasileiro”, possuindo muitas nascentes superficiais em seu território (Lima, 2011).
Apesar da cultura ser adaptada a temperaturas entre 18 e 30°C, com ciclo relativamente curto, podendo ter cultivares com 100-120 dias, alta taxa de produtividade por hectare, no caso 3mil Kg/ha (De Azevedo, Silva, 2007), este ambiente promove à cultura de algodão um certo nível de estresse abiótico. Normalmente, é usado o regime de sequeiro para plantio, porém apesar de autores indicá-lo (De Azevedo, Silva, 2007; Mattos, Et Al., 2020; Mota, 2014), pode não compensar o uso desta técnica pela ocorrência de veranicos durante a fase fenológica, pois o solo pode não manter a umidade necessária para o desenvolvimento da planta. Assim a cultura do algodoeiro por irrigação ganha mais espaço pela maior segurança e produtividade (Mota, 2014), reforçado por 60% de seu cultivo ser por esta técnica (Blanco, et al., 2011; Pereira, 2012).
Pelo alto teor de sais nas fontes aquíferas usadas na irrigação aliada a um solo com baixa retenção, que gera acúmulo de sais, faz com que a cultura esteja exposta a estresses salinos que irá comprometê-la a médio e/ou longo prazo fisiologicamente, bioquimicamente, na absorção de nutrientes, e consecutivamente seu desenvolvimento e produtividade (Amorim, et al, 2010), além do meio ambiente, necessitando de um manejo que minimize as consequências geradas pelo alto teor de salinidade no ambiente e planta, assim como de métodos biotecnológicos que propicie um algodoeiro mais resistente a este fator abiótico.
Devido a esta necessidade, uma alternativa viável é o uso do melhoramento genético vegetal para propiciar cultivares mais adaptadas ambientalmente ao excesso de sais, devido ao seu cultivo em regiões com este perfil (Soares Filho, et al., 2016). Com isto, o objetivo deste estudo é o uso de banco de dados para triagem de proteínas de interesse, realização de análise físico-química e estrutural destas sequências e, a partir disso, desenho dos primers in silico a fim de gerar conhecimento para obtenção experimental de vegetais com estas características.
2. METODOLOGIA
O presente trabalho foi feito inteiramente in silico utilizando softwares e servidores para a obtenção e manipulação de dados referentes as proteínas relacionadas ao estresse salino em algodoeiro. Para o estudo das biomoléculas, foi adaptado o estudo de Jimenez (2019), já o desenvolvimento do desenho e validação de primers, foi utilizado como base metodológica diversos autores, como Queiroz (et al., 2017), Ye (2012), Vilhena (2015), Kalendar, Lee, Schulman (2014) e Kibbe (2007). Foi realizada a triagem das sequências, assim como a obtenção de informações sobre o genoma das espécies de Gossypium que tem depositadas e a coleta das sequências de proteínas taxonômicas referente ao estresse salino, foram adquiridas a partir das ferramentas da plataforma NBCI (National Center for Biotechnology Information) (https://www.ncbi.nlm.nih.gov/pmc/) e pela ferramenta do mesmo servidor, BlastP (https://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE=Proteins), coletando 16 sequências juntamente com a pesquisada, adicionando espécies tanto de algodoeiros quanto de outros organismos na triagem, levando em consideração o grau de similaridade entre a proteína escolhida na coleta (Jimenez, 2019).
• Análise das sequências e Produção e validação de primers
Para estudo das características das sequências de proteína, foi usado o servidor ProtParam (https://web.expasy.org/protparam/) para o estudo físico-químico; o NCBI (https://www.ncbi.nlm.nih.gov/pmc/) para obtenção e estudo dos domínios conservados das sequências (Jimenez, 2019). Para estudo da estrutura terciária foi usado o software Phyre2 (http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index); análise da estrutura tridimensional de hidropaticidade pelo software Chimera; servidor Molprobity (http://molprobity.biochem.duke.edu/) para análise de Ramachandran para avaliação da qualidade conformacional dos modelos teóricos protéicos; análise de Z-score e da energia dos scores foi realizada a partir do servidor ProSA-web(https://prosa.services.came.sbg.ac.at/prosa.php) (Jimenez, 2019).
Na validação dos primers foi usado o servidor NCBI (https://www.ncbi.nlm.nih.gov/pmc/) para coleta das sequências de RNA dos genes que traduzem as proteínas de interesse. Após esta etapa foi feita o corte da sequência nas regiões específicas do gene relacionada com o domínio conservado, relacionado ao estresse salino (Queiroz, et al., 2017). Após esta etapa, foi desenhado os primers nos servidores Primer-Blast no NCBI (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) e Primer3Plus(https://www.bioinformatics.nl/cgi-bin/primer3plus/primer3plus.cgi) (Ye, 2012; Vilhena,2015), além de validação do desenho pelo servidor OligoCalc (http://biotools.nubic.northwestern.edu/OligoCalc.html) (Kibbe, 2007). Para o desenho do primer degenerado respectivos as três proteínas, foi feito alihamento das sequências cortadas no MEGA-X pelo algoritmo Clustal W (Queiroz, et al., 2017) e em seguida desenhada no software FastPCR (Kalendar, Lee, Schulman, 2014).
3. RESULTADOS E DISCUSSÃO
Para o estudo foram utilizadas as sequencias gênicas depositadas no ano de 2021 no banco de dados NCBI. Foi realizada uma triagem referente a genes com potencial de prover tolerância à resistência ao estresse salino no vegetal. Com isto, foi selecionado três proteínas taxonômicas para estudo, Tabela 1, dentre o gênero Gossypium. A primeira, pertencente ao domínio Rop_like, tem na literatura a confirmação sobre seu potencial que, de acordo com Finkelstein, Gampala, Rocha (2002), este domínio está relacionado a resposta ABA (ácido ascórbico), o qual é um fitohormônio que modula processos do crescimento, desenvolvimento e estresses abióticos nas plantas, dentre estes, está o estresse salino.
Já a segunda, do domínio do citocromo P450, tem em sua descrição sobre os mecanismos de ação que os seus domínios conservados estão relacionados, mostra-se diretamente relacionada ao estresse em questão, assim como Yan (et al., 2016) indica em seu estudo com a expressão dessa proteína em plantas transgênicas de Nicotiana benthamiana submetidas a condição de redução hídrica e aumento da salina. E a terceira, do domínio bZIP, respectivamente, está relacionada aos mecanismos de ação à estímulos fisiológicos, dentre os quais, de acordo com Wang (et al., 2021), que indicou associação dos genes deste domínio com estresse abiótico, mais especificamente os JcbZIPs 34, 36, 49 e 50 à resistência ao estresse salino e seca.
Tabela 1 – Informações sobre as proteínas taxonômicas com função sobre estresse salino em plantas do gênero Gossypium, informações retiradas do NCBI
| Proteína | Função | Código de acesso |
| Proteína de ligação a GTP semelhante a rac ARAC8 [Gossypium hirsutum] | Estresse | NP_001314362.1 |
| Citocromo P450 tipo CYP82D47 [Gossypium hirsutum] | Estresse salino | NP_001314395.1 |
| Proteína de fator de choque térmico semelhante a HSF8 [Gossypium hirsutum] | Estresse | NP_001314347.1 |
Após a validação das funções das três proteínas selecionadas, foi utilizada, a mesma, para estudo físico-químico no servidor ProtParam, Tabela 2. Estes resultados são imprescindíveis para o conhecimento da sequência da proteína em estudos experimentais. Sobre o ponto isoelétrico, este é o valor de pH em que a molécula biológica possui bom desempenho em suas funções, imprescindível para estudo experimental, precisamente durante a corrida do gel onde as proteínas são separadas de acordo com seu gradiente de pH na corrida do gel, se movimentando a partir desse diferencial, e parando no momento em que atingem a carga total igual a zero, ou seja, seu ponto isoelétrico (pI), atingindo a posição estacionária (Brum, 2007), além do fato de suas propriedades serem influenciadas pelo pH do meio circundante, ou seja, a carga dos resíduos de aminoácidos muda conforme o meio, e por conseguinte, propriedades também, como a eletrostaticidade (Verli, 2014), possui também, suma importância laboratorial no uso de primers, em que o pH se encontra nos cálculos básicos para definir a temperatura de anelamento ou temperatura de melting destes, primordial na viabilidade prática do desenho, em que tais sequências em estudo, Tabela 5, serão a base para a construção do primer (De Arruda Júnior, 2010).
Já o peso molecular relaciona-se ao estudo experimental da proteína, permitindo identificá-la e isolá-la por cromatografia, por exemplo, pois o peso é característico à quantidade de aminoácidos em sua sequência, sendo uma referência para determinação (Guerra, et al., 2011). Na cromatografia, a separação é de proteínas ocorre conforme o volume e carga molecular, os quais podem ser usados para determinação da massa e consecutivamente o peso molecular, com a eletroforese unidimensional, a qual possui menor capacidade de separação, menos de 50 proteínas geralmente em relação a bidimensional, a massa molecular é o fator determinante na corrida, pois quanto menor a massa, mais rápida é a movimentação da molécula sob o gel. Ao se falar da técnica de eletroforese bidimensional que consiste na separação de proteínas para estudo proteômico, os fatores que influenciam na corrida são o ponto isoelétrico e massa molecular (Moraes, et al., 2013).
O Gravy (Grand average of hydropathicity) é o índice de hidropaticidade que expressa a soma de hidropaticidade dos aminoácidos dividida pelo número de resíduos, este índice aponta o quão solúvel a proteína é com valores positivos para hidrofóbicas e negativos para hidrofílicas (De Lima, 2015), no caso das proteínas em estudo, Tabela 5, todas são hidrofílicas, o que é um bom fator ao se trabalhar na prática pois a forma de se trabalhar com a biomolécula irá se adequar à tal característica, além de indicar uma estrutura mais compacta e globular, funções biológicas mais dinâmicas, como transporte de gases em que a hemoglobina é responsável, já as hidrofóbicas tendem a ter uma conformação fibrilar em forma de um cordão e função mais estrutural, como o colágeno na formação dos tecidos (Marques, 2014).
O índice de estabilidade provém o nível de estabilidade que a biomolécula possui em um tubo de ensaio, a qual é considerada estável abaixo de quarenta e instável acima deste valor (De Lima, 2015). Outra característica importante é o índice de meia vida que mostra o tempo que a proteína se mantém ativa in vitro e in vivo, em diferentes organismos, o que direciona o pesquisador para a manipulação deste material biológico em análise.
Tabela 2 – Características físico-químicas das proteínas, informações retiradas do ProtParam.
| Proteína | Meia vida | Índice de instabilidade | Grande média de hidropatia (GRAVY) | Ponto isoelétrico | Número de aminoácidos | Peso molecular |
| Rop_like | 30 horas (reticulócitos de mamíferos, in vitro). > 20 horas (fermento, in vivo). > 10 horas (Escherichia coli, in vivo). | Grau II = 41,63 | -0,217 | 9,02 | 198 | 21892,23 |
| P450 | Grau II = 36,93 | -0,211 | 8,83 | 522 | 58489,73 | |
| Hsf8 | Grau II = 63,54 | -0,727 | 4,78 | 515 | 56661,67 |
No estudo da estrutura tridimensional das proteínas, Figura 1 (a,b,c), foi observado domínios diferentes, porém apresentam mesma função relacionada a tolerância, resistência ao estresse salino, podendo divergir, provavelmente, em seu mecanismo de ação ou até grupo catalítico. Ademais, quanto as características conformacionais, na Figura 1, (a) referente a proteína do domínio Rop like tem grande quantidade de folhas-beta em relação a alfa-hélice, e ligações de looping ligando estas entre si; a proteína do domínio P450 tem grande quantidade de folhas-beta em relação a alfa-hélice, e ligações de looping ligando estas entre si (b); e a do domínio bZIP tem grande quantidade de regiões de looping com poucas folhas-beta interligadas por estas e sem presença de alfa-hélices (c). De acordo com Lima (2015), a forma terciária revela muitas informações importantes, como a possibilidade de identificar quais resíduos estão mais internos, no núcleo proteico, ou mais na superfície, expostos a interações, além de sua composição molecular com a presença de folhas-beta, alfa-hélice e regiões de looping para caracterizá-la, uso em comparação estrutural onde pode-se identificar sítios funcionais, evoluções e mutações relacionadas a este, e função proteica a partir da sua conformação (De Lima, 2015.
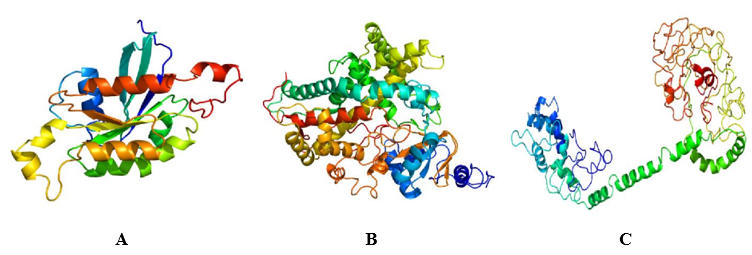
Figura 1 – Análise tridimensional da proteína dos domínios Rop_like. P450, bZIP, respectivamente, A, B e C.
Ao analisar a Figura 2 (a,b,c), do domínio Rop_like, P450 e bZIP, todas retratam a área superficial mais em azul, mostrando que esta tem boa solubilidade em meio aquoso, confirmando previsão obtida quantitativamente pelo teste de GRAVY, e adicionando à esta última análise, o fato de se poder observar quais áreas as proteínas apresentam o perfil mais hidrofílico em sua estrutura.
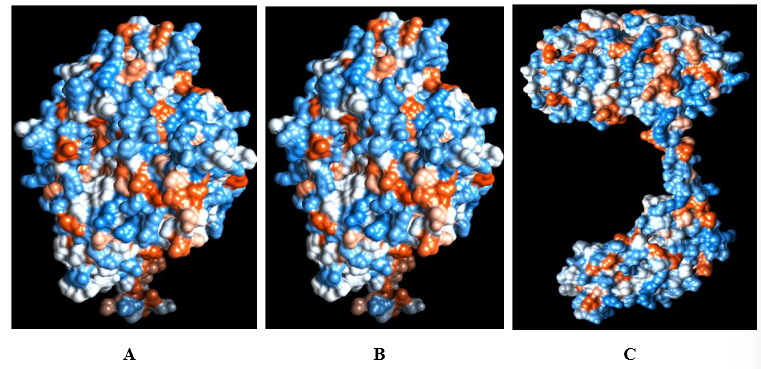
Figura 2 – Perfil de hidrofobicidade em gradiente de cor, regiões em azul mais hidrofílicas e em vermelho mais hidrofóbicas na proteína dos domínios Rop_like. P450, bZIP, respectivamente, A, B e C.
Como etapa de validação do modelo tridimensional das proteínas, o gráfico Ramachandran, mostra as conformações estruturais da alfa-hélice e beta-folha, de forma a mostrar a angulação torcional, phi e psi, da proteína, analisando se esta estabeleceu uma posição favorável à sua estabilidade e atividade biológica, no gráfico estas regiões positivas encontramse dentro das áreas azuis, as áreas ao redor destas são instáveis e não favoráveis a proteína (Da Silva, 2010). Válido, enfatizar, que há exceções para glicina e prolina quanto as áreas de permissibilidade dentro do gráfico, devido a uma maior flexibilidade e rigidez, respectivamente, além disso, para que a resolução da proteína esteja qualitativamente satisfatória, o gráfico deve apresentar valor igual ou maior que 90% de seus resíduos em áreas permissivas (Morris, et al., 1992).
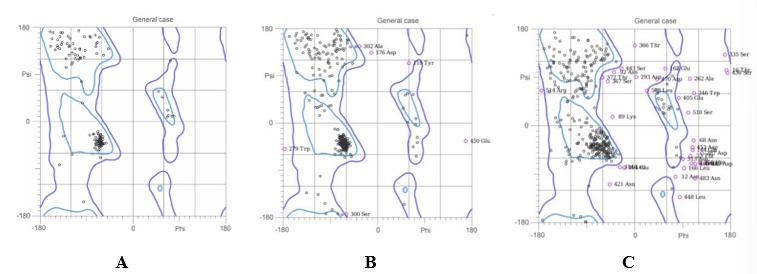
Figura 3 – Gráfico de Ramachandran da proteína dos domínios Rop_like. P450, bZIP, respectivamente, A, B e C.
Atentando-se ao resultado obtido pelo Molprobity, Figura 3, é possível concluir que para a proteína do domínio Rop_like todos resíduos de aminoácidos estão em conformações favoráveis mostrando que estas estão em equilíbrio, estáveis dentro das áreas azuis (a); para a do domínio P450 vê-se que os resíduos de aminoácidos estão em conformações favoráveis mostrando que estas estão em equilíbrio, estáveis dentro das áreas azuis, exceto por cinco resíduos: a alanina na posição 302, aspartato – 176, tirosina – 118, serina – 300 e glutamato – 450 (b); e do domínio bZIP, a maioria dos resíduos de aminoácidos estão em conformações favoráveis mostrando que estas estão em relativo equilíbrio, estáveis dentro das áreas azuis, apesar de grande quantidade de resíduo nas áreas brancas, não favoráveis (c), mostrando como modelos com melhor resolução, o (a) seguido pelo (b) e (c), respectivamente, de acordo com a quantidade de resíduos em conformações favoráveis.

Figura 4 – Análise do Z-score da proteína dos domínios Rop_like. P450, bZIP, respectivamente, A, B e C.
Como ferramentas estatísticas de refinamento qualitativo da validação da proteína, o gráfico de Z-score e o de energia dos scores, mostram a energia conformacional dos resíduos de aminoácidos e sua energia conformacional, a qual espera-se que seja negativo e esteja na faixa azul do gráfico (Jimenez, 2019), mostrando a qualidade e confiabilidade do modelo da estrutura 3D (De Oliveira, Sales, Freire, 2020) a partir de uma média de desvio padrão, no Zscore é através o valor que temos comparativamente a faixa já encontradas em proteínas nativas com o tamanho similar, e o gráfico de energia dos scores, faz a mesma coisa, só que não com a média geral, e sim a partir de fragmentos da sequência total, sendo o mais escuro o com intervalo menor 10 resíduos, e mais claro com hiato maior, 40 resíduos (Data Science, 2020; Prosa-Web, 2021).
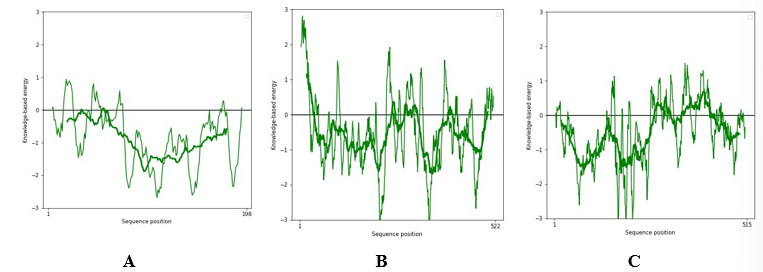
Figura 5 – Gráfico de energia dos scories da proteína dos domínios Rop_like. P450, bZIP, respectivamente, A, B e C.
Na Figura 4 (a), domínio Rop_like, nota-se que o gráfico Z-score encontra-se na região azul mais escura e negativa no gráfico no valor de -6,07, já na Figura 5 (a), além da energia mostrar-se favorável, pois está praticamente toda abaixo da linha preta do Z-score, fora um mínimo trecho que está sob a linha, sendo a com intervalo menor, verde claro, apresentando variação maior em relação à linha do Z-score, como esperado. Para a Figura 4 (b), domínio P450, o ponto preto, referente a média total do desvio padrão da proteína, se encontra na região azul e negativa no gráfico no valor de -7,18, na Figura 5 (b), apesar da posição da sequência inicial e final estar bem acima da linha preta do Z-score, o restante da sequência encontra-se próxima a linha ou bem abaixo, mais aconselhado, similar para as duas janelas de intervalo. Já para o domínio bZIP, Figura 4 (c), o valor do desvio padrão encontra-se na região azul, próxima da área branca, negativa no gráfico no valor de -4,66, na Figura 5 (c), vê-se que no início o modelo está favorável, abaixo da linha preta do Z-score, a outra metade da posição da sequência tem intervalos acima, e em seu final volta a se encontra negativa, apresentando uma leve atenuada na análise com intervalos maiores, em verde escuro.
Pode-se constatar a partir da análise estrutural, Figura 3,4,5, com o gráfico de Ramanchadran e Z-score que, qualitativamente, a do domínio Rop_like é mais estavelmente promissora, seguida da P450 e bZIP, respectivamente. Já no estudo tridimensional e nas análises de hidrofobicidade, Figura 1, 2, as três proteínas não apresentaram diferenças significativas, sobre o bom nível de dissolução em meio aquoso, mostrando maior facilidade de manipulação experimental devido a esta característica química, apesar de estruturalmente terem tamanho, sequência e domínios diferentes, ainda apresentam mesma função relacionada a tolerância, resistência ao estresse salino, em comum.
Após, esta etapa, foi desenhado os respectivos primers específicos das sequências estudadas para viabilização do estudo experimental relacionado ao estresse salino nas espécies do algodoeiro (Tabela 3 e 4), dentre as quais é possível ver tanto a sua sequência, o sentido desta, e outras informações importantes para viabilização do trabalho em bancada durante a execução do PCR para que se encontre a região de interesse no gene que traduzirá as proteínas dos domínios Rop_like, P450 e Hsf8. Além da validação destes desenhos, apresentados pelos fatores físicos RInk, deltaS, deltaG, deltaH, relacionados com as propriedades termodinâmicas de entropia e energia de cada primer apontado. E o domínios de sequências conservadas é ponderada para predição de função (Gossani, 2014).
Ao observar os intervalos funcionais das sequências de cada proteína de interesse, foi feito cortes nestes respectivos intervalos: Rop_like na região 8 – 180; P450 no local 3 – 522; HSF8 no intervalo 167 – 215, para um desenho de primer mais específico possível. Assim, após o corte da sequência, pode-se analisar os desenhos dos primers a partir de três fatores em relação a sua qualidade: tamanho (preferível de 18 a 28 bases, mais específicos e estáveis); temperatura de anelamento (entre 50 a 60°C é o ideal); a porcentagem de guaninas e citosinas que possui (é desejado o intervalo de 45 a 55%). Todos estes aspectos tornam o primer específico, seguro e eficiente (De Mendonça, 2016). Por estes critérios, o primer mais promissor de cada domínio é Rop_like – 3, P450 – 1 e bZIP – 1, Tabela 3.
Tabela 3 – Primers das proteínas Rop_like, P450 E bZIP.
| Primers | Tamanho | Sequência 5’→3’ | Sentido | Temperatura de anelamento (°C) | %CG (Citosina e Guanina) | RInKCal/ (°K*mol) | DeltaS Kcal.mol | DeltaG Kcal.mol | DeltaH Kcal.mol | Plataforma |
| Rop_like – 1 | 20 | TTAGGTCTTTGGGACACGGC | Positivo | 59,96 | 55 | 33,404 | 533,4 | 26,9 | 208,09 | PrimerBlast – NCBI |
| 20 | TTCTCATGGCTTGCTCGACT | Negativo | 59,39 | 50 | ||||||
| Rop_like – 2 | 19 | TTGGAGATGGAGCTGTTGG | Positivo | 59,8 | 52 | 33,404 | 515,6 | 24,2 | 199,95 | Primer3Plus |
| 20 | CCGTGTCCCAAAGACCTAAG | Negativo | 59,6 | 55 | ||||||
| Rop_like-3 | 20 | AGTTGGAGATGGAGCTGTTG | Positivo | 57,9 | 50 | 33,404 | 539,5 | 25,1 | 208,44 | Primer3Plus |
| 20 | CCGTGTCCCAAAGACCTAAG | Negativo | 59,6 | 55 | ||||||
| P450 – 1 | 20 | CCGGTGCATGGCCAATAATC | Positivo | 59,69 | 55 | 33,404 | 539,5 | 26,6 | 208,99 | PrimerBlast – NCBI |
| 20 | TCCAAGTCTCCCAACGCTTC | Negativo | 59,97 | 55 | ||||||
| P450 -2 | 20 | TGGTGGTGAATTCGAGTGAG | Positivo | 59,7 | 50 | 33,404 | 537,1 | 25,6 | 207,08 | Primer3Plus |
| 20 | TTGAGCTGATCGATCCTGTG | Negativo | 59,9 | 50 |
| bZIP – 1 | 19 | GGCGGAGAATCTCAAACGA | Positivo | 61,3 | 52,6 | 33,404 | 512,5 | 25,3 | 197,72 | Primer3Plus |
| 20 | TAGCATCGGTAGCAGGATCA | Negativo | 59,4 | 50 | ||||||
| bZIP – 2 | 19 | GGCGGAGAATCTCAAACGA | Positivo | 61,3 | 52,6 | 33,404 | 512,5 | 25,3 | 197,72 | Primer3Plus |
| 20 | GGTAGCAGGATCATCCACCA | Negativo | 60,9 | 55 |
Tabela 4 – Primers degenerados das proteínas.
| Primers | Tamanho | Sequência 5’→3’ | Sentido | Temperatura de anelamento(°C) | %CG (Citosina e Guanina) | RInKCal/ (°K*mol) | DeltaS Kcal.mol | DeltaG Kcal.mol | DeltaH Kcal.mol | Qualidde (%) | Plataforma |
| Primer 1a | 23 | AGATGGAGCTGTTGGCAAGACTT | Positivo | 54,9 | -29,1 | 33,404 | 478 | 30,1 | 183,4 | 60 | FastPCR |
| 23 | CACCTCTGTAGCTCAATGGC CTT | Negativo | 55,3 | -29,1 | |||||||
| Primer 1b | 22 | GAGCTGTTGGCAAGACTTGCAT | Positivo | 54,9 | -28,3 | 33,404 | 457,1 | 29 | 175,8 | 60 | FastPCR |
| 23 | CACCTCTGTAGCTCAATGGCCTT | Negativo | 55,3 | -29,1 | |||||||
| Primer 1c | 23 | GGCACAACTGTGAACTTAGGTCT | Positivo | 53,6 | -28,2 | 33,404 | 491,4 | 29,3 | 186,7 | 58 | FastPCR |
| 22 | AGACATCTGCACCTCTGTAGCT | Negativo | 53,5 | -27,5 | |||||||
| Primer 1d | 20 | GGCAAAGCTCCACCTGTAGC | Positivo | 54,4 | -26,4 | 33,404 | 429,7 | 27,1 | 165,4 | 60 | FastPCR |
| 21 | TTGCGCATTTCACGCCAGTAT | Negativo | 54,2 | -27,5 |
Já no desenho do primer degenerado, foi utilizado as sequências cortadas das três proteínas e as usou para gerar um primer mais “geral”, pois tem o objetivo de cobrir todas regiões possíveis referentes a síntese das proteínas de interesse, no gene. Como parâmetros para análise de qualidade deste primer, o intervalo de tamanho é de 22 a 24 pares de bases, temperatura de anelamento aproximadamente a 56°C, e porcentagem de citosina e guanina próxima ou superior a 50% (Malone, et al., 2006). Assim, de acordo com os resultados que obtidos, pode-se ver uma baixa porcentagem de citosina e guanina, nos desenhos obtidos, a temperatura está ligeiramente abaixo de 56°C, assim como a quantidade de pares de bases. Com base nestes parâmetros, o primer 1c tem as características mais adequadas, Tabela 4.
4.CONSIDERAÇÕES FINAIS
O estudo in silico, possibilitou a partir do comportamento da planta com contato salino, a triagem de três proteínas com função relacionada a este fator abiótico, promovendo estudo físico-químico e estrutural destas para compreensão do comportamento das proteínas e qualidade conformacional de sua estrutura. Além da análise de domínios conservados das sequências para o desenho de primers mais estáveis, seguros, específicos e validação de sua funcionalidade, os quais atestam a fundamental importância destas ferramentas computacionais para realização experimental da identificação do éxon de interesse no gene com os marcadores moleculares e desenvolvimento da pesquisa de manipulação genética à salinidade em algodoeiro.
5.REFERÊNCIAS
AMORIM, J. R. A.; et al. Determinação da eficiência de uso da água na parcela de irrigação, no perímetro irrigado Califórnia, em Sergipe. Aracaju: Embrapa Tabuleiros Costeiros. 2010. 6 p.
BLANCO, B.S, Gossipol e fatores antinutricionais da soja. In: Toxicologia Aplicada á Medicina Veterinária, H. S. Spinosa, S. L. G´orniak, and J. P. Neto, Eds. Manole, Barueri, p. 531–545, 2008.
BLANCO, F. F.; et al. Crescimento e produção de cultivares de algodão sob irrigação com déficit. In:Congresso Brasileiro de Algodão & I Cotton Expo, VIII, 2011, São Paulo. Anais […]. Campina Grande: Embrapa Algodão, 2011, p; 1753-1760.
BRUM, I. J. B. Ferramentas de bioinformática para proteômica. 2007. Dissertação (Mestrado em Biologia Funcional e Molecular) – Universidade Estadual de Campinas, Campinas, 2007, 83p. Disponível em: <http://repositorio.unicamp.br/jspui/bitstream/REPOSIP/314739/1/Brum_ItarajuJuniorBaracuhy_M.p df>. Acesso em: 04 de mai. de 2023.
CORREIA, R. C.; et al. A região semiárida brasileira. In: VOLTOLLINI, T. V. Produção de caprinos e ovinos no Semiárido. Petrolina: Embrapa Semiárido, 2011, p. 21-48.
DA SILVA, A. A. Adubação nitrogenada em cobertura na cultura do algodão, Vilhena-RO.2020. Monografia (Bacharel em Agronomia) – Faculdade da Amazônia, Vilhena, 2020, 31p.Disponível em: <http://repositorio.famaro.com.br/bitstream/123456789/172/1/TCC%20FINAL%20ANDERSON.pdf>. Acesso em: 24 de abr. de 2021.
DA SILVA, K. N. Modelagem comparativa da metaloprotease vmbmp de Bothrops moojeni por homologia estrutural. 2010. Dissertação (Mestrado em Biotecnologia e Recursos Naturais) – Universidade do Estado do Amazonas, Manaus, 2010, 103p. Disponível em:<https://pos.uea.edu.br/data/area/titulado/download/18-4.pdf>. Acesso em: 15 de mai. 2023.
DE ARRUDA JÚNIOR, R. G. Temperatura de melting: um estudo comparativo. 2010.Monografia (Bacharel em Ciência da Computação) – Universidade Federal de Mato Grosso do Sul, Campo Grande, 2010, 55p. Disponível em: <http://www.facom.ufms.br/wpcontent/uploads/2015/11/Temperatura-de-Melting.pdf>. Acesso em: 05 de mai. de 2023.
DE AZEVEDO, P. V.; SILVA, F. D. DOS S. Risco climático para o cultivo do algodoeiro na região nordeste do Brasil. Revista Brasileira de Meteorologia, v. 22, n. 3, p. 408-416, 2007.
DE LIMA, E. B. Detecção de subfamílias proteicas isofuncionais utilizando integração de dados e agrupamentos espectral. 2015. Tese (Doutorado em Bioinformática) – Universidade Federal de Minas Gerais, Belo Horizonte, 2015, 194p. Disponível em:<http://www.pgbioinfo.icb.ufmg.br/defesas/61D.PDF>. Acesso em: 06 de mai. de 2023.
DE MENDONÇA, I. B. Acompanhamento das principais técnicas biomoleculares no laboratório de biotecnologia e biologia molecular e no núcleo de genômica e bioinformática. Relatório de Estágio Obrigatório. Fortaleza, 2016.
DE OLIVEIRA, C.; SALES, L. S.; FREIRE, J. E. DA C. Modelagem estrutural de uma nova quitinase (GH19) de melão, e análise de docking molecular com N-acetil-β(1-4)-D-glicosamina. Revista Arquivos Cientpificos (IMMES), Macapá, v. 3, n. 1, p. 162 – 171, 2020.
DE SENA, L. M. M. Conheça e Preserve a Caatinga – O Bioma Caatinga. Associação Caatinga, Fortaleza, v. 1, 54p., 2011.
DE SOUSA, L. B. O algodoeiro: alguns aspectos importantes da cultura. Revista Verde de Agroecologia e Desenvolvimento Sustentável, Mossoró, v. 5, n. 4, p. 19-26, dez. 2010.
FINKELSTEIN, R. R.; GAMPALA, S. S. L.; ROCHA, C. D. Abscisic acid signaling in seeds and seedlings. The Plant Cell, v. 14, n. 1, p. S15 – S45, 2002.
GOSSANI, C. M. D. Evolução de genes que conferem resistência a múltiplas drogas em fungos. 2014. Dissertação (Metrado em Biociências e Biotecnologia) – Universidade Estadual do Norte Fluminense Darcy Ribeiro, Compos dos Goytacazes, 2014, 59p. Disponível em:<https://uenf.br/posgraduacao/biociencias-biotecnologia/wpcontent/uploads/sites/12/2020/12/dissertacao_cristiani_gossani.pdf>. Acesso em: 06 de mai. de 2023.
GUERRA, R. A. T.; et al. Ciências biológicas: Cadernos CB Virtual 1. João Pessoa: Ed. Universitária, 2011. 516p.
HOFFMANN NETO, E. G. Tecnologias para a produção de forragem em solos de cerrado do Brasil central. 1999. Monografia (Bacharel em Agronomia) – Universidade Federal de Santa Catarina, Florianópolis, 1999, 67p. Disponível em: <https://repositorio.ufsc.br/bitstream/handle/123456789/117714/138632.pdf?sequence=1>. Acesso em: 06 de abr. de 2023.
JIMENEZ, H. J. Análise Molecular in silico e Palinológica de espécies de Amaryllidaceae J. ST. – HIL. 2019. Tese (Doutorado em Agronomia) – Universidade Federal Rural de Pernambuco. Recife, 2019, 111p. Disponível em: <http://www.tede2.ufrpe.br:8080/tede/handle/tede2/8381>. Acesso: 04 de abr. de 2024.
KALENDAR, R.; LEE, D.; SCHULMAN, A. H. FastPCR Software for PCR, In Silico PCR, and Oligonucleotide Assembly and Analysis. In: Valla S., Lale R. (eds) DNA Cloning and Assembly Methods. Methods in Molecular Biology (Methods and Protocols). Humana Press, Totowa, v. 1116, p. 271 – 302, 2014.
KIBBE, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Research. v. 35, p. 43 – 46, 2007.
Levantamento Sistemático da Produção Agrícola – Tabela 1618: Área plantada, área colhida e produção, por ano da safra e produto das lavouras. IBGE, 2021. Disponível em: <https://sidra.ibge.gov.br/tabela/1618#resultado>. Acesso em: 06 de abr. de 2021.
LIMA, J. E. F. W. Situação e perspectivas sobre as águas do cerrado. Ciência Cult., São Paulo, v. 63, n. 3, p. 27-29, 2011.
MALONE, G.; et al. Prospecção de genes em bibliotecas de cDna. Revista Brasileira de Agrociência, Pelotas, v. 12, n. 1, p. 7 – 13, mar. 2006.
MARQUES, M. R. F. Bioquímica. 1 ed. Florianópolis: Universidade Federal de Santa Catarina, 2014, 182p.
MATTOS, L. C.; et al. A saga do algodão no semiárido nordestino: histórico, declínio e as perspectivas de base agroecológica. Revista Desenvolvimento e Meio Ambiente, UFPR, v. 55, p. 556-580, dez. 2020.
MORAES, C. S.; et al. Série em biologia celular e molecular: métodos experimentais no estudo de proteínas. Rio de Janeiro: Fiocruz, 2013. 84p.
MORRIS, A. L.; et al. Stereochemical quality of protein structure coordinates. Proteins: Structure, Function, and Bioinformatics, v. 12, p. 345-364, 1992.
MOTA, A. F. Cultivo do algodoeiro irrigado com águas salinas de acordo com a fase de desenvolvimento da cultura. 2014. Dissertação (Mestrado em Manejo de Solo e Água) – Universidade Rural do Semi-Árido, Mossoró, 2014, 64p. Disponível em: <https://ppgmsa.ufersa.edu.br/wp-content/uploads/sites/59/2014/10/Disserta%C3%A7%C3%A3oANDYGLEY-FERNANDES-MOTA.pdf>. Acesso em 07 de abr. de 2024.
PEREIRA, E. R. DE L. Tolerância de genótipos do algodão colorido ao estresse salino. 2012.
Dissertação (Mestrado em Ciências Agrárias) – Universidade Estadual da Paraíba, Campina Grande,2012, 105p. Disponível em: <http://pos-graduacao.uepb.edu.br/ppgca/download/outros_documentos/DISSERTA%C3%87%C3%83O%20%20ELIZANDRA%20RIBEIRO%20DE%20LIMA%20PEREIRA%20-%202012.pdf>. Acesso em 05 de abr. de 2021.
QUEIROZ, J. A. DA S.; et al. Desenho e Validação de Primers In Silico para Detecção do Vírus Sincicial Respiratório Humano. Revista Fimca, v. 4, n.1, p. 17 – 30, dez. de 2017.
SALDANHA, C. I.; BELO, M. A. DE A. Produção Animal Universidade Brasil – Fossipol: Toxicidade dos produtos do algodão. Universidade Brasil – Boletim técnico, Descalvado – SP, 25p., dez. 2016.
SOARES FILHO, W. DOS S.; et al. Melhoramento genético e seleção de cultivares tolerantes à salinidade. In: Manejo da salinidade na agricultura: estudos básicos e aplicados, GHEY, H. R.; DIAS, N DA S., LACERDA, C. F. DE; GOMES FILHO, E. INCTSal, Fortaleza, p.259-274, 2016.
VERLI, H. Bioinformática da biologia à flexibilidade molecular. 1 ed. São Paulo: Sociedade Brasileira de Bioquímica e Biologia Molecular – SBBq, 2014, 282p.
VILHENA, A. P. Brimer: Um sistema web para gerenciamento de primers para o laboratório de tecnologia biomolecular da UFPA. 2015. Monografia (Bacharel em Sistemas de Informação) – Universidade Federal do Sul e Sudeste do Pará, Marabá, 2015, 70p. Disponível em:<https://faceel.unifesspa.edu.br/images/works/TCC/2015/BRIMER-UM-SISTEMA-WEB-PARA-GERENCIAMENTO-DE-PRIMERS-PARA-O-LABORATORIO-DE-TECNOLOGIA-BIOMOLOCULAR-DA-UFPA.pdf>. Acesso em: 28 de abr. de 2021.
WANG, Z.; et al. Caracterização de todo o genoma de fatores de transcrição bZIP e seus padrões de expressão em resposta à seca e estresse salino em Jatropha curcas. International Journal of Biological Macromolecules, mai. de 2021.
YAN, Q.; et al. GmCYP82A3, um gene da família do citocromo P450 da soja envolvido na via de sinalização de ácido jasmônico e etileno, aumenta a resistência das plantas a estresses bióticos e abióticos. Journals Plos One, set. de 2016.
YE, J.; et al. Primer-BLAST: Uma ferramenta para projetar primers específicos para a reação em cadeia da polimerase. BMC Bioinformática, v. 13, n. 134, p. 1 – 11, 2012.
1 Universidade Federal de Campina Grande, Centro de Desenvolvimento Sustentável (UFCG-CDSA), Rua Luiz Grande, S/N, Frei Damião, Sumé, Paraíba, CEP 58540-000;
2 Universidade Federal Rural de Pernambuco (UFRPE).
