TOPOLOGICAL ANALYSIS OF NK1-ANTAGONIST 2D INTERACTION
REGISTRO DOI: 10.5281/zenodo.7786628
João Victor Carvalho Magalhães¹
Valter Luiz da Conceição Gonçalves²
Rodrigo da Silva Bitzer³
Resumo
A substância P é um neuropeptídeo que pertence à família das taquicininas e pode ser encontrada tanto em vertebrados como em invertebrados. São produzidas a partir da hidrólise da proteína protaquicinina-1 (PPT) e ligam-se, preferencialmente, ao receptor SPR (NK1).
Foi utilizada a base de dados UniProtKB para obter informações estruturais da proteína e encontradas quatro estruturas cristalográficas. O programa utilizado para fazer o mapeamento topológico foi o LigPlot, que realiza representações esquemáticas em duas dimensões (2D) em complexos proteína/ligante de arquivos do PDB (Protein Data Bank).
Como resultados, foram encontrados modelos que dispusessem valores de RMSD menores que 3,4 Å, deixando a estrutura equivalente, e o alto valor da resolução das estruturas implicou em resultados ainda melhores.
Palavras chave: Topologia, NK1, antagonista.
Abstract
Substance P is a neuropeptide that belongs to the tachykinin family and can be found in both vertebrates and invertebrates. They are produced from the hydrolysis of protachinin-1 protein (PPT) and preferentially bind to the SPR receptor (NK1).
The UniProtKB database was used to obtain protein structural information and four crystallographed structures were found. The program used for topological mapping was LigPlot, which performs two-dimensional (2D) schematic representations on protein / ligand complexes of PDB (Protein Data Bank).
As a result, models with RMSD values lower than 3.4 Å were found, leaving the equivalent structure, and the high resolution value of the structures implied even better results.
Keywords: Topology, NK1, antagonist.
Introdução
Substância P
Substância P (SP) é um neuropeptídeo originalmente isolado de cérebro e intestino de equinos¹. Ela pertence à família das taquicininas, peptídeos encontrados tanto em vertebrados como em invertebrados, que estimulam neurônios, evocam respostas comportamentais, são potentes vasodilatadores e secretagogos, e participam (direta ou indiretamente) da contração de muitos músculos lisos². Especificamente, a SP é um neuromodulador envolvido em processos inflamatórios, na geração de vômito, na ansiedade e na nocicepção (resposta a estímulos aversivos/agressivos)³.Em humanos, as principais taquicininas são: substância P (SP), neurocinina A (também conhecida como substância K) (NKA), neuropeptídeo K (NPK) e neuropeptide γ, codificadas pelo gene TAC1; e neurocininaB (NKB), codificada pelo gene TAC34. Os membros da família das taquicininas estão distribuídos por todos os tecidos. No sistema nervoso, a SP é encontrada na raiz posterior da medula espinhal, na substância negra e na amígdala5. Além disso, há evidências de que a SP seja expressa nos gânglios da raiz dorsal (DRG) e corno dorsal, mas não no corno ventral, já que ela está presente em fibras aferentes primárias6.
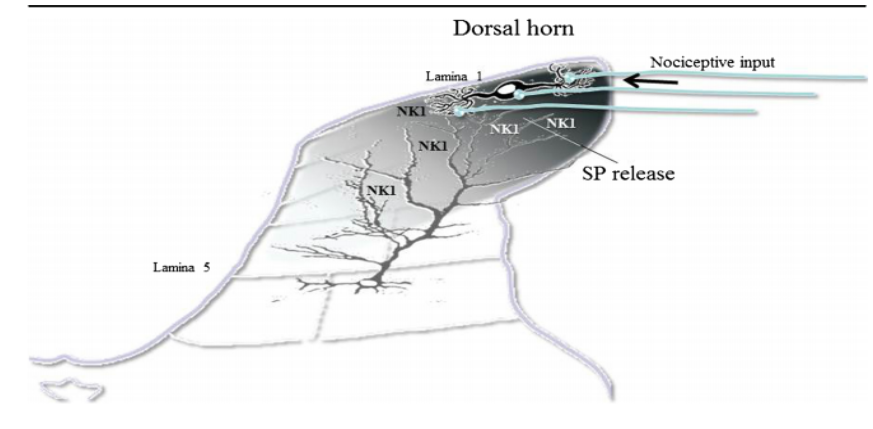
Figura – 1: Fonte W. Zieglgänsberger, Substance P andpainchronicity, 2018.
A SP é produzida a partir da hidrólise da proteína protaquicinina-1 (PPT), que, por sua vez, consiste num derivado proteolítico da preprotaquicinina-1, proteína precursora codificada pelo gene TAC1 em humanos, identificado no início do século XX7. A SP exibe 11 resíduos de aminoácidos: RPKPQQFFGLM. Assim como ocorre com outras taquicininas, ela apresenta a sequência C-terminal Phe-X-Gly-Leu-Met, onde X = Phe. A SP é sintetizada no ribossomo como uma proteína e transportada até as terminações nervosas, onde será convertida através de um processo enzimático e armazenada, na forma ativa, em vesículas8. Suas ações são mediadas pelo receptor para substância P (SPR), anteriormente conhecido como receptor de taquicinina (ou neurocinina) 1 (NK1). A SP liga-se preferencialmente ao receptor SPR (NK1), mas também pode se ligar, com menor afinidade, aos receptores NK2 e NK39.
As ações da SP não se limitam apenas à transmissão e integração de sinais nociceptivos; ela também controla suas consequências, como a depressão por estarem expressas em áreas relacionadas ao stress e ao medo (córtex frontal, hipotálamo, amígdala e hipocampo)9. Desse modo, a SP ajuda a expandir o processo inflamatório para o campo de fibras nervosas do tipo C, adjacentes à área lesada, estabelecendo, assim, a hiperalgesia tanto primária quanto secundária10.
A associação da SP com o seu receptor NK¹ induz o aumento da permeabilidade vascular e subsequente extravasamento de proteínas plasmáticas11, a degradação de mastócitos acaba aumentando esses processos vasculares e, consequentemente, ativando os nociceptores12. Essas células possuem receptores de SP, que podem produzir citocinas, levando, assim,à produção de mediadores inflamatórios, como histamina, NO, citocinas (IL-1, IL-6) e cininas13. Tendo em vista o exposto, a SP pode ser considerada um importante mediador na inflamação neurogênica e pode ser associada com a hiperalgesia14. Sendo assim, a SP representa um alvo importante para esclarecer processos fisiopatológicos relacionados à dor, a fim de buscar formas de antagonizar o receptor SPR e minimizar as consequências do efeito dessa substância nos tecidos lesionados, mostrando através de dados estatísticos e diagramas em 2D o mapeamento do ligante/receptor para a busca de possíveis modelos.
Receptor SPR (NK1)
SPR é um típico receptor associado à proteína G (GPCR) pertencente à classe A. Após ser sensibilizado, ele ativa a fosfolipase C, culminando na mobilização intracelular de íons Ca2+ 15. O receptor SPR é expresso, predominantemente, em neurônios do SNC, em células musculares lisas, células endoteliais, fibroblastos e em células inflamatórias16.
Receptor SPR (NK1) como alvo terapêutico
A ligação de SP no seu receptor tem causado implicação na regulação de diversos processos fisiopatológicos como: dor, inflamação, salivação, depressão, resposta ao estresse, êmese, entre outros17. O bloqueio desse receptor é um importante alvo para tais desordens fisiológicas citadas acima18.
Dentre todas essas patologias ligadas ao receptor SPR NK1 foi sujeito apenas ao desenvolvimento clínico no tratamento de vômitos induzido por quimioterápicos, originando assim através de ensaios clínicos o primeiro antagonista registrado em agências reguladoras chamado de Aprepitant e Netupitan19.
Métodos
Usou-se a base de dados UniProtKB(https://www.uniprot.org/) para obter informações adicionais sobre sua expressão, função e estrutura do SPR NK1 (entrada P25103). A base de dados UniProtKB informava que três fragmentos do receptor NK1 tinham as suas estruturas em solução determinadas por RMN, que são: PDB ID 2KS9, 2KSA e 2KSB.
Em seguida, foram encontradas quatro estruturas de raios X em três dimensões (3D) disponíveis para o receptor SPR humano (hSPR) no Protein Data Bank (PDB): PDB ID 6E59 (complexo com o antagonista L760735; resolução de 3,4 Å), 6HLL (complexo com o antagonista CP-99,994; resolução de 3,3 Å), 6HLO (complexo com o antagonista Aprepitant; resolução de 2,4 Å) e 6HLP (complexo com o antagonista Netupitant; resolução de 2,2 Å).O valor máximo de RMSD (root mean square deviation) entre essas estruturas é de 1,22 Å, indicando que a interação entre o receptor SPR e um antagonista preserva a conformação inativa do receptor.
Para análise topológica das 4 estruturas citadas neste trabalho, foi utilizado software LigPlot+ v.1.4 (https://www.ebi.ac.uk/thornton-srv/software/LigPlus/). O programa apresenta as distâncias interatômicas de interação entre o ligante e os aminoácidos por meio do tipo de ligação de hidrogênio e, também, identifica formas distintas de interação, como coordenação com metais, ligações de hidrogênio e interações hidrofóbicas realizando essas representações esquemáticas em 2 dimensões (2D) em complexos proteína/ligante através de arquivos do ID de proteínas no PDB em três dimensões (3D). O programa em si estipula padrões de análises para obter tais resultados, sendo eles: raio dos átomos do ligante até 0.330 Å, raio dos átomos não ligante até 0.330Å, raio dos átomos de água até 0.400Å, raio dos resíduos hidrofóbicos 1,150Å, a máxima distância e um grupo aceptor e um hidrogênio 2,70 Å, a máxima distância e um grupo doador e um hidrogênio 3,35 Å.
Através de dados estatísticos feitos pelo Excel, foi elaborada duas tabelas mostrando a quantidade de aminoácidos envolvidos em cada estrutura e o percentual dos mesmos que se repetem.
Resultados e Discussões
Resultados
Foi elaborado um diagrama com as representações esquemáticas das interações envolvendo ligante/receptor pelo software LigPlot+.
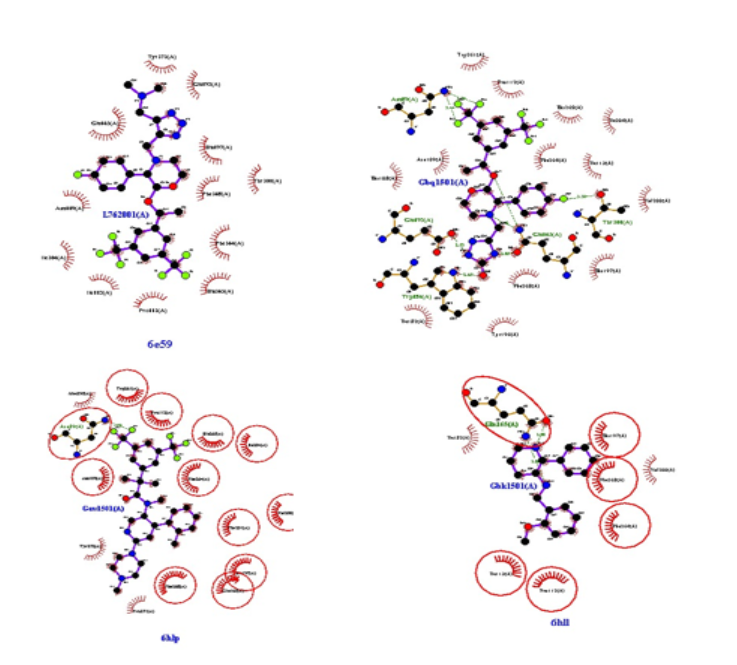
Figura 2 – Imagem elaborada pelo autor
Em seguida, criou-se uma tabela mostrando os aminoácidos envolvidos em cada interação Ligante/receptor com a intenção de elucidar cada um deles presentes e o número de vezes que se repetem no modelo.

Tabela 1 – Microsoft Excel.
Após uma análise dos modelos gerados pelo software LigPlot+, foram encontrados sete aminoácidos que têm relação com as quatro estruturas cristalográficas. Junto com as proteínas, dois desses aminoácidos se repetiram, tendo uma percentagem de 28,6% cada. São eles: Glutamina 165 e Asparagina 89. Já os outros três (Ácido Glutâmico 193, Treonina 201 e Triptofano 184) apareceram apenas uma única vez. O software não conseguiu identificar uma interação entre o ligante co-cristalizado com a proteína (ID:6E59), podendo ser decorrente do alto valor de resolução.
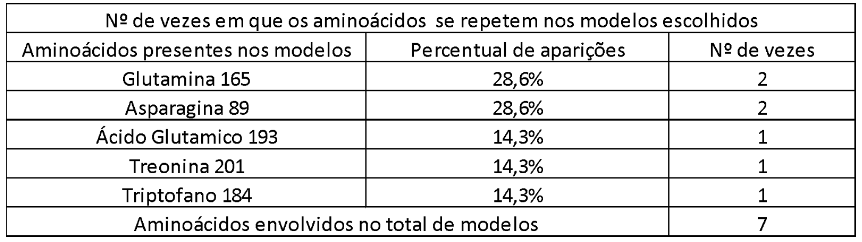
Tabela 2 – Microsoft Excel.
Discussão
O receptor SPR é expresso em neurônios do SNC, em células musculares lisas, células endoteliais, fibroblastos e em células inflamatórias19. Sendo seletiva para SP que, ao ligar-se, desencadeia uma série de processos inflamatórios13.
Através do software LigPlot, foram realizados diagramas de interações ligante/receptor das proteínas ID: 6E59, 6HLL, 6HLO e 6HLP. Onde essas proteínas foram mapeadas e catalogadas através da tabela 1, que mostra os aminoácidos envolvidos em cada interação ligante/receptor, exceto 6E59 que não apresenta interação. As demais proteínas apresentaram interações com os resíduos de aminoácidos das mesmas.
A tabela 2 mostra o percentual de aminoácidos que se repetem em cada estrutura, mostrando que a Glutamina 165 e Asparagina 89 são consideradas essenciais para um bom antagonista do receptor SPR.
Como mostrado na figura 2 através de representações em 2D das interações do modelo 6HLL, Gln165 mantém interações hidrofóbicas com uma distância de 2,88 Å entre os resíduos e o ligante. O modelo 6HLO, mantém interações hidrofóbicas com os resíduos Asn89 (mantendo uma distância de 2,94 Å e 3,44 Å de cada átomo de Flúor); Glu193 (mantendo distância de 2,53 Å da ligação do Oxigênio com Nitrogênio); Trp184 (mantendo distância de 2,65 Å da ligação do Nitrogênio com Oxigênio); Gln165 (mantendo distância de 3,08 Å da ligação do Nitrogênio com Oxigênio, 3,46 Å da ligação do Nitrogênio do aminoácido com o Nitrogênio do ligante, 2,85 Å da ligação do Oxigênio do aminoácido com o Nitrogênio do ligante); Thr201 (mantendo distância de 3,30 Å da ligação do Oxigênio do aminoácido com o Flúor do Ligante). O modelo 6HLP, mantém interação hidrofóbica com o resíduo Asn89 (mantendo uma distância de 3,01 Å da ligação do nitrogênio do aminoácido com o Flúor do ligante)20.
Desta forma, podemos predizer através de diagramas usados pelo software LigPlot+ o mapeamento topológico do receptor SPR de cada proteína descrita neste trabalho com os seus respectivos antagonistas complexados, mostrando também suas interações interatômicas com os seus principais aminoácidos citados acima.
Dado o valor de RMSD calculado após a superposição, qualquer valor acima de 3,5Å é considerado não equivalente. Sendo assim, com um valor de 1,22 Å como resultado, quatro estruturas são consideradas equivalentes21.
Com os resultados obtidos pode-se concluir que o modelo 6E59 não obteve nenhum tipo de interação apresentada pelo software, podendo ser por conta do alto valor de resolução encontrado. Já o modelo 6HLP apresentou uma interação do aminoácido com o ligante e os outros dois (6HLL e 6HLO) apresentam duas ligações de aminoácidos com os ligantes apresentados, tendo em vista que para se apresentar um bom padrão estrutural é necessário ter três aminoácidos essenciais e uma resolução abaixo de 2,0 Å
O fator limitante deste trabalho foi a resolução encontrada para as quatro estruturas cristalográficas sendo acima de 2,0Å, dificultando assim obter um bom resultado para os modelos propostos.
Referências
- V Euler US, Gaddum JH. An unidentified depressor substance in certain tissue extracts. J Physiol. 1931 Jun 6;72(1):74-87;
- Mantyh PW. Neurobiologyofsubstance P andthe NK1 receptor J ClinPsychiatry. 2002;63 Suppl 11:6-10;
- Minamino N, Kangawa K, Fukuda A, Matsuo H. Neuromedin L: a novel mammaliantachykininidentified in porcinespinalcord. Neuropeptides. 1984 Mar;4(2):157-66;
- Ribeiro-da-Silva A, Hökfelt T (2000) Neuroanatomical localisation of substance P in the CNS and sensory neurons. Neuropeptides 34(5):256–271;
- Lembeck (1953) ZurFrage der zentralenÜbertragungafferenter Impulse. III. Mitteilung. Das Vorkommen und die Bedeutung der Substanz P in den dorsalenWurzeln des Rückenmarks. Naunyn- Schmiedeberg’s Arch Exp PatholPharmakol219:S197–S213;
- Lembeck F, Holzer P (1979) Substance P as neurogenic mediator of antidromic vasodilation and neurogenic plasma extravasation. NaunynSchmiedeberg’s Arch Pharmacol 310(2):175–173;
- Lembeck F (2008) The archeology of substance P. Neuropeptides 42(4): 444–453;
- Pernow B (1953) Distribution of substance P in the central and peripheral nervous system. Nature 171(4356):746;
- Parent AJ, Beaudet N, Beaudry H, Bergeron J, Bérubé P, Drolet G, Sarret P, Gendron L (2012) Increased anxiety-like behaviors in rats experiencing chronic inflammatory pain. Behav Brain Res 229(1): 160–167;
- Otten U, Goedert M, Mayer N et al – Requirement of nerve growth factor for development of substance P-containing sensory neurones. Nature, 1980;287:158-159;
- Holzer P (1998). Neurogenic vasodilatation and plasma leakage in the skin. Gen Pharmacol 30: 5–11;
- Moriarty D, Selve N, Baird AW, Goldhill J. Potent NK1 antagonism by SR 140333 reduces rat colonic secretory response to immunocyte activation. Am J Physiol Cell Physiol. 2001 Apr;280(4):C852-8;
- Averbeck B, Reeh PW (2001). Interactions of inflammatory mediators stimulating release of calcitonin gene-related peptide, substance P and prostaglandin E(2) from isolated rat skin. Neuropharmacology 40: 416–423;
- Sacerdote P, Levrini L. Peripheral mechanisms of dental pain: the role of substance P. Mediators Inflamm. 2012;2012:951920. doi: 10.1155/2012/951920;
- Muñoz M, Rosso M, Casinello F, Coveñas R. Paravertebralanesthesia: how substance P and the NK-1 receptor could be involved in regional block and breast cancer recurrence. Breast Cancer Res Treat. 2010 Jul;122(2):601-3.doi:10.1007/s10549-010-0850-y;
- Lundy FT, Linden GJ (2004). Neuropeptides and neurogenic mechanisms in oral and periodontal inflammation. Crit Reviews Oral Biol Med 15: 82–98;
- Regoli D, Boudon A, Fauchere JL (1994). Receptors and antagonists for substance P and related peptides. Pharmacol Rev 46: 551–599;
- Garland AM, Grady EF, Payan DG, Vigna SR, Bunnett NW (1994). Agonist-induced internalization of the substance P (NK1) receptor expressed in epithelial cells. Biochem J 303: 177–186;
- Schaffer M, Beiter T, Becker HD, Hunt TK (1998). Neuropeptides: mediators of inflammation and tissue repair? Arch Surg 133: 1107–1116;
- Lan Feng, Nannan Shi, Shasha Cai, XueQiao, Peng Chu, Hui Wang, Feida Long, Huaixin Yang, Yongliang Yang, Yipeng Wang, and Haining Yu, ‘’De Novo Molecular Design of a Novel Octapeptide That Inhibits In Vivo Melanogenesis and Has Great Transdermal Ability’’, J. Med. Chem. 61, 6846−6857, 2018;
- Roman A. Laskowski* and Mark B. Swindells; LigPlot+: Multiple Ligand Protein Interaction Diagrams for Drug Discovery, Journal of Chemical Information and Modeling, nº 51, 2011.
1UNIFESO, Teresópolis
2Orientador, UNIFESO, Teresópolis
3Co-orientador, IQ/UFRJ, Rio de Janeiro. Centro Universitário Serra dos Órgãos – Unifeso
Farmácia, CCS – Centro de Ciência e Saúde
