REGISTRO DOI: 10.69849/revistaft/ar10202408062100
Flávio César Thiago;
Orientador Prof. Dr. Matheus de Souza Gomes;
Coorientador Prof. Dr. Fábio Ribeiro Queiroz.
RESUMO
Os miRNAs são pequenos RNAs não codificadores de proteínas que auxiliam e atuam no controle da expressão gênica impedindo a tradução de diversos genes (em sua maior parte). É consenso na literatura científica que os mecanismos de controle da expressão gênica são essenciais para o desenvolvimento do organismo orquestrando vários processos biológicos. A análise de miRNAs em moluscos, sobretudo em caramujos, é muito importante já que possuem poucos miRNAs identificados, quando comparado ao vasto número de espécies de moluscos. Os caramujos do gênero Pomacea são considerados espécies invasoras prejudiciais ao planeta incluindo P. canaliculata e P. maculata. A família Ampullariidae em moluscos constitui um modelo emergente para estudos evolutivos devido à alta diversidade, história antiga e ampla distribuição geográfica. Dessa forma, o objetivo deste trabalho foi identificar e caracterizar através de análises in silico miRNAs maduros conservados e seus respectivos precursores, as prováveis proteínas envolvidas na via de processamento de miRNAs e seus respectivos genes, utilizando banco de dados públicos de genoma e transcriptoma de P. canaliculata e P. maculata. Foi possível identificar 269 pré-miRNAs preditos, 296 miRNAs maduros e 7 prováveis proteínas da via de miRNAs em P. canaliculata. Já em P. maculata foram identificados 299 precursores, 316 miRNAs maduros e 8 prováveis proteínas da biogênese de miRNAs. A caracterização foi realizada baseada na conservação filogenética, na estrutura primária e secundária característica dos precursores e das proteínas da via de miRNAs. Os dados obtidos embasarão estudos de filogenia, divergência populacional, especiação e padrões de diversidade na família Ampullariidae.
Palavras chave: miRNAs. Análise Computacional. Caramujos. Ampullariidae. Genoma.
ABSTRACT
miRNAs are small non-protein coding RNAs that help and act in the control of gene expression, preventing the translation of several genes (for the most part). There is a consensus in the scientific literature that gene expression control mechanisms are essential for the development of the organism, orchestrating several biological processes. The analysis of miRNAs in molluscs, especially in snails, is very important since they have few identified miRNAs, when compared to the vast number of mollusc species. The snails of the genus Pomacea are considered invasive species harmful to the planet, including P. canaliculata and P. maculata. The Ampullariidae family in molluscs is an emerging model for evolutionary studies due to its high diversity, ancient history and wide geographic distribution. Thus, the objective of this work was to identify and characterize, through in silico analysis, conserved mature miRNAs and their respective precursors, the putative-putative proteins involved in the miRNA processing pathway and their respective genes, using public database of genome and transcriptome of P. canaliculata and P. maculata. It was possible to identify 269 predicted pre-miRNAs, 296 mature miRNAs and 7 probable miRNA pathway proteins in P. canaliculata. In P. maculata, 299 precursors, 316 mature miRNAs and 8 probable miRNA biogenesis proteins were identified. The characterization was performed based on phylogenetic conservation, primary and secondary structure characteristic of precursors and proteins of the miRNA pathway. The data obtained will support studies of phylogeny, population divergence, speciation and diversity patterns in the Ampullariidae family.
Keywords: miRNAs. Computational Analysis. Snails. Ampullariidae. Genome.
1 INTRODUÇÃO
1.1 PROBLEMA DE PESQUISA
Os miRNAs exercem mecanismos de controle gênico orquestrando vários processos biológicos. Diante disso, quais miRNAs poderão ser identificados nas espécies do gênero Pomacea em estudo? Se é possível a identificação de miRNAs nas espécies citadas, quais os genes envolvidos na biogênese dos miRNAs?
1.2 HIPÓTESES
Os miRNAs, precursores e maduros, e os genes envolvidos na via de processamento de pequenos RNAs estão presentes no genoma e transcriptoma de P. canaliculata e P. maculata e esta descoberta poderá embasar estudos futuros a respeito de doenças e pragas agrícolas nos quais os caramujos estão envolvidos.
1.3 OBJETIVOS
1.3.1 Objetivo Geral
O objetivo deste trabalho foi a identificar e caracterizar miRNAs e genes envolvidos em sua via de processamento, tanto no genoma quanto no transcriptoma das espécies de moluscos P. canaliculata e P. maculata.
1.3.2 Objetivos específicos
a) Identificar e caracterizar miRNAs conservados, maduros e precursores, no genoma e transcriptoma de P. canaliculata.
b) Identificar as prováveis proteínas envolvidas na via da biogênese de miRNAs em P. canaliculata e P. maculata
c) Caracterizar as prováveis proteínas da via de miRNAs em P. canaliculata e P. maculata, através do alinhamento de proteínas, análise de domínios conservados, sítios ativos e filogenia.
1.4 JUSTIFICATIVA
As duas espécies de caracóis P. canaliculata e P. maculata são muito semelhantes em características morfológicas e adaptativas, apresentando crescimento rápido, alta taxa de reprodução, tolerância ao estresse ambiental, resistência a altas temperaturas e seca. As duas espécies de moluscos são consideradas pragas para algumas culturas agrícolas.
Para tanto, os estudos relativos aos miRNAs contribuirão para uma melhor compreensão a respeito da adaptabilidade e biologia de P. canaliculata e P. maculata. Isto é possível devido a capacidade dos pequenos RNAs não codificantes de proteínas de interferir no perfil de expressão de genes específicos relacionados a possíveis processos de resistência e plasticidade ambiental da espécie.
P. canaliculata tem a propriedade de realizar o controle biológico de B. glabrata, podendo alterar a transmissibilidade da esquistossomose em humanos. A maneira que os miRNAs de P. canaliculata interferem no perfil de expressão de genes poderá fornecer informações importantes para o desenvolvimento de estratégias mais eficientes para o controle da transmissão da esquistossomose. O estudo de miRNAs em P. canaliculata poderá conferir inúmeras respostas ao processo de interação do molusco com o nematódeo A. cantonensis, fornecendo mecanismos para compreensão da transmissão da meningite eosinofílica em humanos e prováveis instrumentos de controle da patologia.
Identificar miRNAs em P. canaliculata e P. maculata fornecerá embasamento para trabalhados futuros, dentre estes o possível mecanismo de controle que os miRNAs destes moluscos exercem na invasividade das culturas agrícolas. Nossos resultados ampliarão o estudo de miRNAs em moluscos, propiciando novos conhecimentos para o entendimento da regulação de miRNAs na família Ampullariidae e nas espécies P. canaliculata e P. maculata.
2 REFERÊNCIAL TEÓRICO
2.1 MOLUSCOS
Moluscos são animais de corpo mole invertebrados não segmentados. O corpo é organizado em um pé musculoso, cabeça e massa visceral que contém a maioria dos sistemas orgânicos. Alguns têm um manto que secreta a concha calcária (BROWN; LYDEARD, 2010). O filo Mollusca é o segundo com maior diversidade de espécies dentre os metazoários e o mais rico em subgrupos. Os habitats podem ser marinhos, de água doce ou terrestres. As espécies podem viver em temperaturas de até 40°C ou ambientes com gelo permanente (HASZPRUNAR; WANNINGER, 2012).
O filo Mollusca é o mais morfologicamente distinto dentre os outros filos, sendo composto por oito classes Chefalopoda (polvos, lulas, etc), Gastropoda (caracóis, lesmas, etc) Bivalvia (ostras, mexilhão,etc), Monoplacophora (moluscos marinhos que vivem em altas profundidades), Solenogastres, Caudofoveata, Polyplacophora e Scaphpoda (GOMES-DOS- SANTOS et al., 2020; SIGWART; LINDBERG, 2015).
A classe gastrópode é a única classe de moluscos que tem representantes tanto em ambientes marinhos como de água doce e também terrestres, possuindo três subclasses: Prosobranchia, Opisthobranchia e Pulmonata (BROWN; LYDEARD, 2010). A classe gastrópode tem mais de 100.000 espécies que compreendem cerca de 80% de todos os moluscos. É o grupo mais diversificado dentro do filo Mollusca. Alguns moluscos gastrópodes de água doce são hospedeiros intermediários de vários trematódeos patogênicos em seres humanos como na esquistossomose (HASZPRUNAR; WANNINGER, 2012). Os gastrópodes quando com concha são univalves e possuem uma boca com rádula (língua com vários dentículos) (BROWN; LYDEARD, 2010).
Os moluscos são usados como fonte de proteína em todo mundo, dentre estes bivalves (mexilhões), cefalópodes (polvos e lulas) e gastrópodes (abalones, lapas, búzios, caracóis terrestres). Há matérias-primas de valor econômico e cultural para humanidade como conchas, opérculos e pérolas, usadas como decoração, joias e moedas (HASZPRUNAR; WANNINGER, 2012). Os moluscos são utilizados para monitoramento da poluição ambiental, sendo espécies- chave do ecossistema que promovem a reciclagem de nutrientes e filtração de água (FIGUERAS et al., 2019; RENAUT et al., 2018).
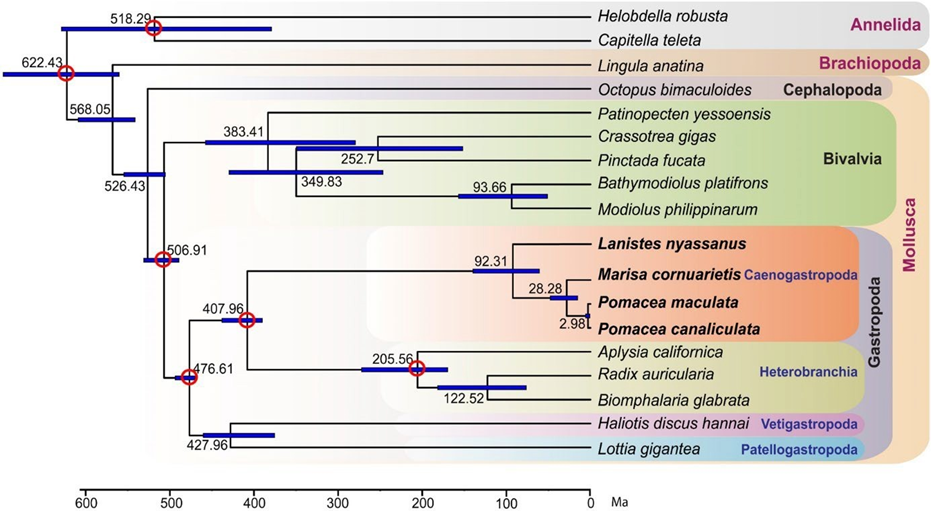
Os moluscos da família Ampullariidae apresentam várias adaptações morfológicas e fisiológicas aos seus habitats que os tornam candidatos ideais para estudo de divergência populacional, especiação, adaptação e padrões de diversidade em maior escala incluindo a biogeografia de populações nativas e invasivas (IP et al., 2018). Há quatro ampularídeos com genomas sequenciados abrangendo os clados do Velho mundo (Lanistes nyassanus) e Novo mundo (P. canaliculata, P. maculata e Marisa cornuarietis) que podem ser objeto de estudos genômicos comparativos e de análise de miRNAs.
A divisão entre as linhagens do Novo e Velho Mundo (figura 1) pode ter ocorrido há cerca de 92,3 milhões de anos atrás (intervalo de confiança de 95% de 61,3–139,0 milhões de anos atrás) que é condizente com a época da separação de Gondwana, em torno de 120 milhões de anos atrás (JOKAT et al., 2003; SUN et al., 2019). Marisa cornuarietis e o gênero Pomacea divergiram a cerca de 28,3 milhões de anos atrás. As duas espécies de Pomacea parecem ter separado há cerca de 3,0 milhões de anos (intervalo de confiança de 95% de 2,0 – 5,0 milhões de anos atrás). O grau de semelhança entre as espécies P. canaliculata e P. maculata é dimensionado a ponto de poderem hibridizar e produzir progênie F1 viável (JOKAT et al., 2003; MATSUKURA et al., 2013; SUN et al., 2019).
Figura 1: Árvore filogenética de P. canaliculata, P. maculata e espécies, construída usando 1.357 genes ortólogos de cópia única com um total de 455.177 aminoácidos.
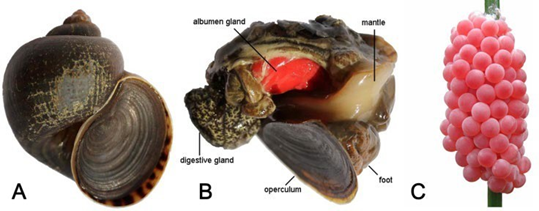
A espécie P. canaliculata (figura 2) é um caramujo de água doce da classe Gastrópode pertencente à família Ampullariidae. Apesar de aquático e possuir brânquias, precisa ir à superfície para realizar as trocas gasosas nos pulmões (JOSHI, 2005). O molusco é popularmente conhecido como caracol maçã, possuindo fertilização interna e são ovíparos (ACCORSI et al., 2013). O gênero Pomacea deposita os ovos acima da superfície da água em objetos sólidos, como rochas e caules de vegetação (HAYES et al., 2009). O fluido perivitelino em torno do embrião em desenvolvimento contém não apenas todos os nutrientes e doadores de energia para o desenvolvimento embrionário (HERAS; GARIN; POLLERO, 1998), mas também muitas macromoléculas que protegem os ovos de estressores ambientais e predadores (DREON et al., 2004; HERAS et al., 2008). Cerca de 21 genes do fluido perivitelino são específicos para a glândula albúmen, indicando que este órgão é a origem de muitas proteínas deste fluido (SUN et al., 2012). A espécie P. canaliculata tem alta variação interpopulacional em relação ao tamanho, forma e espessura da concha e habita ampla gama de ecossistemas como pântanos, lagoas, lagos e rios (ESTEBENET; MARTIN, 2003).
Figura 2: A) Fotos fêmea de Pomacea canaliculata com uma concha intacta (comprimento da concha 4,0 cm). B) O mesmo animal com concha removido para mostrar estruturas corporais, incluindo glândula albumen. C) Ninhada de ovos (cada um com aproximadamente 2,5 mm de diâmetro).
P. canaliculata é o Ampulariídeo com a maior distribuição geográfica conhecida, sendo originário da América do Sul. Está presente na Argentina, Uruguai, Paraguai e Bolívia, sendo abundante nas bacias dos rios da Prata, Uruguai e Paraguai (SILVANA THIENGO, 1993). O molusco também é encontrado na América Central e parte da América do Norte (HALWART, 1994). No Brasil, ocorre principalmente nas regiões Sul, Sudeste, floresta Amazônica e Pantanal (SILVANA THIENGO, 1995). A introdução do molusco na Ásia ocorreu na década de 1980 através de empreendedores das Américas do Sul e do Norte, como iguaria para alimentação. No entanto, houve uma migração para agricultura onde se tornou inesperadamente uma praga do arroz (HALWART, 1994). Na Indonésia, quando introduzido em plantações de arroz, causa uma queda de 15% da produção (NOVARINO, 2011). É descrita como uma espécie invasora e foi classificada entre as 100 maiores pragas em todo mundo (LUQUE et al., 2014). P. canaliculata tem crescimento rápido, alta taxa de reprodução, tolerância e adaptação ao estresse ambiental e distribuição geográfica em todos os continentes. Por estes motivos é considerado uma praga para agricultura, constituindo ameaça grave para o equilíbrio de um ecossistema (LIU et al., 2018).
O molusco tem várias propriedades adaptativas e alta plasticidade ecológica. A espécie é resistente a altas temperaturas e seca (MATSUKURA et al., 2009; YUSA; WADA; TAKAHASHI, 2006). P. canaliculata é capaz de sobreviver em ambientes com metais pesados ocasionando grande concentração nas brânquias (KRUATRACHUE et al., 2011). Na microbiota intestinal do molusco, diversos parasitos reduzem os efeitos tóxicos destes metais (LIU et al., 2018). A lectina neurotoxina presente nos ovos do caramujo defende os embriões dos predadores (DREON et al., 2013). Os ovos de P. canaliculata e P. maculata são depositados na terra, diferente de outros moluscos que os depositam na água. Os ovos depositados em terra evitam o parasitismo e predação. A transição do meio aquático para o terrestre exigiu adaptações morfológicas tais como alongamento das vias respiratórias e aumento do tamanho do pulmão em adultos das espécies P. canaliculata e P. maculata (HAYES et al., 2009). Há presença de um sistema imune neuroendócrino com memória imune específica em P. canaliculata (OTTAVIANI et al., 2013). O sistema de biotransformação de substâncias tóxicas está desenvolvido em moluscos onde foram identificados 157 citocromos P450s nestes animais. A expansão dos genes na família do citocromo P450 em P. canaliculata contribui para resistência ao estresse ambiental. Foram encontradas enzimas antioxidantes (superóxido dismutase, catalase, peroxidase e glutadionaperoxidase) que são fatores inibidores dos radicais livres e condições patológicas (LIU et al., 2018).
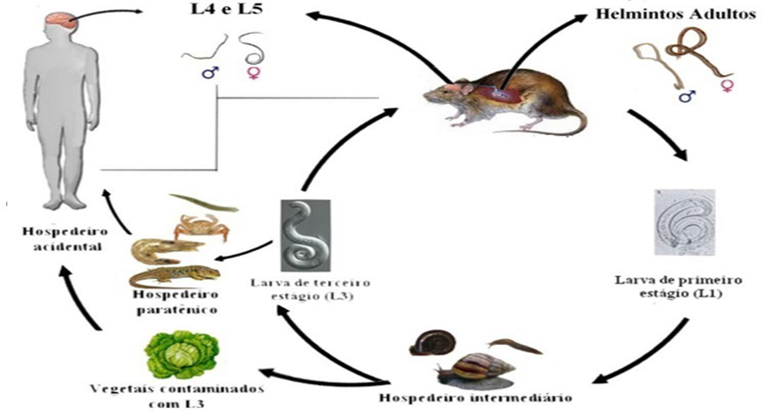
P. canaliculata está envolvido na transmissão do helminto A. cantonensis, causador da meningite eosinofílica em humanos. O ciclo biológico do nematódeo A. cantonensis (Figura 3) é heteroxênico, sendo os roedores hospedeiros definitivos. Nos animais contaminados, a fêmea do helminto adulto faz a postura de seus ovos nas artérias pulmonares dos roedores. Em seguida, ocorre a eclosão dos ovos surgindo a larva de primeiro estágio (L1), que são engolidas e excretadas pelas fezes. Moluscos são os hospedeiros intermediários que se contaminam pela ingestão da Larva L1, pela penetração dessas larvas no poro respiratório ou parede corporal. Ocorre duas mudas larvais dentro dos moluscos, passando pelo estágio larvar L2 originando L3. As larvas de terceiro estágio são transmitidas a hospedeiros paratênicos. Pode ocorrer a contaminação de seres humanos por estes nematódeos através de três maneiras: i) ingestão de hospedeiros paratênicos infectados (caranguejos, lagartos); ii) consumo de hospedeiros intermediários contaminados (moluscos crus ou malcozidos) e; iii) vegetais infestados pelas larvas infectantes. Os humanos são hospedeiros acidentais e no caso de infecção humana, a larva infectante se move até o Sistema Nervoso Central (SNC). O helminto não completa o ciclo de vida em humanos, embora permaneça no SNC causando meningite eosinofílica ou no globo ocular causando angiostrongilíase ocular (LUESSI et al., 2009; WANG et al., 2012, 2008). O episódio aconteceu pela primeira vez no leste da Ásia, sendo P. canaliculata hospedeiro intermediário para infecção em humanos (LV et al., 2008).
A meningite eosinófilica em humanos geralmente apresenta baixa gravidade. A resposta imune produzida pelo hospedeiro contribui para os sinais e sintomas da doença. Os pacientes apresentam meningite caracterizada pela presença de eosinófilos no líquido cefalorraquidiano (ESPÍRITO-SANTO et al., 2013; LUESSI et al., 2009; TSAI et al., 2001). Pode acontecer alguns casos graves da doença com sequelas neurológicas severas, coma e mortes, principalmente em crianças (CHEN et al., 2014a; LINDO et al., 2002; TSAI et al., 2001).
Figura 3: Ciclo biológico de A. cantonensis.
P. canaliculata realiza o controle biológico de B. glabrata, consumindo a desova deste molusco. Em suma, P. canaliculata pode ser uma bioferramenta de controle do hospedeiro intermediário da esquistossomose no ambiente em que coexistem (OLIVEIRA et al., 2012).
Figura 4: Conchas de P. canaliculata (A) e P. maculata (B).

P. maculata (Figura 4) é um grande caracol de água doce nativo da América do Sul que coloniza desde o sul da Amazônia até bacia do Rio de da Plata (HAYES et al., 2012). Ele apresenta características fisiológicas e adaptativas semelhantes a P. canaliculata, sendo ambas as espécies reconhecidas por sua rápida propagação e aumento da população, constituindo ameaça grave para o equilíbrio de um ecossistema. Os custos econômicos do dano, controle e reparação podem ser imensos (PIMENTEL; ZUNIGA; MORRISON, 2005).
O sequenciamento de genes esclareceu muito a distribuição de P. maculata e P. canaliculata nos Estados Unidos, que apesar de serem parecidos fisiologicamente são substancialmente diferenciados geneticamente (HAYES et al., 2012). Levantamentos de uma série de populações sugerem que P. maculata é a espécie predominante em relação a P. canaliculata em pelo menos sete estados nos Estados Unidos: Carolina do Sul, Geórgia, Flórida, Alabama, Mississippi, Louisiana e Texas (MUECK et al., 2018). A extensão da destruição das regiões invadidas causada por P. maculata e P. canaliculata variaram consideravelmente, apesar das semelhanças ambientais e ecológicas entre os habitats afetados (HORGAN; STUART; KUDAVIDANAGE, 2014). Essas diferenças sugerem que pode haver variação fisiológica significativa entre espécies individuais (P. maculata e P. canaliculata) e entre populações das mesmas espécies (MATSUKURA et al., 2016).
2.2 SEQUENCIAMENTO DE DNA
O sequenciamento de DNA ganhou notoriedade em 1975 com o desenvolvimento de tecnologia amplamente utilizada, que ficou conhecida como Sequenciamento de Sanger (SANGER; COULSON, 1975). Este sequenciamento é altamente preciso e permaneceu por décadas como principal forma de sequenciamento de DNA, até o surgimento da nova tecnologia Next Generation Sequencing (NGS). Essa técnica devido ao menor custo e esforço necessário, permitiu que pesquisadores gerassem grande conjunto de dados genômicos (SCHULTZ; ADEMA, 2017). A enorme quantidade de dados gerados por essa tecnologia impulsionou o desenvolvimento, em paralelo com a bioinformática, de novas ferramentas e técnicas de análise (ZHANG et al., 2011).
2.2.1 Sequenciamento em moluscos
O primeiro sequenciamento de genoma de metazoário ocorreu em 1998 no nematoide Caenorhabditis elegans. Desde então, várias sequências genômicas foram identificadas (TAKEUCHI et al., 2012). Há uma tendência crescente no número de publicações que incluem dados genômicos aplicados a moluscos. A maioria dos recursos foram aplicados em gastrópodes e bivalves. A maior parte das abordagens está associada a estudos genéticos populacionais ou mapeamento de características por locus gênico (GOMES-DOS-SANTOS et al., 2020). No banco de dados National Centre for Biotechnology Information (NCBI – http://.ncbi.nlm.nih.gov/), em maio de 2020, há registro de 48 genomas de espécies de moluscos sequenciados.
Moluscos apresentam níveis elevados de diversidade genética (WANG et al., 2017), a taxa de Polimorfismos de Nucleotídeo Único (SNPs) e polimorfismos observados em ostras do pacífico é de 6 a 10 vezes maior que observado em humanos (HOLLENBECK; JOHNSTON, 2018). Outro fator que contribui para a diversidade gênica é a alta taxa de mutação, que pode ser elevada devido ao grande número de eventos necessários na meiose, para produzir milhões de ovos na reprodução (PLOUGH; HEDGECOCK, 2016). Esta variabilidade genética está sendo revelada nos genomas sequenciados e um importante passo foi dado no sequenciamento de genoma completo de moluscos. Em um breve resumo, descreve-se o sequenciamento de genoma completo de alguns moluscos gastrópodes, cefalópodes e bivalves.
O sequenciamento de genoma completo do caramujo de água doce: B. glabrata (classe: gastrópode) foi um marco devido a importante relevância clínica para os humanos. A alteração genética do molusco poderá modificar a transmissibilidade da esquistossomose, doença negligenciada em países subdesenvolvidos. B. glabrata é hospedeiro intermediário do Schistosoma mansoni causador da esquistossomose. O genoma tem um tamanho estimado de 916,4 megabases compreendidas em 18 cromossomos, nos quais 14.423 genes foram identificados. A caracterização deste genoma define melhor o clado Lophotrochozoa quando comparado ao clado Ecdysozoa e aos animais deuterostômios (ADEMA et al., 2017).
O genoma completo sequenciado do molusco Lottia gigantea (gastrópode marinho) foi publicado em 2013 e tem o tamanho estimado em 359,5 megabases (SIMAKOV et al., 2013). Foi verificada a presença de genes que codificam neuropeptídios e neurohormônios neste molusco. Quatro genes codificam peptídeos relacionados à insulina. Foi encontrado um peptídeo semelhante ao GnRH (hormônio liberador de gonadotrofina) e pequenos peptídeos cardioativos, revelando similaridades com outros moluscos e insetos (VEENSTRA, 2010).
O molusco da classe gastrópode, Achatina fulica (caramujo gigante africano), foi o primeiro molusco terrestre a ser sequenciado de forma completa. A extensão do genoma é de 2.120 megabases, com o conteúdo de repetições de até 71% (GUO et al., 2019). Estas repetições são parecidas (70%) no caramujo gastrópode pulmonar marinho Radix auricularia, devido principalmente aos transposons de DNA (SCHELL et al., 2017). No total 23.726 genes codificadores de proteínas foram previstos em A. fulica, compreendidos em 31 cromossomos e também a presença de 675 genes ortólogos relacionados a outros moluscos. O sequenciamento do genoma completo revela inferências filogenéticas entre este caramujo e o molusco P. canaliculata. Ambos são gastrópodes com ampla adaptabilidade ambiental, alta taxa de crescimento e capacidade reprodutiva. As espécies são pragas invasoras de culturas agrícolas que causam queda na produção, prejuízos econômicos e também são hospedeiros intermediários para patologias humanas (GUO et al., 2019)
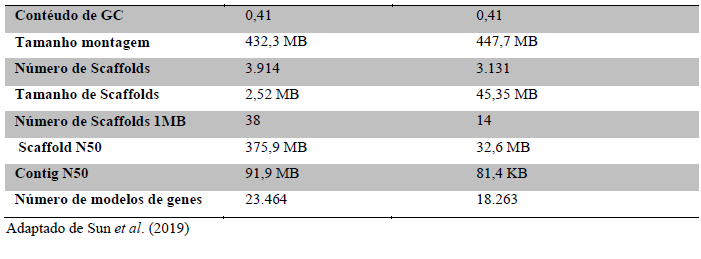
Os genomas de P. canaliculata e P. maculata e foram montados após o sequenciamento de bibliotecas de DNA de inserção curta e longa usando plataformas de sequenciamento Illumina. Os genomas das espécies são considerados compactos quando comparado com outros moluscos, P. canaliculata contém 447,7 megabases e P. maculata 432,3 megabases (Tabela 1).
Tabela 1: Principais estatísticas de montagem de genomas de moluscos.

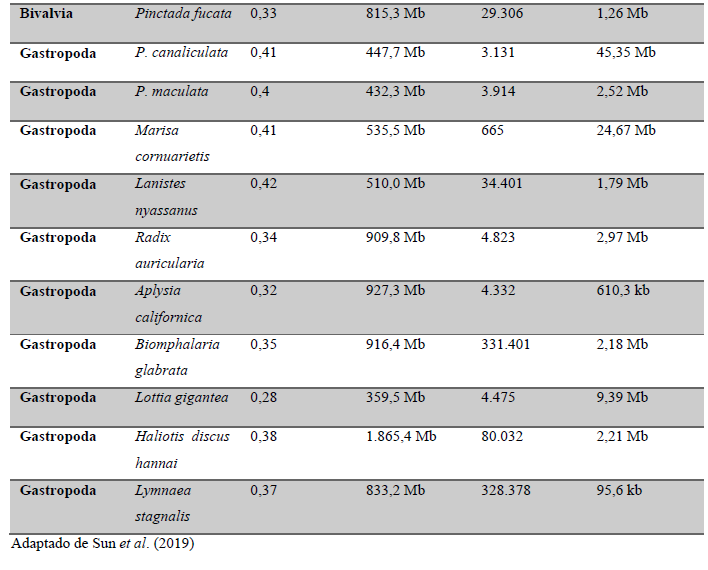
Os genomas de P. canaliculata, P. maculata e Lanistes nyassanus foram montados após o sequenciamento de bibliotecas de DNA de inserção curta e longa usando plataformas de sequenciamento Illumina. Esses três genomas variam de 432,3 a 510,0 megabases de comprimento total com os dois genomas de Pomacea sendo menores, P. canaliculata com 447,7 megabases e P. maculata com 432,3 megabases. O genoma de Marisa cornuarietis foi montado usando leituras curtas Illumina e leituras longas Nanopore, com o comprimento estimado de 535,5 megabases. Os valores finais de scaffold/contig N50 de P. canaliculata é de 32,6 Mb e de M. cornuarietis de 4,4 Mb, excedem a maioria dos outros genomas de moluscos sequenciados, mostrando a alta continuidade das montagens (Tabela 1) (SUN et al., 2019).
O sequenciamento de genoma completo em moluscos cefalópodes foi realizado primeiramente no polvo Octopus bimaculoides no ano de 2015. O tamanho do genoma foi estimado em 2.338,2 megabases (maior genoma conhecido do mundo dos invertebrados) e foram identificados 33.638 genes codificadores de proteína. Quase 45% do genoma é composto por elementos repetitivos, com presença de elementos de transposição. Há elementos sugestivos de duplicação de genoma inteiro, embora não possa ser confirmado. Este fato permite que genes adicionais adquiram novas funções. O desenvolvimento genético neuronal em cefalópodes é semelhante ao encontrado em bilatérios vertebrados. A família de genes protocaderinas está desenvolvida, sendo necessária para o aprimoramento normal dos circuitos neurais. Acreditava- se que as protocaderinas eram desenvolvidas somente em vertebrados. A enorme edição do RNA mensageiro gera diversidades de proteínas envolvidas na excitabilidade neuronal. Justifica-se a maior inteligência dos polvos em relação a outros moluscos. Foram identificados centenas de genes cefalópodes específicos, muitos destes mostraram elevados níveis de expressão na pele (mudança de coloração), ventosas e sistema nervoso (ALBERTIN et al., 2015). No molusco Lymnae stagnalis foi identificado o miR-124 (WALKER et al., 2018) que é também verificado no polvo O. bimaculoides. Este miRNA é bastante abundante no Sistema Nervoso de vertebrados e invertebrados, que contribui para desenvolvimento e diferenciação neuronal (LIU et al., 2011). A análise de microRNAs conservados revela funções parecidas em espécies distintas, as quais estão ligadas evolutivamente.
O sequenciamento do genoma completo do molusco bivalve Pinctada fucata martensii (ostra de pérola), com a extensão de genômica de 1.150 megabases, identificou 32.937 modelos de genes codificadores de proteínas. A análise filogenética de genomas sequenciados de P. fucata martensii (bivalve), Crassostrea gigas (bivalve) e L. gigantea (Gastropoda) forneceu estimativas de divergências em árvores filogenéticas. P. fucata martensii divergiu de seu ancestral comum ao bivalve descritos a 316 milhões de anos atrás, enquanto divergiu do gastrópode descrito a 485 milhões de anos atrás (DU et al., 2017). Já no mexilhão dourado Limnoperna fortunei, molusco de água doce da classe bivalve, o sequenciamento de seu genoma total apresentou o tamanho de 1.600 megabases. Foram identificados 60.717 modelos de genes (58% destes com evidencias transcricionais baseadas em RNA). Estes moluscos são invasores agressivos prejudiciais as espécies nativas na região que colonizam, possuindo alta taxa de reprodução e causam prejuízos econômicos na agricultura. Estas espécies podem detectar produtos químicos na água e fechar suas válvulas como resposta defensiva, sendo tolerantes ao estresse ambiental. A partir do seu genoma sequenciado poderão ser apresentadas as bases moleculares desta invasividade e os mecanismos para controlar as pragas aquáticas (ULIANO et al., 2018).
2.2.2 Sequenciamento nas espécies P. canaliculata e P. maculata
A escassez de genomas sequenciados de moluscos foi apontada pela Aliança Global Genômica de Invertebrados (Global Invertebrate Genomics Alliance) como um problema. A espécie P. canaliculata foi designada como espécie prioritária para sequenciamento de genoma total em 2017 (VOOLSTRA et al., 2017). A importância do sequenciamento genômico deste molusco refere-se a um organismo modelo para estudo, apresentando diversas características inerentes para investigações ecológicas, evolutivas e funcionais. Dessa forma, grande esforço foi realizado para o sequenciamento da espécie, que foi publicado em 11 de maio de 2018 (LIU et al., 2018).
Há um estudo de sequenciamento de genoma total para P. maculata (SUN et al., 2019) e dois estudos para P. canaliculata (LIU et al., 2018; SUN et al., 2019). O primeiro trabalho em 2018 para P. canaliculata revela sequenciamento de alta qualidade com montagem genômica próxima ao nível cromossómico. O tamanho médio do genoma foi estimado em torno de 440 megabases, sendo considerado compacto quando comparado a outros moluscos como por exemplo, Achatina fulica (2.120 megabases) e Octopus bimaculoides (2.338,2 megabases). Os genomas ampularídeos tem um conteúdo de repetições menores do que a maioria dos outros genomas de moluscos (24,2 a 61,1%) (SUN et al., 2019). O baixo conteúdo de elementos repetidos em P. canaliculata, contra 71% em A. fulica e 45% em O. bimaculoides torna o ampularídeo com genoma compacto quando comparado com outros moluscos (ALBERTIN et al., 2015; GUO et al., 2019; LIU et al., 2018). Essa analogia aplica-se também a P. maculata com presença de genoma denso (432,3 megabases). Além disto, o comprimento dos íntrons de ampularídeos é semelhante ao de outros gastrópodes, mas é mais curto do que outros moluscos (SUN et al., 2019).
O genoma de P. canaliculata (Tabela 2) foi montado após sequenciamento de bibliotecas de DNA em plataformas de sequenciamento Illumina. Os valores finais de scaffold / contig N50 de P. canaliculata (32,6 Mb) excede a maioria dos outros genomas de moluscos sequenciados, mostrando alta continuidade das montagens (SUN et al., 2019). No entanto, este estudo tem mais scaffolds (3131) que o trabalho de LIU et al., 2018 (24). O cariótipo haploide é composto por 14 cromossomos e foram estimados 21.533 genes em uma região codificadora de 32,2 mega bases, que corresponde a 7,3% do genoma (LIU et al., 2018). Foi encontrado menor quantitativo de genes (18.263) identificados no estudo de Sun et al. (2019). Os valores menores neste estudo podem ser devido a mais dados transcriptômicos e proteômicos usados para refinamento da predição do modelo de genes na espécie.
Tabela 2: Resumo da montagem e anotação genoma P. canaliculata e P. maculata

Grande parte das abordagens genômicas em moluscos está relacionada ao mapeamento de características por locus gênico (GOMES-DOS-SANTOS et al., 2020). Após o sequenciamento genômico das espécies, foi facilitada a identificação de alguns genes em P. canalicula e P. maculata. Estudos futuros poderão identificar quais miRNAs interagem com a 3’ UTR dos mRNAs alvos codificados por estes genes para verificar se ocorre regulação da expressão gênica (HA; KIM, 2014). Em seguida, há alguns genes de P. canaliculta P. maculata de características evolutivas identificadas, que poderão ser objetivo de estudo de miRNAs.
O gene Pca_154_3.36 de P. canaliculata codifica a proteína CaBP (ligação de cálcio proteína), que pode desempenhar um papel importante na formação da casca dura do ovo, protegendo fisicamente e evitando a dessecação. A proteína pode ter contribuído para a transição evolutiva da deposição de ovos aquática para terrestre nas espécies de ampularídeos. O molusco próximo evolutivamente a P. canaliculata, Marisa cornuarietis (da mesma família) não tem CABP e deposita ovos gelatinosos de forma subaquática. Apesar de haver uma presença generalizada de CaBP no filo molusca, ocorreu uma expansão da família de genes CaBP após a divergência de ampularídeos de outros moluscos. Além disso, a análise de dados proteômicos mostram que o CaBP é altamente abundante no fluido perivitelínico de duas espécies (P. canaliculata e P. maculata), que põem ovos terrestres com uma casca calcária em ampularídeos (HERAS et al., 2008; SUN et al., 2019).
O gene Pca_1306_1.39 de P. canaliculata e o gene Pma_3499_0.31 de P. maculata codificam a proteína MAPCF. Esta proteína é uma subunidade das perivitelinas tóxicas (PV2), as quais compõem o fluido perivitelínico (PVF) nos ovos de P. canaliculata e P. maculata. As PV2 demonstraram ser altamente neurotóxicas para camundongos. É considerada uma proteína defensiva para proteger os ovos ricos em nutrientes de predadores terrestres.
O complexo (PV2) é composto por duas proteínas, a MACPF (provavelmente a porção toxica) e a taquilectina. A análise filogenética mostrou que os genes do tipo MACPF de ampularídeos estão agrupados em dois grupos. O grupo A compreende sete sequências ampularídeas instaladas entre as não ampularídeas, indicando uma possível origem antiga desses genes remontando ao ancestral comum de todos os moluscos. O grupo B é específico para quatro ampularídeos (M. cornuarietis, Lanistes nyssanus, P. maculata e P. canaliculata), compreendendo 17 sequências, indicando que ocorreu extensos eventos de duplicação de genes. Estes fatos aconteceram após a divergência de Ampullariidae de outros moluscos. Dentro do
Grupo B, há uma sequência que codifica a subunidade MACPF de P. canaliculata (Pca_1306_1.39) e Pma_3499_0.31 de P. maculata. Estas aquisições podem ter contribuído para a transição da postura aquática para terrestre de ovos em ampularídeos (DREON et al., 2013; SUN et al., 2019).
Há relatos de expansão das famílias de genes relacionados à detecção ambiental e digestão de celulose, o que pode ter influenciado alguns ampularídeos serem pragas invasivas notórias. Na detecção ambiental, há sete genes de P. canaliculata (Pca_112_8.10, Pca_112_8.16, Pca_112_7.37, Pca_112_8.52, Pca_112_8.32, Pca_112_8.45, Pca_112_8.8) que codificam a família das proteínas G (GRL101) (SUN et al., 2019), as quais estão envolvidas na quimiorrecepção em caracóis aquáticos (SCHULTZ; ADEMA, 2017). Em Ampullariidae ocorreu expansão massiva da família GRL101, com pelo menos dois principais eventos de duplicação de genes nesta família, após a divergência de ampulárideos de outros moluscos. Muitos desses genes GRL101 são altamente expressos nos tentáculos cefálicos e palpos labiais de P. canaliculata. Esta quimiorrecepção sensorial é extremamente importante para ocupação de ambientes transitórios e colonização de territórios, traços inerentes da invasividade e plasticidade ambiental de P. canaliculata e P. maculata (MENDOZA; SEBÉ-PEDRÓS; RUIZ- TRILLO, 2014; LIU et al., 2018; SUN et al., 2019).
2.3 PEQUENOS RNAS NÃO CODIFICADORES DE PROTEINAS
2.3.1 Histórico
Os três tipos principais de pequenos RNAs não codificadores de proteínas em animais são microRNAs (miRNAs), pequenos RNAs interferentes (siRNAs) e RNAs que interagem com PIWI (piRNAs) (OKADA et al., 2009). Os siRNAs têm aproximadamente 21 nucleotídeos de comprimento, são derivados de RNAs em formato de grampo longos (hairpins) e processados pela RNAase Dicer. Os siRNAs medeiam a supressão pós-transcricional através da clivagem de mRNA e contribuem para a defesa antiviral (GHILDIYAL; ZAMORE, 2009).
Os piRNAs têm de 24 a 30 nucleotídeos de comprimento e não dependem de proteínas do tipo RNase III para processamento e são obtidos a partir de precursores de fita simples por uma endonuclease. A expressão gênica é inibida por piRNAs através da clivagem do mRNA (IPSARO et al., 2012; NISHIMASU et al., 2012; SIOMI et al., 2011). A principal função dos piRNAs é silenciar elementos transponíveis em células germinativas.
Os miRNAs foram descobertos em 1993 por Victor Ambros e colaboradores ao estudar o nematódeo C. elegans. Foram verificados indícios de controle da expressão genica por pequenos RNAs, com funções diferentes até então conhecidas. No gene lin-4 foi observada a presença de pequenos RNAs não codificantes de proteínas. Estes miRNAs continham algumas sequências complementares à região 3’UTR do mRNA transcrito do gene lin-14, que reprimia a expressão pós-transcricionalmente impedindo a tradução (LEE; FEINBAUM; AMBROS, 1993). Desde então, miRNAs foram detectados em vários organismos e mostraram ser altamente conservados entre as espécies (FRIEDLÄNDER et al., 2014).
Os miRNAs são pequenos RNAs não codificantes de proteínas com cerca de 22 nucleotídeos. A maioria dos miRNAs é transcrita de sequências de DNA em miRNAs primários (pri-miRNAs), processados em miRNAs precursores (pré-miRNAs) e subsequentemente em miRNAs maduros. Na grande parte dos casos, os miRNAs interagem com a 3′ UTR dos mRNAs alvos para suprimir expressão gênica (HA; KIM, 2014). A interação de miRNAs com outras regiões, incluindo a 5’ UTR , a sequência de codificação e genes promotores também foram relatados para inibição gênica (BROUGHTON et al., 2016). Os miRNAs também demonstraram ativar a expressão gênica em determinadas situações (VASUDEVAN, 2012).
Cerca de metade de todos os miRNAs identificados são intragênicos e processados principalmente a partir de íntrons, os demais são intergênicos regulados por seus próprios promotores. O domínio na extremidade 5ʹ dos miRNAs que se estende da posição 2 a 7 é crucial para o reconhecimento do alvo (mRNA) e foi denominada seeds, “semente de miRNA ou resíduos-semente”. Os nucleotídeos 13 a 16 também contribuem com o emparelhamento de mRNAs alvos (RIE et al., 2017; HA; KIM, 2014; KIM; KIM, 2007).
É consenso na literatura científica os mecanismos reguladores dos miRNAs tais como: diferenciação celular, apoptose, proliferação e tumorigênese, hematopoiese, organogênese; desenvolvimento das células tronco pluripotentes e testicular (FABIAN; SONENBERG; FILIPOWICZ, 2010; KIM, 2005; LEONARDO et al., 2012; RAKOCZY et al., 2013). Os miRNAs desempenham papéis importantes na plasticidade cerebral e memória e em algumas situações se comportam como hormônios, sendo encapsulados em exossomas e transportados a distância (ANFOSSI et al., 2017; BREDY et al., 2011). Da mesma forma, como elemento regulador, o próprio miRNA é modulado de maneira coordenada por diversos fatores (FABIAN; SONENBERG; FILIPOWICZ, 2010).
Os miRNAs têm papel regulatório em processos patológicos (KIM, 2005). No câncer há miRNAs envolvidos na metástase e tumorigênese (BRACKEN et al., 2009). Há relatos de expressões disfuncionais de miRNAs característicos nesta patologia (IORIO; CROCE, 2009). No diabetes, alguns miRNAs estão envolvidos na produção de células β pancreáticas deformadas, prejudicando a produção de insulina. Os miRNAs são possíveis alvos para desenvolvimento de fármacos, como nestes casos (OETJEN, 2012).
Os miRNAs podem servir como biomarcadores para exames de diagnóstico. Nas infecções por Epstein-barr crônicas há elevação de miRNAs virais específicos, sendo potenciais biomarcadores da gravidade da doença ou prognóstico (KAWANO et al., 2013). Dois miRNAs (mir-k12-10b, k12-12) provenientes do Herpes vírus causador do sarcoma de Kaposi (KSHV) foram identificados em pacientes que desenvolveram sepse em grandes cirurgias (TUDOR et al., 2014). A desregulação destes dois miRNAs podem levar a falência dos órgãos e morte. A identificação precoce destes marcadores poderá detectar pacientes com alto risco de choque séptico (GIZA et al., 2016). Em 2007, pela primeira vez, foi relatado miRNAs disfuncionais no sangue de pacientes com linfoma. Já em 2008 verificou-se a potencialidade para serem utilizados como biomarcadores no câncer. Há vários estudos a este respeito, um destes diz respeito a níveis de expressão dos miRNAs (miR-21, miR-126, miR- 155, miR199a e miR-335), associando às manifestações clínico patológicas do câncer de mama. A possibilidade de utilizar miRNAs como biomarcadores apresenta vantagens por não ser um método invasivo, já que o padrão ouro para diagnóstico na patologia é a biopsia (FERNANDEZ-MERCADO et al., 2015; LAWRIE, 2007; LAWRIE et al., 2008; WANG et al., 2010).
2.3.2 Biogênese dos miRNAs
2.3.2.1 Via Canônica
A via canônica da biogênese de miRNAs é a via dominante no qual os miRNAs são processados pelas RNAases III, Drosha e Dicer, consecutivamente. O miRNAs podem ter origem intragênica (em sua maior parte íntrons) ou intergênica. As unidades de transcrição independentes podem ser transcritas individualmente de forma monocistrônica ou constituindo uma unidade de transcrição policistrônica, sendo transcritos em sequências longas chamadas clusters. Os pri-miRNAs (transcrito primário) são transcritos de seus genes pela RNA polimerase II eventualmente regulados pela RNA polimerase III e depois gerados os pré- miRNAs, que possuem aproximadamente 65 nucleotídeos no núcleo celular. Tanto os pri- miRNAs quanto pré-miRNA têm a estrutura secundária em forma de grampo de cabelo (hairpin). Em seguida, os pré-miRNAs são processados em miRNAs maduros no citoplasma (LEE, 2002; TANZER; STADLER, 2004; ZHANG; ZHANG; SU, 2009).
O complexo de microprocessadores, composto por uma proteína de ligação (DiGeorge Syndrome Critical Region 8 – DGCR8) e uma enzima ribonuclease III (Drosha), realiza a clivagem do pri-miRNA. O DGCR8, também conhecido como Pasha em Drosophila melanogaster, se liga em uma estrutura no pri-miRNA, enquanto Drosha realiza a cisão do duplex. Depois que os pré-miRNAs são gerados, eles são transportados para o citoplasma através do complexo exportina 5 (XPO5) / RanGTP, em seguida processados pela endonucelase RNA III Dicer (ALARCÓN et al., 2015; DENLI et al., 2004; HAN et al., 2004; OKADA et al., 2009).
No citoplasma os pré-miRNAs são clivados por Dicer, sendo esta constituída por três módulos, dois domínios de RNase III e um domínio PAZ (Piwi, Argonauta e Zwille) (MACRAE et al., 2006). A clivagem por Dicer pode ocorrer na extremidade 5’ ou 3’. Na extremidade 3’, a clivagem ocorre geralmente a uma distância fixa de 21 a 25 nucleotídeos, dependendo da espécie e tipo de Dicer. Na extremidade 5’, em mamíferos e moscas, a divisão acontece a 22 nucleotídeos de distância (MACRAE; ZHOU; DOUDNA, 2007; PARK et al., 2011). A cadeia 5p surge da extremidade 5’ do hairpin, enquanto que a cadeia 3p é originária da extremidade 3’ do pré-miRNA. Ambos os fios derivados do duplex miRNA maduro podem ser carregados pela família de proteínas Argonauta (AGO), em humanos (AGO1-4) de maneira dependente de ATP (KHVOROVA; REYNOLDS; JAYASENA, 2003; MEIJER; SMITH; BUSHELL, 2014; YODA et al., 2010).
2.3.2.2 Via não canônica
A via não canônica de biogênese dos miRNAs foge do processamento sequencial canônico por DGCR8/Drosha e Dicer. Os miRNAs das vias não canônicas podem ser processados por diferentes combinações do processo canônico via DGCR8/Drosha, Dicer, Exportina 5 e Argonauta (AGO2). Nesta via, a biogênese de miRNAs pode ser independente de Drosha ou Dicer (BABIARZ et al., 2008; RUBY; JAN; BARTEL, 2007).
Alguns íntrons imitam a característica estrutural em formato de grampo dos prémiRNAS, para entrar na via de processamento de miRNA sem clivagem por Drosha. Estes íntrons são chamados de mirtrons. Da mesma forma, alguns transcritos curtos com “capeamento” de 7-metilguanosina (m7G) na extremidade 5’ são exportados diretamente para o citoplasma através de Exportina 1 sem a necessidade de clivagem por Drosha. A presença do “capeamento” do miRNA na cadeia 5’ impede o carregamento por Argonauta, favorecendo a utilização de miRNA-3p (RUBY; JAN; BARTEL, 2007; XIE et al., 2013).
Alguns miRNAs podem ser processados independentes de Dicer, onde Drosha gera pequenos transcritos de RNA em forma de grampo (shRNA). Estes pré-miRNAs são de comprimento insuficiente para serem processados por Dicer. Dessa forma, são carregados e clivados pelo núcleo catalítico de Argonauta (AGO2) (CHELOUFI et al., 2010; YANG et al., 2010).
2.3.3 Mecanismos de regulação gênica desenvolvidos por miRNAs
Depois que os miRNAs maduros são sintetizados, eles induzem o silenciamento da expressão gênica (em sua maior parte). São formadas estruturas efetoras de ribonucleoproteínas chamado de Complexo de Silenciamento Induzido por miRNAs, miRISC. A formação deste complexo silenciador começa com o recrutamento da família de proteínas GW182, as quais induzem o recrutamento de outras proteínas efetoras (BEHM-ANSMANT et al., 2006). No núcleo de miRISC está a família de proteínas Argonauta (AGO), que é carregada com uma fita guia de miRNA. Os miRNAs pareiam suas bases com uma relação do tipo Watson-Crick com os RNAs alvo. O complexo é direcionado para o pareamento das bases com mRNA resultando em degradação do mRNA ou repressão da tradução (HAMMOND et al., 2000).
Alguns membros da família Argonauta tem a atividade de fatiamento de mRNAs, exigindo grau de complementariedade entre as bases do miRNA e mRNA para tal ação (ELBASHIR; LENDECKEL; TUSCHL, 2001; JO et al., 2015). Quando a clivagem do alvo é inibida devido às limitações no pareamento de bases ou falta da atividade catalítica do Argonauta, a expressão de mRNAs alvo é reprimida via repressão da tradução ou deadenilação seguida pela degradação do mRNA (HUNTZINGER; IZAURRALDE, 2011). Nas células animais, a maioria das interações miRNA-RNA alvo não são totalmente complementares, gerando “mismatches”. Dessa forma, é impedida a atividade de endonuclease de AGO2, que passa a atuar como mediador da interferência com RNA (JONAS; IZAURRALDE, 2015). A ligação de miRISC com mRNA resulta em quatro vias de reação, clivagem do alvo, ligação transitória com o alvo, ligação estável com o alvo e por fim, ligação da fita guia com o alvo e descarregamento da Argonauta (JO et al., 2015; KAWAMATA; TOMARI, 2010).
A maioria dos estudos relatam inibição da expressão gênica, alguns citam a ativação da tradução mediada por miRNAs. A presença de AGO2 (Argonauta) e outras proteínas como FXRP1 (proteína do retardo mental do X frágil) estão associadas ao complexo miRNA-proteína (microRNPs). Estes elementos estão relacionados a elementos ricos em AU no terminal 3’ UTR para ativar a tradução (VASUDEVAN; STEITZ, 2007). Outros exemplos de ativação de genes por miRNAs inclui a ligação ao terminal 5 ′ UTR de mRNAs, que induz a codificação de proteínas ribossômicas durante a privação de aminoácidos (NIELSEN; LUND, 2008).
2.4 miRNAs EM MOLUSCOS E NAS ESPÉCIES P. canaliculata E P. maculata
Em um estudo em B. glabrata utilizando análises in sílico e in vitro foi possível identificar 89 miRNAs maduros, sendo 5 deles molusco específicos, além de piRNAs identificados no genoma de B. glabrata pela primeira vez. Alguns miRNAs se destacaram por apresentar número elevado de leituras; como bgl-bantam, bgl-let-7, bgl-miR-100, bgl-miR-184, bgl-miR-71, bgl-miR-1984, bgl- miR-1a, bgl-miR-315, bgl-miR-375, bgl-miR-750, bgl-miR-8 e bgl-miR-92; sugerindo que estes miRNAs possam ter um papel importante na biologia dos caramujos. Para tanto, bgl-miR-100 e bgl-miR-1a foram os mais expressos, podendo desempenhar papel preponderante na regulação do desenvolvimento e na resposta à infecção de B. glabrata. Um número significativo de alvos previstos em B. glabrata estão envolvidos na resposta ao estresse, que podem ser determinantes do processo adaptativo dos caramujos à constante colonização por inúmeros microrganismos. Há vários estudos que relatam a susceptibilidade de B. glabrata para a infecção com o parasito Schistosoma mansoni, alguns dos quais tentam desvendar os mecanismos genéticos que regulam fisiologicamente o molusco. No entanto, nenhum estudo está disponível atualmente que elucida o papel de pequenos RNAs na relação caramujo hospedeiro-parasita (QUEIROZ et al., 2020).
Assim como nos outros metazoários, os miRNAs realizam diversos mecanismos de controle gênico. No bivalve (ostra plana) Ostrea edulis após ser exposto a parasitas, revelou expressão diferencial de 63 miRNAs. Desses, 15 miRNAs têm um papel direto nas respostas como proliferação, ativação de macrófagos, inflamação, apoptose e/ou dano oxidativo, conforme relatado para outros organismos. Dessa forma, dentre as diversas funções desempenhadas nos moluscos pelos miRNAs, eles atuam como biomarcadores, que elucidam efeitos imunotóxicos causados por fatores bióticos e abióticos (BURGOS-ACEVES et al., 2018; MARTÍN-GÓMEZ et al., 2014)
Os miRNAs mostraram-se altamente conservados, principalmente entre os metazoários e também em moluscos. No gastrópode Littorina littorea cinco miRNAs precursores foram expressos no músculo do pé (miR-1a-1, -34a, -133a, -125b, -29b e -2a) e três (miR-1a-1, -34a, -29b) no hepatopâncreas, após serem expostos a condições estressantes como hipóxia e gelo. Estes miRNAs mostraram-se conservados em espécies de invertebrados, o que é provável inferir que estariam presentes em todos os moluscos (BIGGAR et al., 2012; OWENS; MALHAM, 2015). OmiRNA-124a está relacionado a processos regenerativos do Sistema Nervoso Central (SNC) de vertebrados, quando associado ao metabólito ácido retinóico. Este miRNA foi identificado pela primeira vez no filo Mollusca, no SNC do gastrópode Lymnaea stagnalis, com padrões de expressões semelhantes aos vertebrados (WALKER et al., 2018), corroborando a alta conservação de miRNAs em moluscos e outras espécies.
Há poucos miRNAs de moluscos identificados no miRBase versão 22 (http://www.mirbase.org/), sendo 245 miRNAs maduros e 154 precursores. Apenas 3 espécies de moluscos já tiveram miRNAs registrados: Lottia gigantea (64 miRNAs maduros), Haliotis rufescens (5 miRNAs maduros) e Melibe leonina (176 miRNAs maduros). Já foram catalogados 48.885 miRNAs maduros e 38.589 precursores de 271 espécies identificadas. Há necessidade de maior identificação de miRNAs em moluscos, devido ao extenso número de espécies e poucos registros. Nos artrópodes, filo com maior variedade de espécies, já foram identificados 8.753 miRNAs maduros. Justifica-se assim o estudo de miRNAs em moluscos especificamente nas espécies P. canaliculata e P. maculata. Estes dados estão disponíveis no website do banco de dados miRBase versão 22 (http://www.mirbase.org/) que foi consultado em 16 de maio de 2020.
A regulação da expressão gênica em eucariotos é realizada através de um controle fino, na qual miRNAs e suas vias de silenciamento realizam uma regulação específica da expressão gênica e manutenção da integridade do genoma. Quase todos os processos biológicos, incluindo diferenciação celular, desenvolvimento de tecidos e metabolismo celular podem ser modulado por miRNAs (BARTEL, 2009).
A identificação de miRNAs conservados em moluscos revela diversos mecanismos de modulação nos processos biológicos dentre eles a estivação, que é um fenômeno caracterizado por redução da taxa metabólica para menos de 30% nas condições de repouso. Em um estudo realizado no molusco gastrópode pulmonar terrestre Otala láctea (caracol de leite), o fenômeno da estivação é necessário devido às condições ambientais estressantes, com altas temperaturas, comida e água limitadas. Este molusco reduz sua taxa metabólica em um fenômeno parecido com a hibernação dos ursos. Este fenômeno foi verificado também nos caracóis maçã invasores como P. maculata, sendo fundamental à sobrevivência da espécie em climas com períodos prolongados de seca (HOYECK; HADJ-MOUSSA, 2019; MUECK et al., 2018).
Os miRNAs são potenciais candidatos desta regulação na estivação, uma vez que são rápidos, reversíveis e com baixa demanda de energia. Neste estudo foi verificada a expressão de 75 miRNA conservados, dos quais 26 miRNAs foram superexpressos durante a estivação no molusco O. láctea, sendo seus genes suprimidos durante a dormência. Estes miRNAs superexpressos são implicados nos mecanismos de sobrevivência celular como: i) anti- apoptose; ii) supressão de tumores e; iii) respostas de manutenção muscular. O presente estudo mostrou níveis elevados de cinco miRNAs anti-apoptose (miR-2a-3p, miR-2c-3p, miRNA- 124c, miRNA-153) e miRNA-190 no músculo do pé (HOYECK; HADJ-MOUSSA, 2019).
Outro mecanismo de controle gênico é exercido por miRNAs no abalone Haliotis midae (molusco gastrópode marinho) cultivado na África do Sul. O animal é considerado uma iguaria na culinária e perdas comerciais ocorrem devido a doenças infecciosas no abalone. Os miRNAs são capazes de modular a respostas imune em resposta ao estresse, causado pelos processos infecciosos. No estudo preliminar pretendia-se identificar possíveis pequenos RNAs moduladores da resposta imune no molusco, para evitar processos infecciosos e perdas econômicas (PICONE; RHODE; ROODT-WILDING, 2017). Os miRNAs silenciam genes alvo através da destruição específica de mRNA. A detecção de genes nos hospedeiros e não nos patógenos envolvidos na infecção podem ser alvos potenciais para o desenvolvimento de fármacos (HONG-GELLER; MICHEVA-VITEVA, 2010).
Em um estudo em Lymnaea stagnalis (molusco gastrópode de água doce) foram identificados 264 miRNAs maduros conservados e 219 novos miRNAs (WALKER et al., 2018). Já em Haliotis discus hannai (molusco gastrópode marinho) foram identificados 205 miRNAs maduros, dentre estes 15 conservados e 190 novos miRNAs (HUANG et al., 2018). Em animais bivalves (Mytilus galloprovincialis) foram identificados 104 miRNAs conservados e 33 novos miRNAs (YU et al., 2020). Os novos miRNAS maduros identificados não correspondem a nenhuma outra espécie conhecida no miRBase versão 22 (http://www.mirbase.org/). Revela-se a importância da pesquisa de novos miRNAs em moluscos (LAGOS-QUINTANA et al., 2001).
A identificação de miRNAs maduros e seus precursores é realizada por estratégias experimentais e computacionais. As abordagens experimentais, apesar de apresentarem evidências da presença de miRNAs, podem excluir moléculas expressas em certos estágios e tecidos (GOMES et al., 2011). Assim, análises in silico são úteis para espécies com genoma completo sequenciado, permitindo a descoberta de miRNAs usando informações de DNA de genoma inteiro (LI et al., 2010).
Os miRNAs let-7a, miR-279-3p e miR-8-3p mostraram-se abundantes em múltiplos tecidos de moluscos, enquanto miR-9a-3p, miR-183, miR-1986, miR-1990-3p e miR-1993-3p foram expressos de uma maneira específica em alguns tecidos. Os miRNAs 9a-3p, miR-1986 e miR-1990-3p foram altamente expressos nos tecidos do manto em comparação com outros tecidos, o que pode indicar um papel vital por parte desses miRNAs no processo de biomineralização em Pinctada Fucata (HUANG et al., 2019, 2021).
Não há registros de miRNAs em P. canaliculata e P. maculata cadastrados no banco de dados miRBase versão 22 (http://www.mirbase.org/). Os miRNAs, precursores e maduros, e os genes envolvidos na via de processamento de pequenos RNAs estão presentes no genoma e transcriptoma de P. canaliculata e P. maculata. Desta forma, o objetivo deste trabalho foi identificar e caracterizar miRNAs e, genes envolvidos em sua via de processamento, tanto no genoma e quanto no transcriptoma das espécies de moluscos P. canaliculata e P. maculata.
3 MATERIAIS E MÉTODOS
3.1 BANCO DE DADOS DOS MOLUSCOS P. canaliculata E P. maculata
As sequências genômicas e transcriptômicas dos moluscos em estudo foram buscadas no banco de dados NCBI (National Centre for Biotechnology Information ― http://.ncbi.nlm.nih.gov/) e do GigaDB (http://gigadb.org)(GIGADB), [s.d.]., com a finalidade de identificar e analisar miRNAs e os genes envolvidos na via de processamento de pequenos RNAs.
3.2 PREDIÇÃO DE miRNAs PERCURSORES E MADUROS
A análise in silico foi realizada utilizando um algoritmo robusto desenvolvido por GOMES et al., 2011 para predição de miRNAs e seus precursores. As análises computacionais foram realizadas no Laboratório de Bioinformática e Análises Moleculares (LBAM), da Universidade Federal de Uberlândia (UFU) no Campus Patos de Minas, seguindo a metodologia estabelecida para predição de miRNAs e precursores a partir do genoma e transcriptoma (GOMES et al., 2011). Foram obtidas as sequências do genoma com tendência em formar estruturas de hairpin ou semelhança com miRNAs precursores utilizando o programa Einverted (ferramenta EMBOSS) e o programa BLASTn (NCBI). A identificação de miRNAs homólogos foi realizada a partir do miRBase versão 22 (http://www.mirbase.org/). Estas sequências foram submetidas a uma série de filtros para descartar sequências indesejáveis, permanecendo apenas as sequências correspondentes aos prováveis miRNAs. Os filtros utilizados foram baseados em características conservadas de precursores de miRNAs. Estes filtros foram: i) homologia com miRNAs maduros conservados; ii) conteúdo de GC (guanina e citosina) entre 30% a 65%; iii) Energia Mínima Livre (MFE); iv) homologia com regiões repetidas e; v) homologia com RNAs não codificadores de proteínas, exceto miRNAs.
3.3 ALINHAMENTO DOS miRNAs COM ORTÓLOGOS E CONSTRUÇÃO DA ÁRVORE FILOGENÉTICA
Os prováveis precursores de miRNAs foram comparados com seus ortólogos em vários níveis, através do alinhamento múltiplo de sequências e construção de árvores filogenéticas. As sequências de pré-miRNAs e seus respectivos miRNAs foram submetidos ao alinhamento múltiplo, por meio das ferramentas ClustalX 2.1 e RNAalifold (THOMPSON; GIBSON; HIGGINS, 2002). Para a ferramenta ClustalX 2.1 foram utilizados os seguintes parâmetros: abertura de espaço (“gap opening”) 22,50 e extensão do espaço (“gap extension”) 0,83 (TAKANE et al., 2010). Os prováveis miRNAs precursores escolhidos pela metodologia descrita foram submetidos ao alinhamento entre as espécies mais próximas evolutivamente, bem como aquelas presentes em grupos taxonômicos importantes para comparação. Através do banco de dados miRBase versão 22 (http:// mirbase.org/) foi utilizada a ferramenta BLASTn, na qual foram escolhidos os pré-miRNAs ortólogos. Houve a comparação dos prováveis precursores da espécie estudada com os precursores de miRNAs ortólogos conservados, tanto a nível primário (sequências nucleotídicas) quanto ao nível de estrutura secundária dos precursores.
A análise filogenética dos prováveis pré-miRNAs de P. canaliculata e P. maculata foi realizada com o auxílio do programa MEGA versão X (TAMURA et al., 2007) utilizando o método Neighbor-joining e aplicando o modelo Kimura dois parâmetros (“Kimura- twoparameters”), para estimular a divergência entre as sequências (SAITOU; NEI, 1987). As árvores consenso foram obtidas utilizando “bootstrap” de 5000 réplicas.
3.4 PREDIÇÃO DOS GENES E PROTEÍNAS ENVOLVIDOS NA BIOGÊNESE DA VIA DE miRNAs EM P. canaliculata E P. maculata
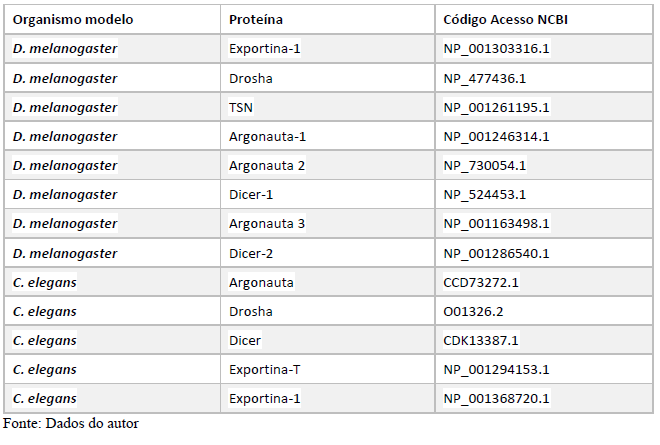
A princípio, para pesquisar as supostas sequências proteicas envolvidas na via miRNA de P. canaliculata e P. maculata, foi utilizada a ferramenta BLASTp (National Centre for Biotechnology Information – http://www.ncbi.nlm.nih.gov/) usando como consultas a sequência de proteínas de referência de espécies animais, como Drosophila melanogaster e C. elegans, organismos modelos (Tabela 3). As supostas sequências de proteína e os prováveis genes ortólogos de P. canaliculata e P. maculata foram encontrados e coletados no proteoma predito das espécies no National Centre for Biotechnology Information (NCBI – http://www.ncbi.nlm.nih.gov/) e utilizados para análise comparativa de sequências. As sequências de aminoácidos obtidas foram utilizadas para caracterização e validação da maquinaria de processamento de miRNAs em P. canaliculata e P. maculata. No genoma da espécie foram localizadas as sequências dos genes utilizando informações oriundas dos arquivos de anotação disponível no banco de dados da espécie e um script em PERL, para a busca das posições dos nucleotídeos no genoma (CARDOSO et al., 2020; GOMES et al., 2011).
Tabela 3 – Proteínas ortólogas de organismos modelo utilizado para pesquisa no banco de dados
3.5 ANÁLISE DE DOMÍNIOS CONSERVADOS, ALINHAMENTO MÚLTIPLO DE SEQUÊNCIAS E ANÁLISE FILOGENÉTICA DAS PROTEÍNAS DA VIA DE miRNAs
As sequências preditas de aminoácidos das prováveis proteínas envolvidas na via de processamento de miRNAs em P. canaliculata e P. maculata foram submetidas ao alinhamento múltiplo de sequência com suas prováveis proteínas ortólogas. Foi utilizada a ferramenta ClustalX 2.1 para analisar alinhamento de estruturas primárias e secundárias das proteínas preditas e seus ortólogos, realizado com parâmetros default (LARKIN et al., 2007).
A busca por famílias de domínios e motivos conservados das sequências preditas de aminoácidos das prováveis proteínas envolvidas na via de miRNAs de P. canaliculata e P. maculata foi realizada utilizando o banco de dados de família de proteínas PFAM versão 32.0 (Protein Family database – https://pfam.xfam.org/) (EL-GEBALI et al., 2019; FINN et al., 2010) e o banco de dados de domínios conservados CDD (https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi) (LU et al., 2020).
As sequências das proteínas preditas de P. canaliculata, P. maculata e possíveis ortólogos de referência (RefSeq) foram submetidos a análises filogenéticas comparativas utilizando o programa MEGA versão X (TAMURA et al., 2007). As árvores filogenéticas foram construídas utilizando o método Neighbor-joining (NJ) (SAITOU; NEI, 1987) e calculada com o modelo de substituição JTT. A árvore filogenética consenso foi elaborada utilizando a análise de teste de bootstrap para 2000 réplicas representando a história evolutiva do grupo de sequências estudadas. A porcentagem de réplicas das árvores filogenéticas foi realizada pelo teste de boostrop apresentando no final a árvore consenso. As posições contendo espaços ou dados perdidos de sequências foram subtraídos do conjunto de dados (GOMES et al., 2011).
4 RESULTADOS E DISCUSSÃO
4.1 BANCO DE DADOS DOS MOLUSCOS P. canaliculata E P. maculata.
Foi realizada uma busca em alguns bancos de dados, a fim de identificar informações genômicas e transcriptomicas das espécies P. canaliculata e P. maculata incluindo o banco de dados do NCBI (National Centre for Biotechnology Information – http://www.ncbi.nlm.nih.gov/), com registros descritos na Tabela 4 abaixo.
Tabela 4: Dados extraídos do banco de dados NCBI de P. canaliculata e P. maculata.

4.2 PREDIÇÃO DE miRNAs PERCURSORES E MADUROS
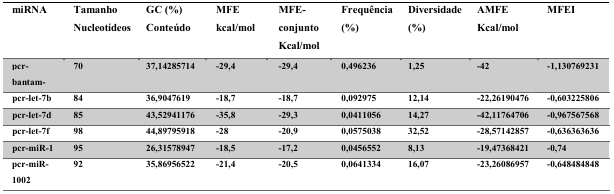
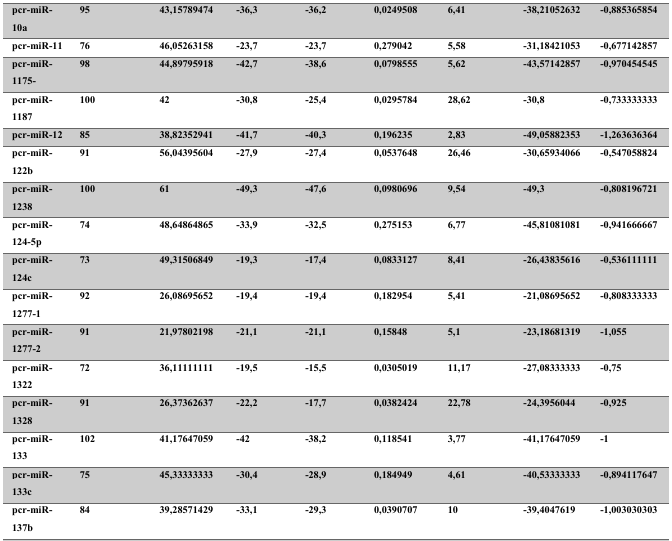
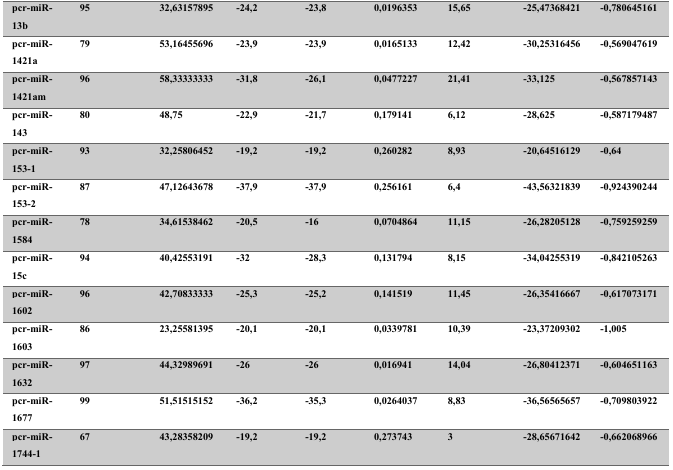
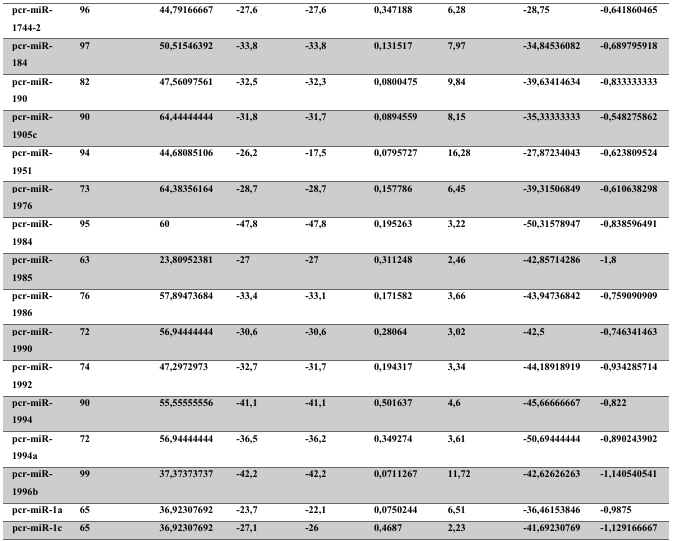
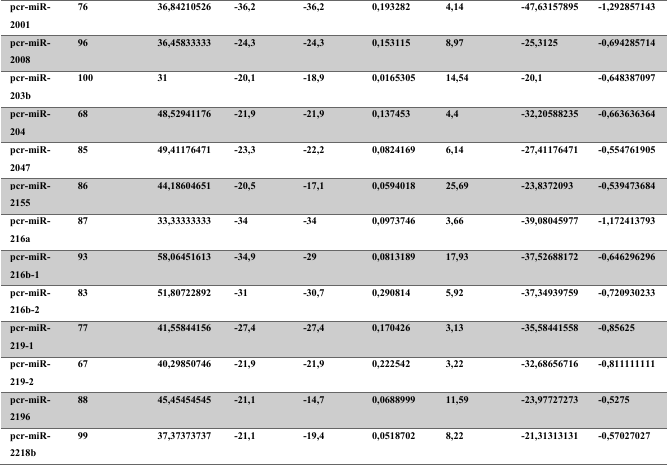
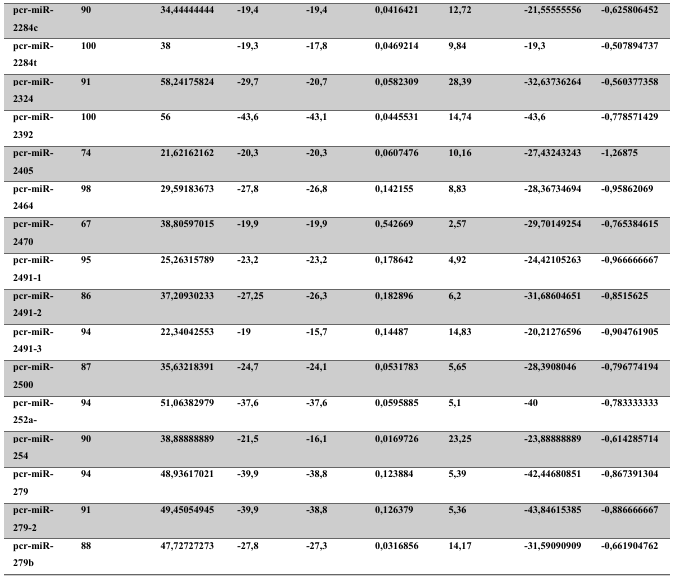
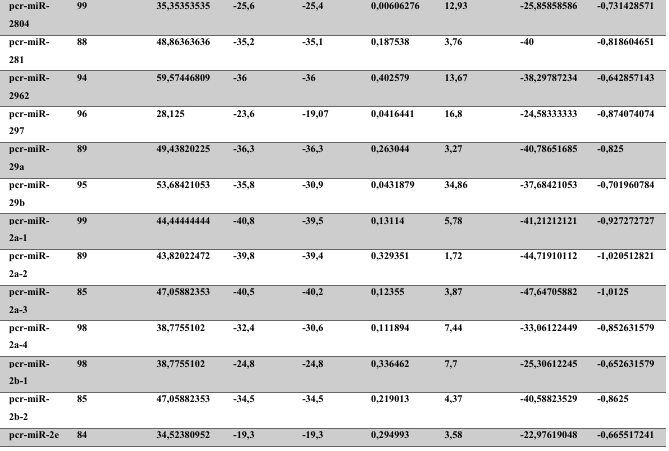
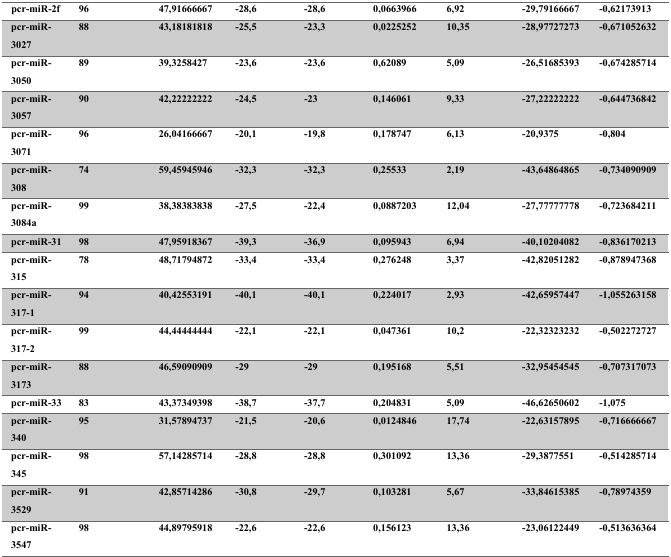
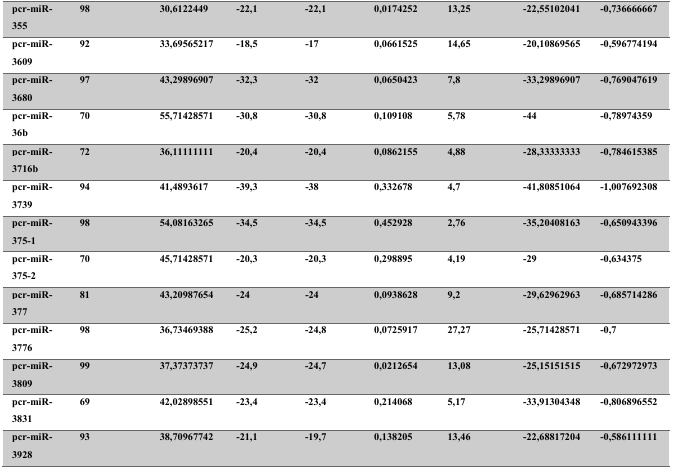
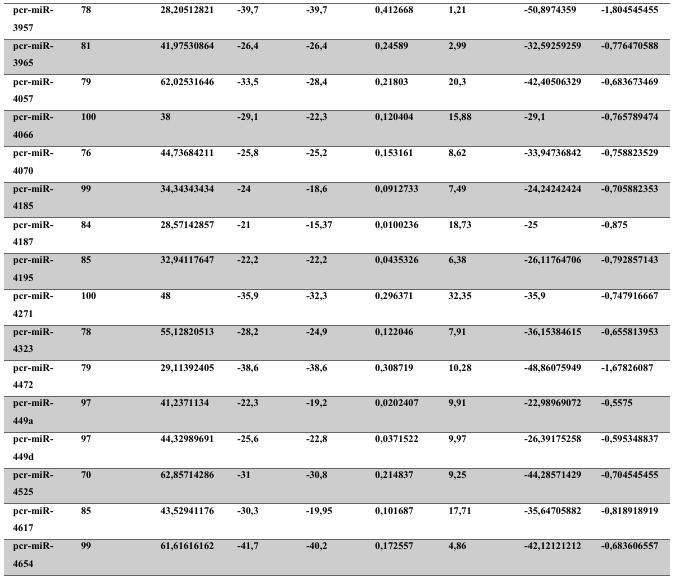
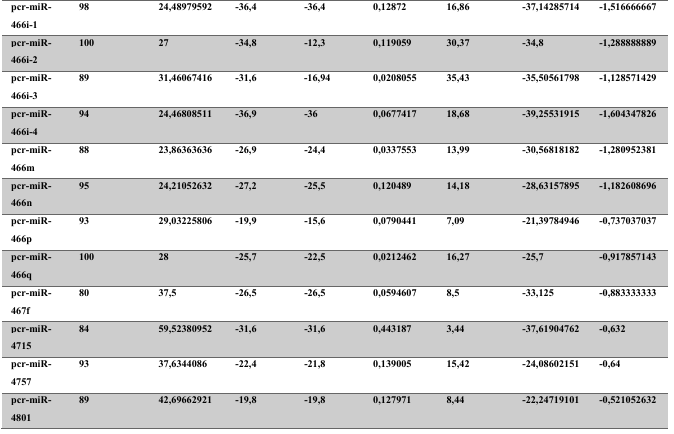
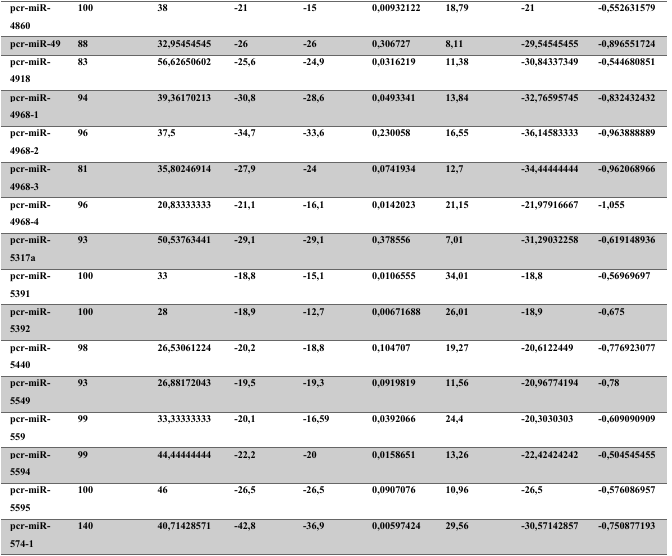
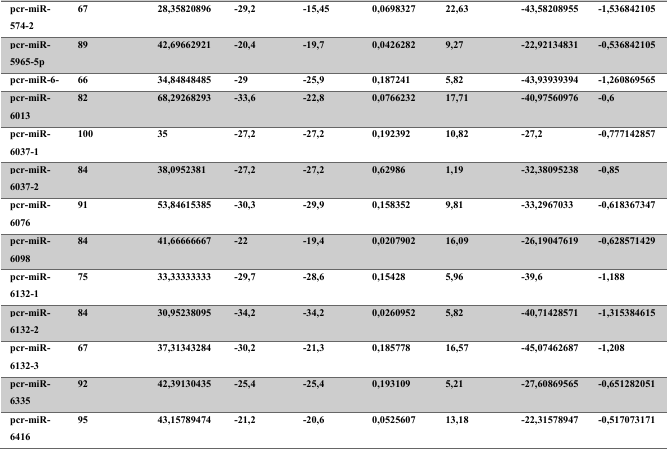
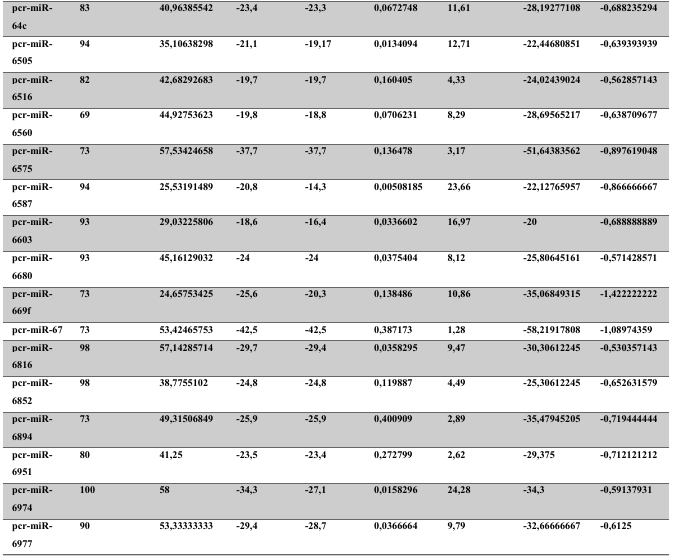
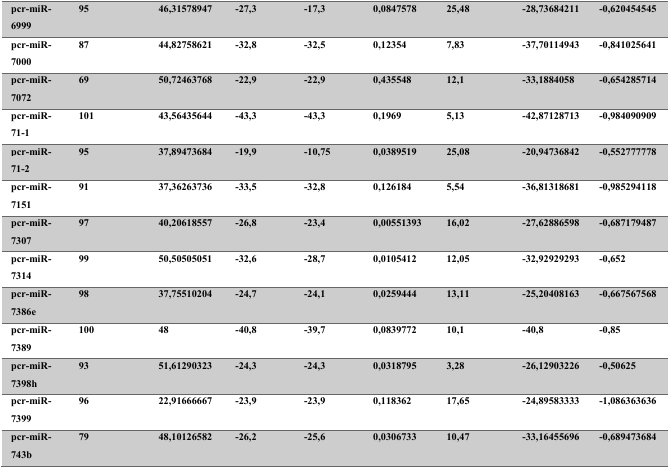
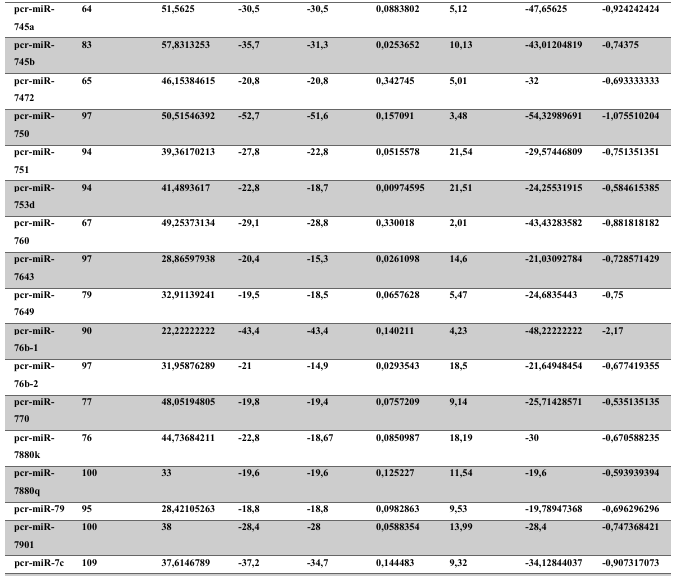
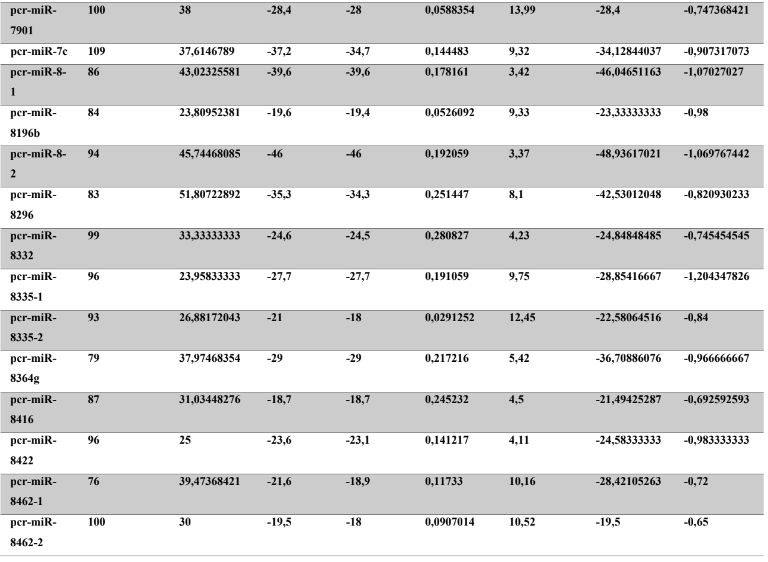
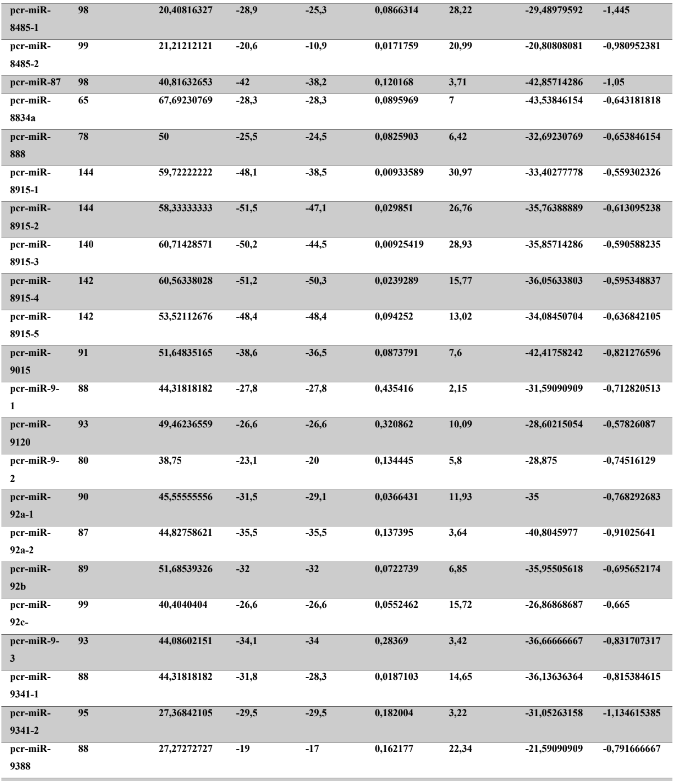
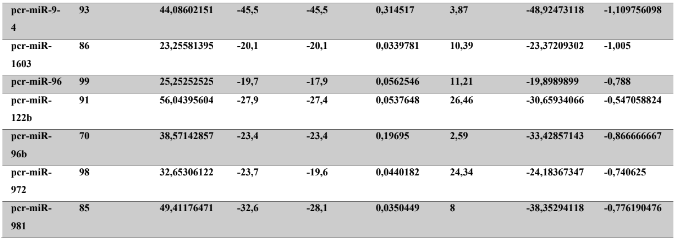
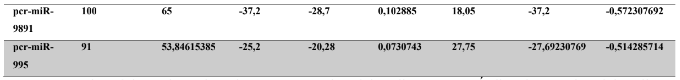
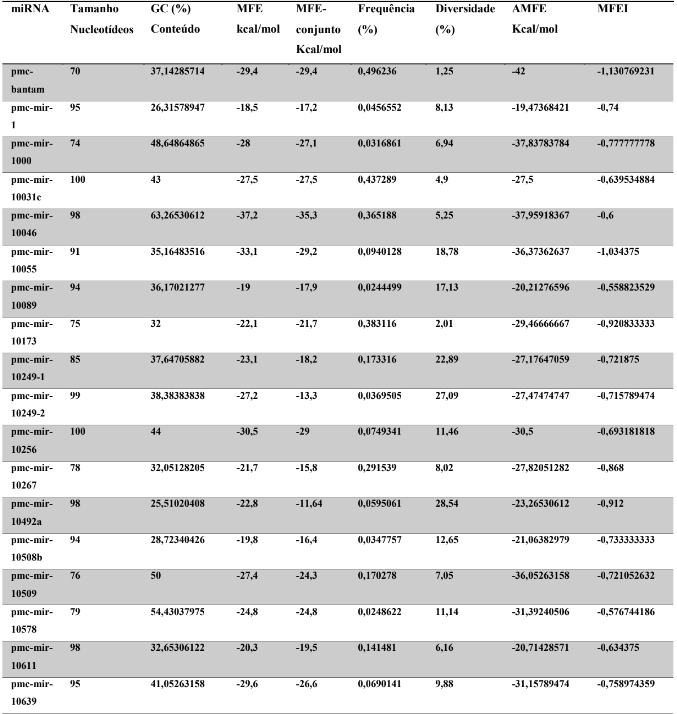
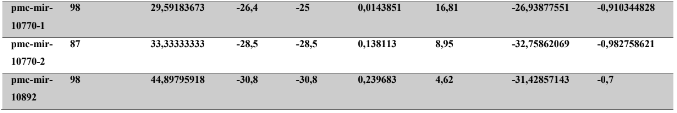
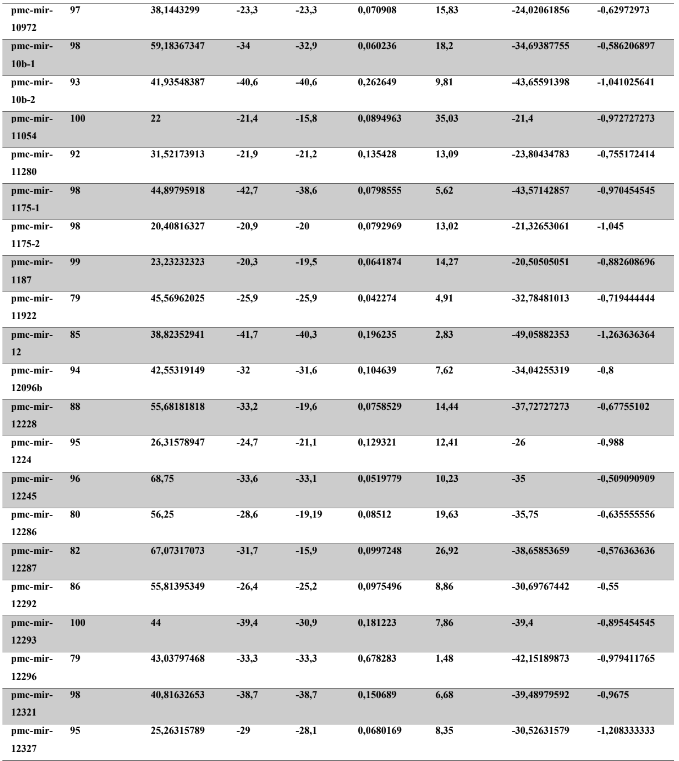
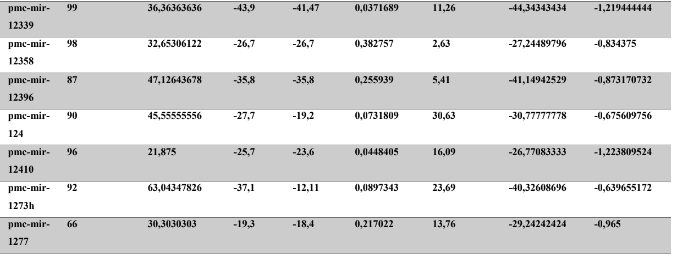
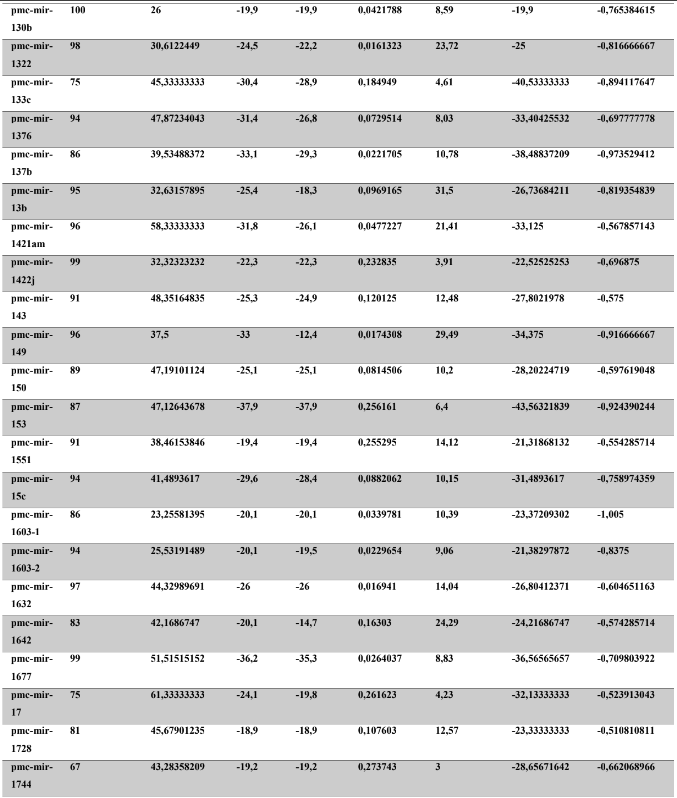
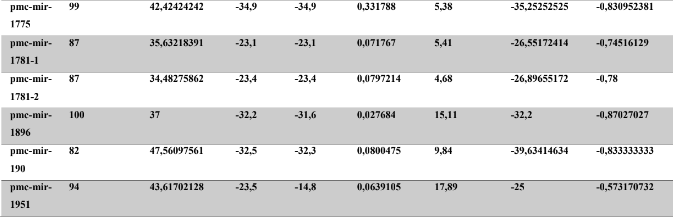
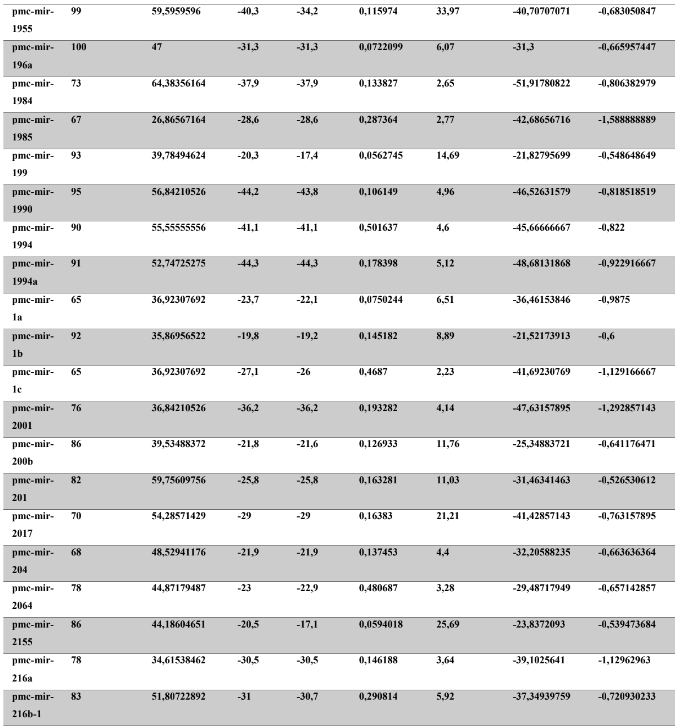
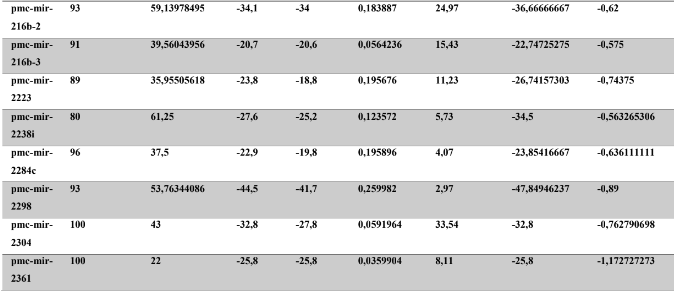
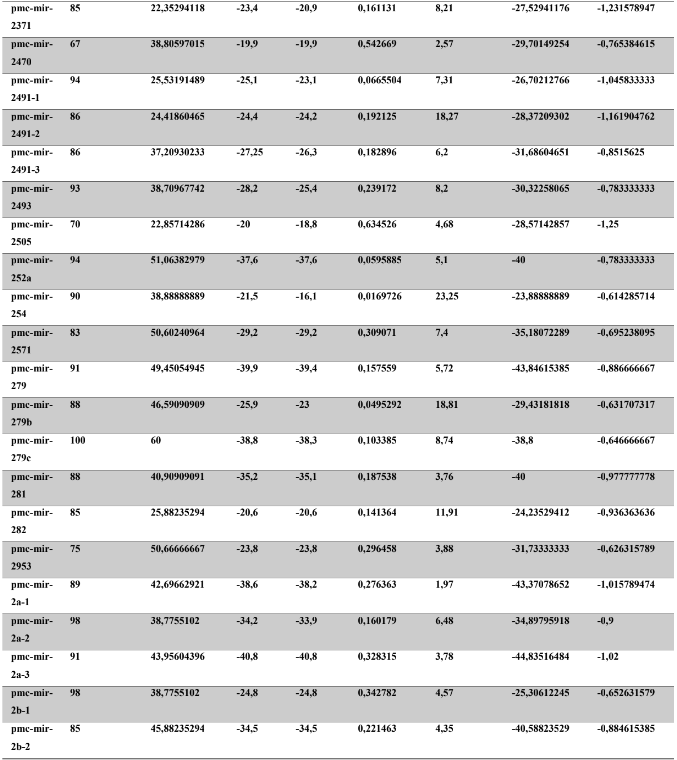
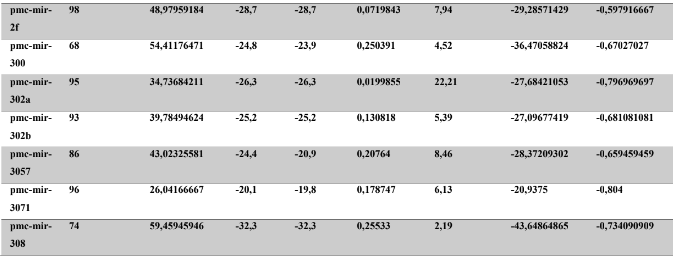
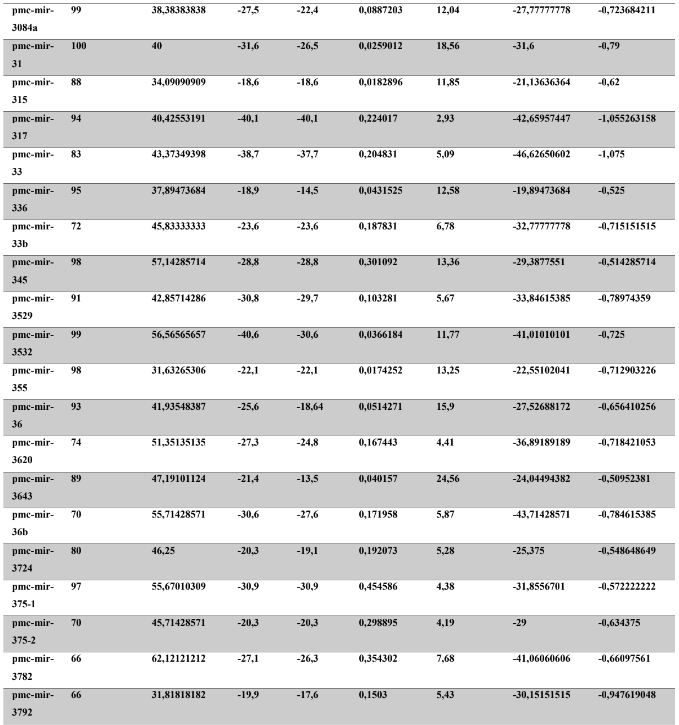
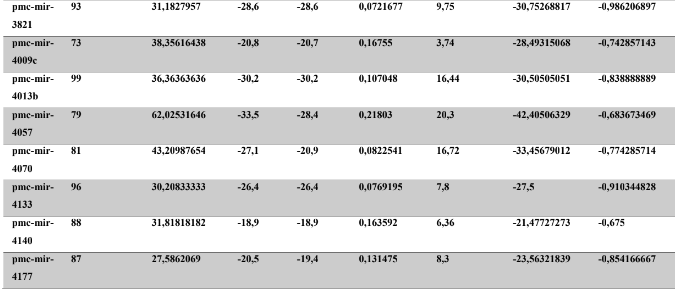
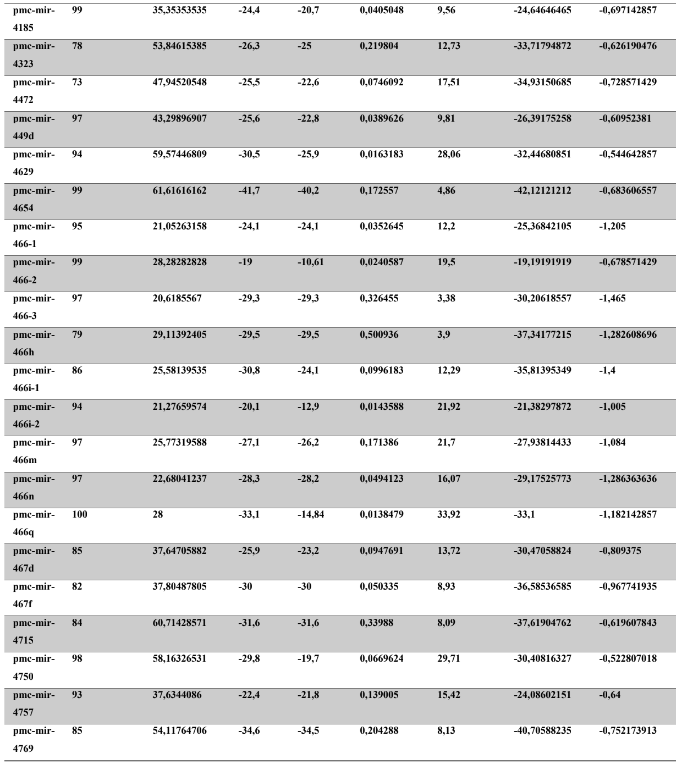
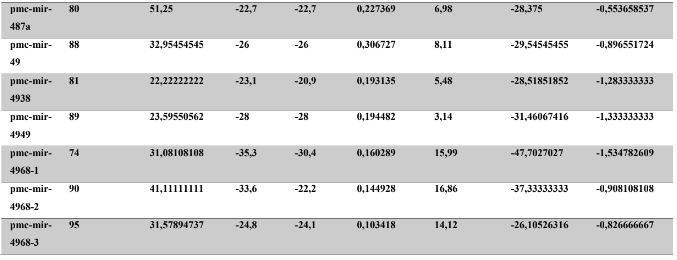
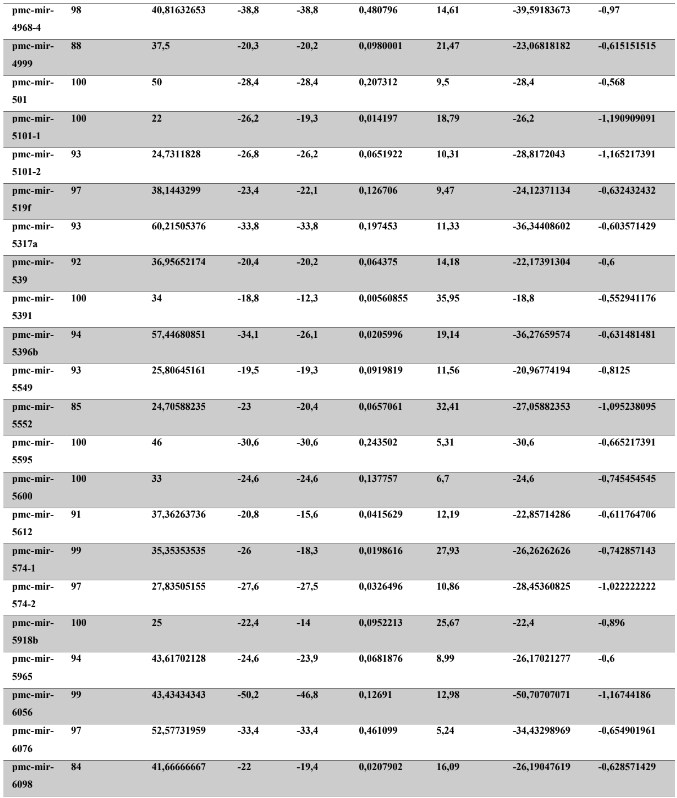
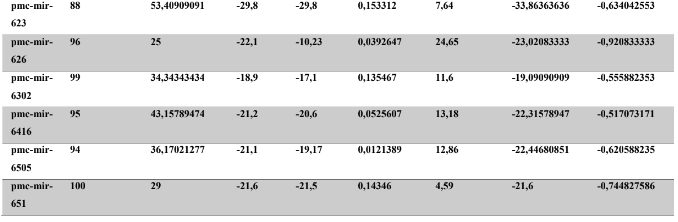
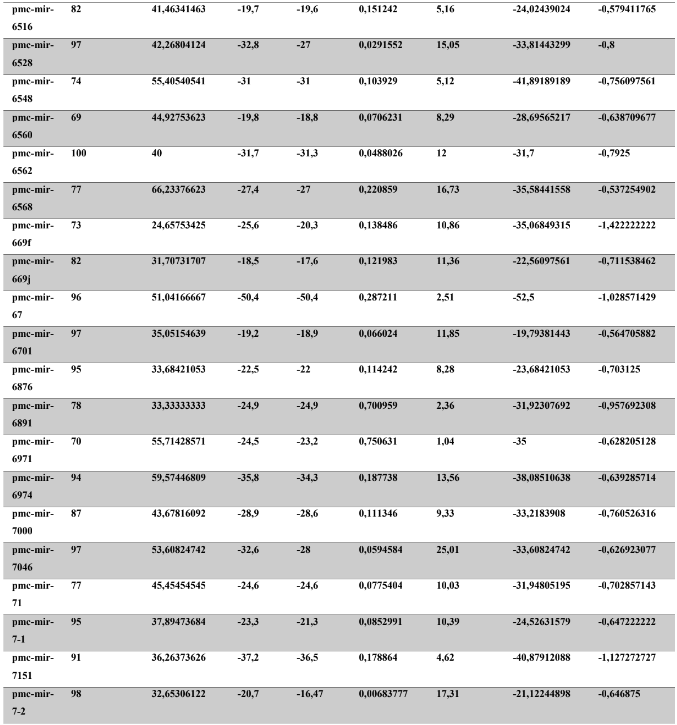
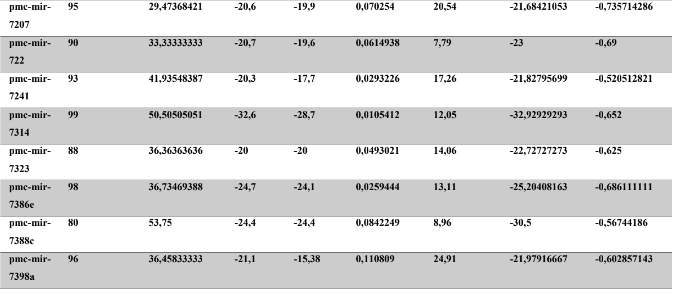
A baixa conservação nucleotídica nos pré-miRNAs restringem as buscas baseadas apenas em métodos de alinhamento de sequências tradicionais (WANG et al., 2005). No entanto, para identificação e caracterização destas moléculas são utilizados alguns parâmetros tais como: energia mínima da estrutura secundária, energia livre ajustada, conteúdo de GC dentre outros (Tabelas 5 e 6) (XUE et al., 2005). A forma mais exata para estudar os pré- miRNAs é feita através de suas características estruturais e termodinâmicas (ZHANG et al., 2009). Para identificação de miRNAs, maduros e precursores, no genoma e transcriptoma de P. canaliculata e P. maculata, as sequências foram analisadas por suas características estruturais e termodinâmicas e processados computacionalmente de forma a encontrar estruturas secundárias semelhantes aos pré-miRNAs (hairpins). As moléculas passaram por diversos filtros como descritos em Materiais e Métodos. Os crivos utilizados foram baseados em características de miRNAs conhecidos (precursores e maduros). Todos os miRNAs identificados obtiveram estrutura secundária estável, ou seja, com energia mínima livre menor que -20 kcal/mol.
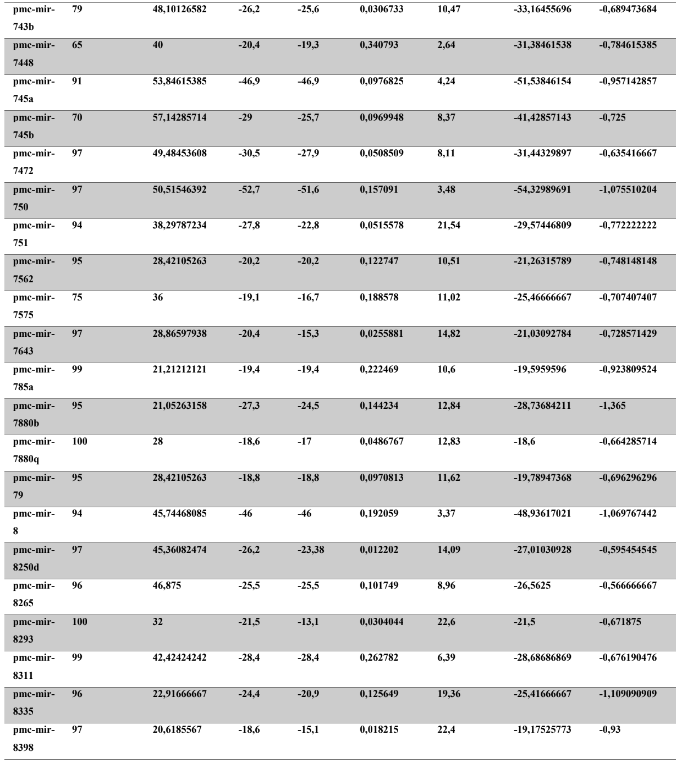
Baseado nestas características, neste trabalho foi possível identificar 269 preditos pré- miRNAs em P. canaliculata e 299 prováveis precursores em P. maculata. O tamanho dos precursores das espécies Lophotrochozoa situa-se entre 57 a 153 nucleotídeos, com uma média de 90 nucleotídeos por sequência (GOMES et al., 2011). No estudo, o tamanho médio dos precursores de P. canaliculada é de 89,55 nucleotídeos e 89,27 nucleotídeos em P. maculata. A variação do tamanho dos pré-miRNAs em P. canaliculata (63 a 144 nucleotídeos) e P. maculata (65 a 100 nucleotídeos) (Tabela 5, coluna 2 e Tabela 6, coluna 2) respectivamente. O tamanho dos pre-miRNAs em espécies animais varia entre 47 a 177 nucleotídeos com um média de 87 ± 29 nucleotídeos (ZHOU et al., 2009). Os dados do presente estudo corroboram com o tamanho médio de pré-miRNAs das espécies Lophochotrozoa e metazoários de forma geral.
Os precursores identificados neste trabalho apresentaram estrutura secundária característica estável, com capacidade de dobramento. No formato de grampo de cabelo (hairpin), o modelo canônico de pré-miRNAs apresenta duas hastes na configuração de dúplex e um contorno na extremidade da sequência. As características termodinâmicas das estruturas secundárias destas moléculas se mostraram dentro dos valores padrões de pré-miRNAS. Conforme observado nas Tabela 5, coluna 4 e Tabela 6, coluna 4 respectivamente, a característica MFE (energia mínima livre) para P. canaliculata apresenta um valor médio de – 28,70 kcal/mol e -27,55 kcal/ mol para P. maculata, sendo valor próximo quando comparado as espécies Lophotrochozoa (-31,27 kcal/mol). Os parâmetros estruturais como a porcentagem de GC (conteúdo) também não apresentaram diferenças estatísticas; P. canaliculata, P. maculata e espécies Lophotrochozoa apresentam média de 41,70% , 40,74% e 40,49% respectivamente (GOMES et al., 2011). Dessa forma, verifica-se que as características estruturais e termodinâmicas dos pré-miRNAs de P. canaliculata e P. maculata são conservadas, quando comparados com miRNAs de espécies identificadas corroborando achados relacionados aos pré-miRNAs das espécies em estudo.
Tabela 5: Características estruturais e termodinâmicas dos pré-miRNAs de P. canaliculata
Fonte: Dados do autor.
Tabela 6: Características estruturais e termodinâmicas dos pré-miRNas de P. maculata.

Fonte: Dados do autor
Através de uma abordagem computacional desenvolvida neste trabalho, no genoma e transcriptoma das espécies em estudo, foi possível detectar miRNAs (maduros e precursores).
Foram identificados 296 (P. canaliculata) e 316 (P. maculata) prováveis miRNAs maduros conservados, semelhantes aos ortólogos das espécies com miRNAs catalogados no miRbase versão 22 (http://www.mirbase.org). O número de miRNAs identificados corrobora com o número de miRNAs conservados referentes ao molusco próximo evolutivamente Lymnaea stagnalis, gastrópode de água doce (264 miRNAs maduros) (WALKER et al., 2018).
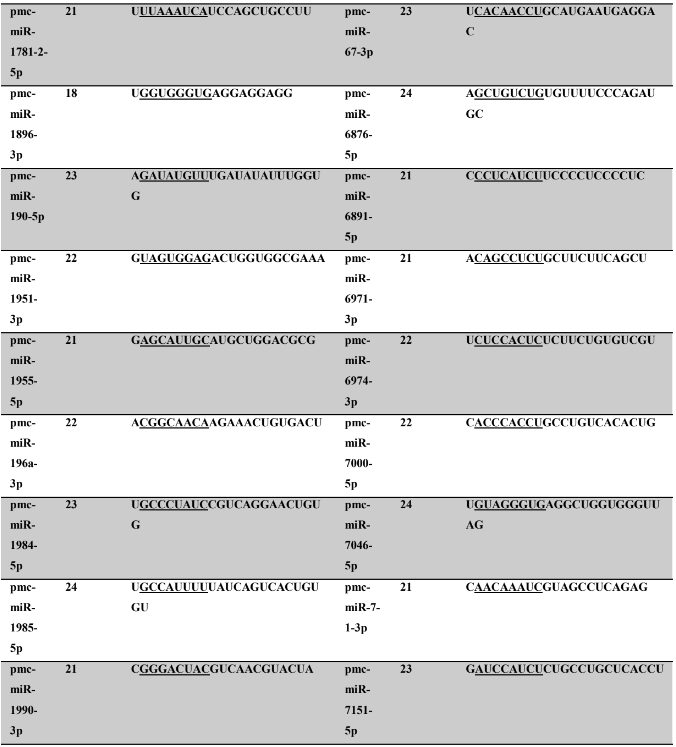
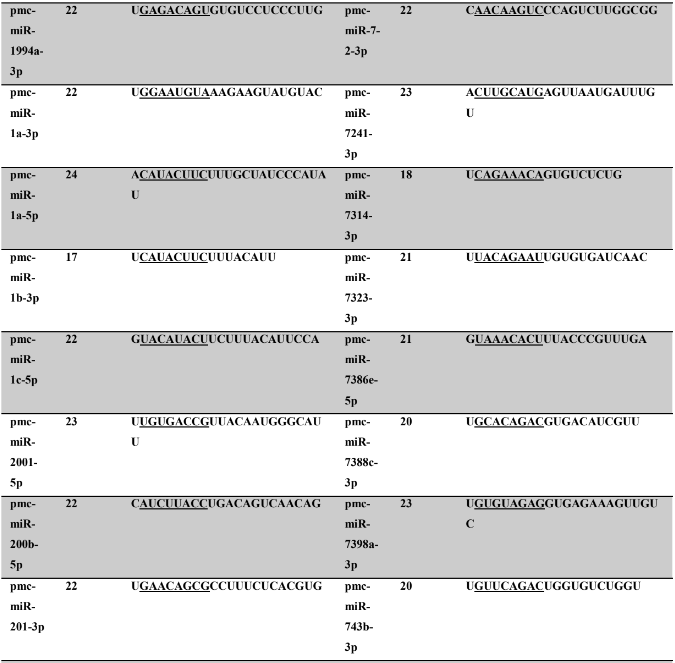
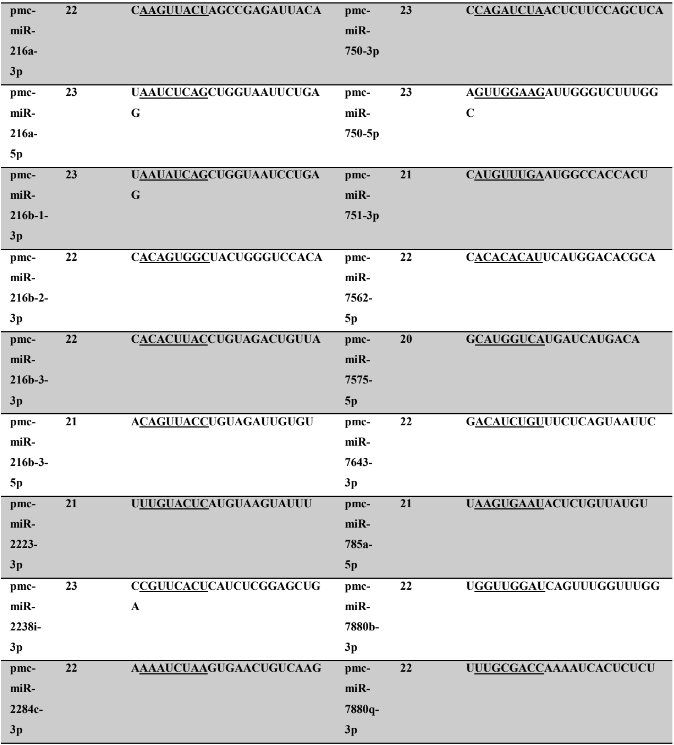
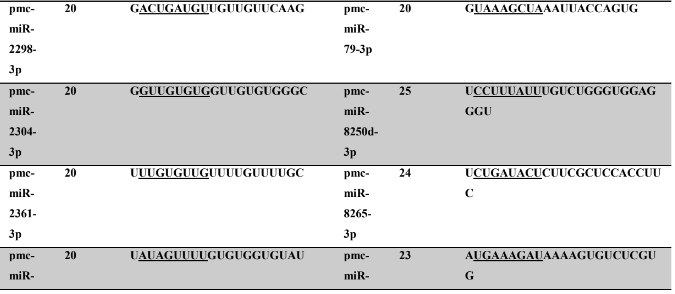
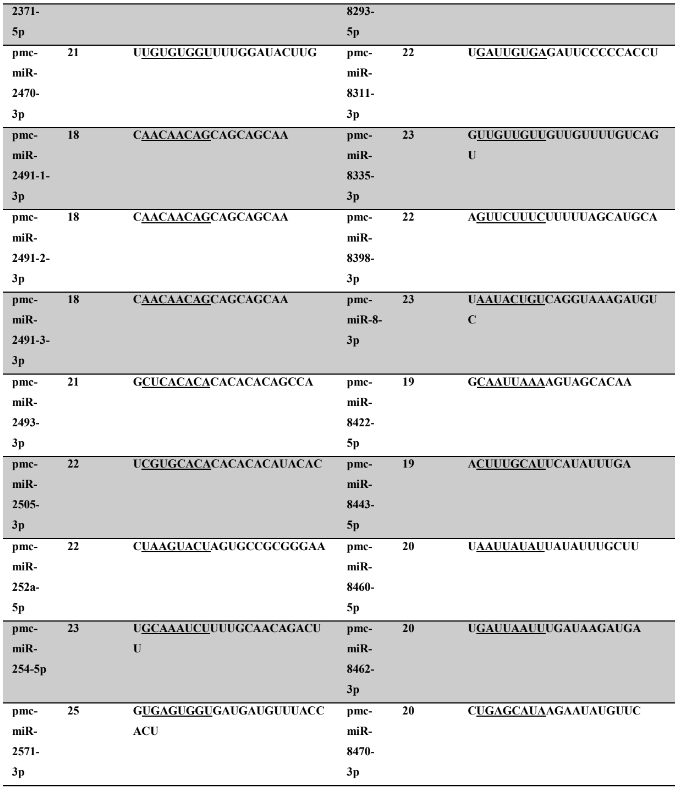
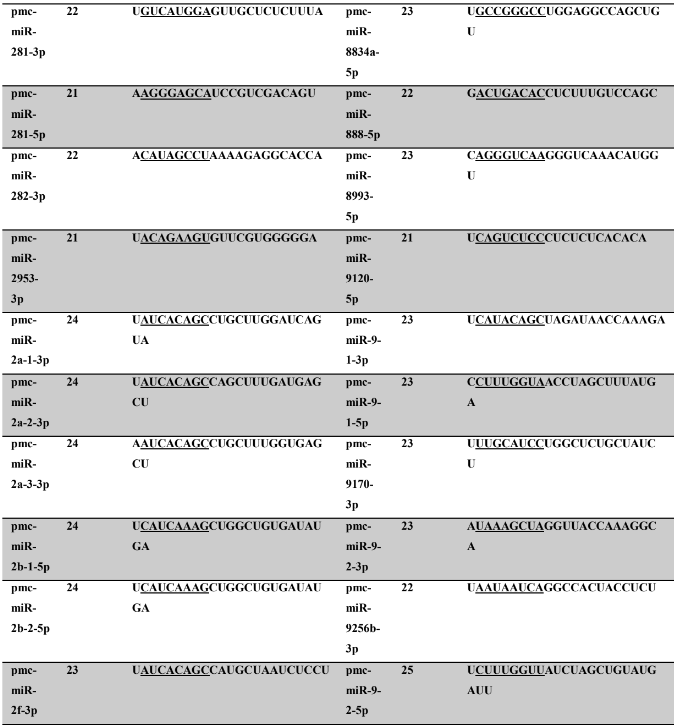
Os miRNA não codificantes de proteínas têm aproximadamente 22 nucleotídeos em sua extensão, variando em média de 17 a 25 nucleotídeos (HA; KIM, 2014; RACHAGANI; KUMAR; BATRA, 2010). Os miRNAs maduros identificados neste trabalho possuem em média 21,86 nucleotídeos (P. canaliculata) – Tabela 7, coluna 2 e 21,77 nucleotídeos (P. maculata) – Tabela 8, coluna 2; com variação de tamanho entre 17 a 25 nucleotídeos para ambas as espécies, corroborando com dados de espécies animais.
A proteína Ago1 apresenta preferência pelo nucleotídeo uracila na primeira posição da sequência do miRNA na extremidade 5’ a ser carregado, para ser inserido no complexo RISC. A proteína Ago 2 também pode ser carregada com o miRNA (uma das fitas do duplex), intermediando a clivagem de alvos de miRNAs (GHILDIYAL et al., 2010; SEITZ; TUSHIR; ZAMORE, 2011; ZHANG; STELLWAG; PAN, 2009). Neste trabalho, os miRNAs maduros de P. canaliculata e P. maculata, respectivamente, apresentaram quantitativo de nucleotídeos na primeira posição de 41,2% e 42,40% de uracila, 20,9% e 17,8% (citosina), 20,4% e 24,5% de adenina e 17,5% e 15,3% de guanina. Os resultados encontrados corroboram os dados da literatura acima descritos.
A sequência madura dos miRNAs tem se mostrado conservada evolutivamente (WIT et al., 2009). Esta conservação se acentua na região denominada seed (semente), sendo considerada importante para reconhecimento dos alvos de miRNAs (LEWIS; BURGE; BARTEL, 2005). Todos os miRNAs identificados neste trabalho apresentaram 100% de identidade nas regiões seed (posição de 2 a 8 nucleotídeos) (LEWIS et al., 2003) e no máximo 30% dos nucleotídeos diferentes (mismathes) do miRNA maduro, quando comparado com estrutura do seu respectivo ortólogo.
A conservação da região seed, segundo Friedman et al. (2009) é muito importante evolutivamente para conservação dos alvos de miRNAs silenciados. Como ocorre a correspondência de bases do tipo Watson-Crick, ambos, miRNAs e alvos, necessitam de uma estrutura conservada, apesar de que todas as interações não sejam totalmente complementares (FRIEDMAN et al., 2009). Na caracterização dos miRNAs, é possível verificar no alinhamento dos precursores a identificação dos miRNAs, apresentando 100% de identidade nas regiões seed. Todos os miRNAs catalogados neste trabalho possuem um ortólogo correspondente no qual a busca foi baseada.
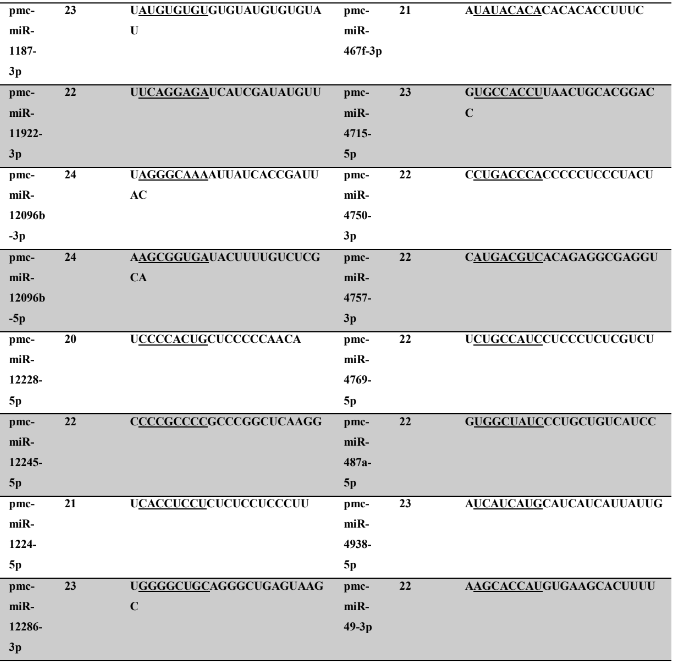
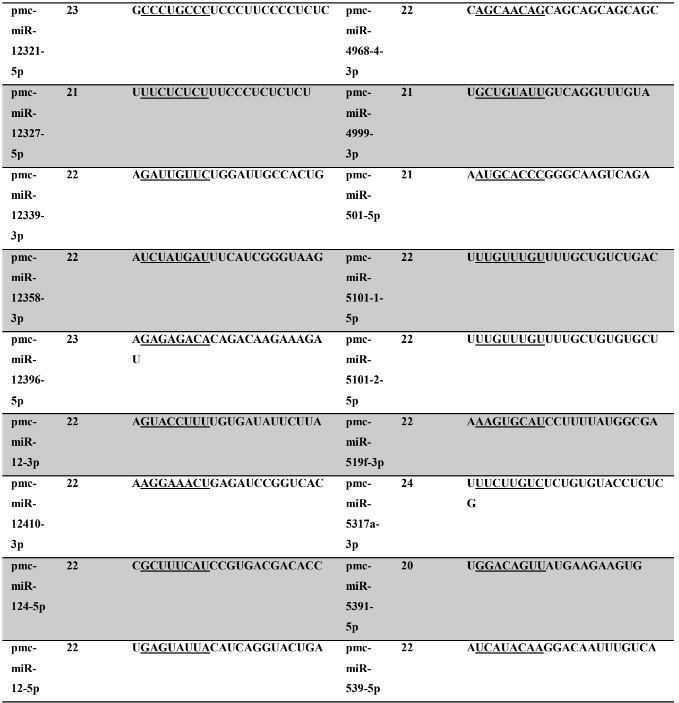
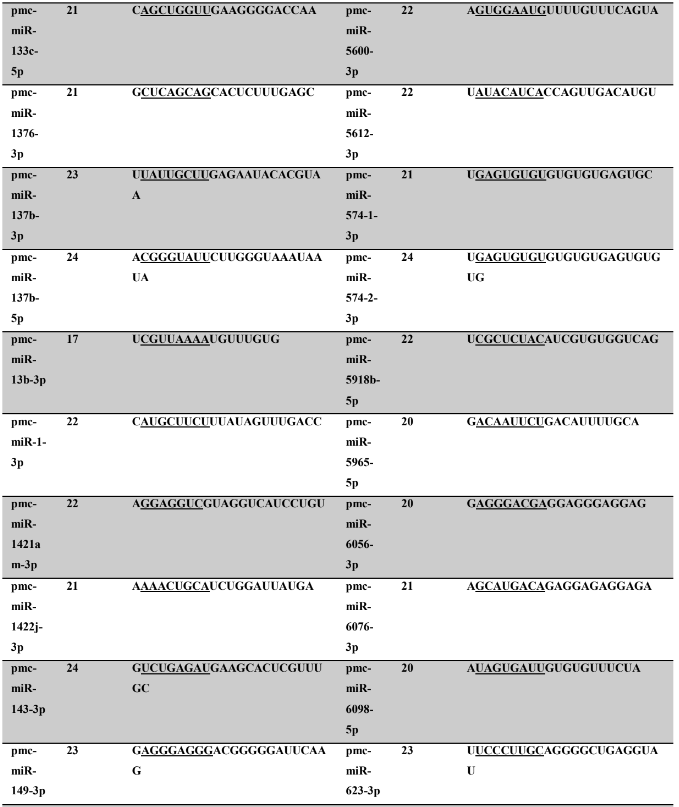
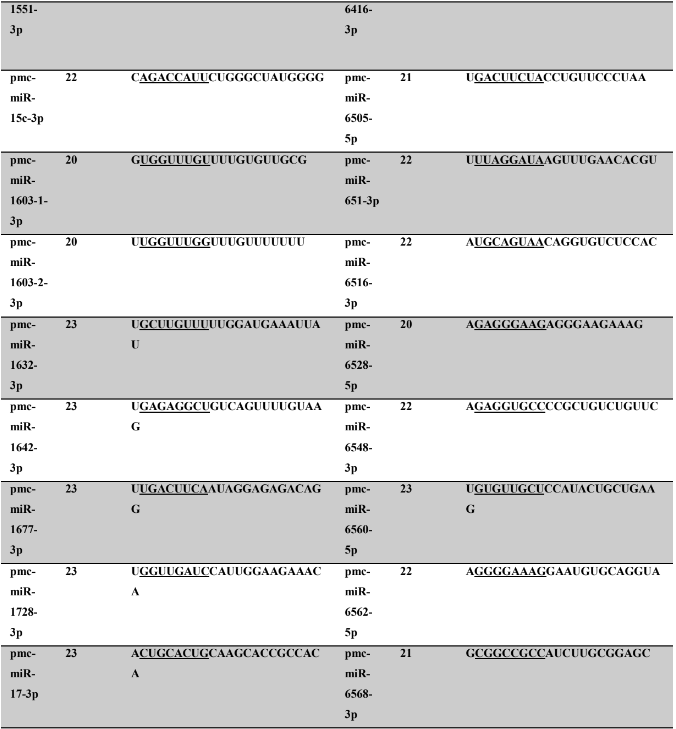
Tabela 7: Prováveis miRNAs maduros conservados identificados em P. canaliculata, tamanho dos nucleotídeos (nt), sequência nucleotítica do miRNA maduro, região seed.
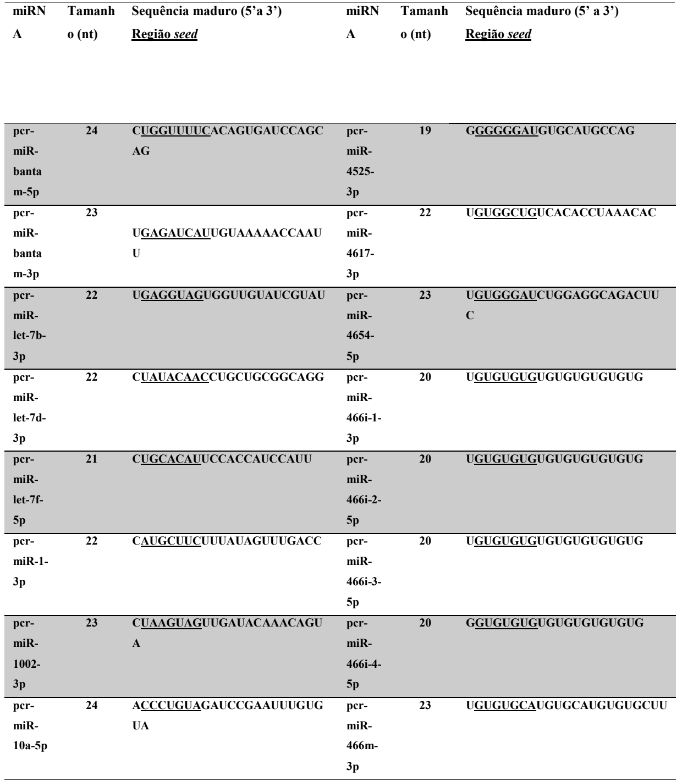

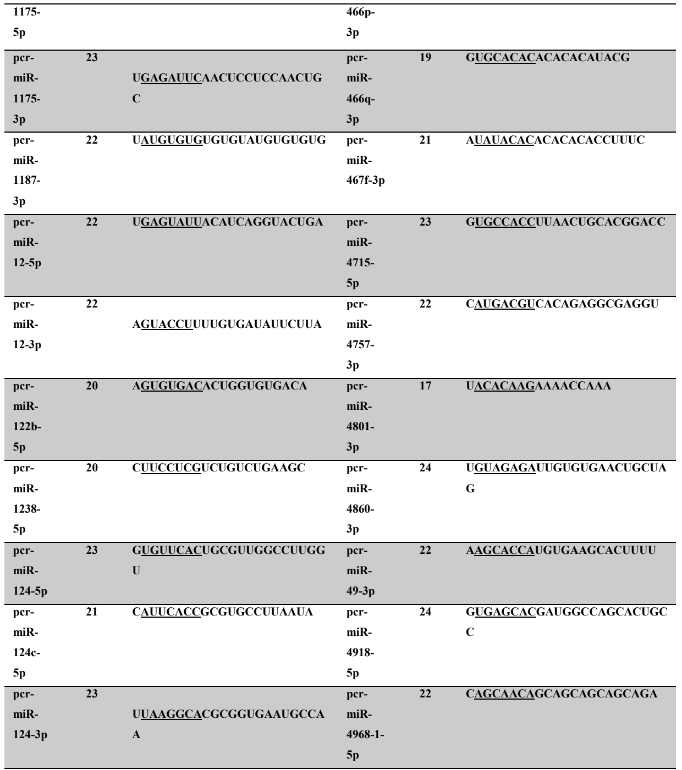

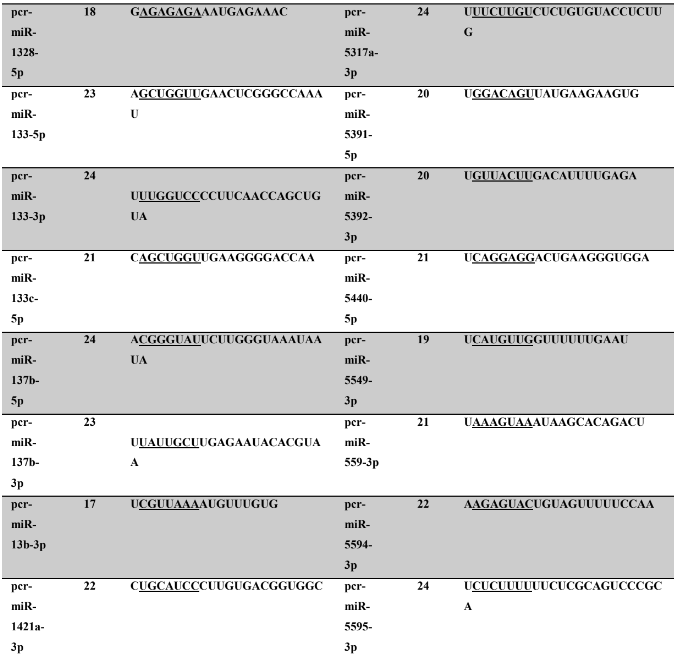

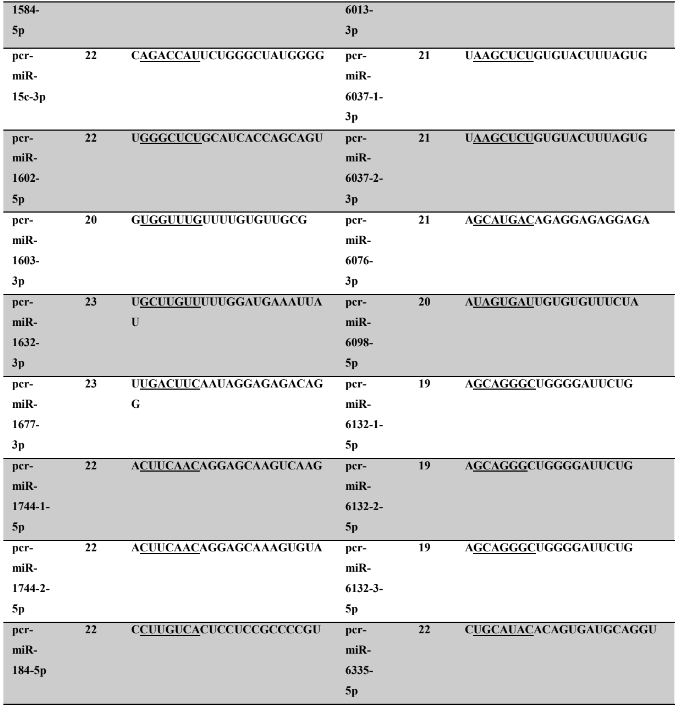

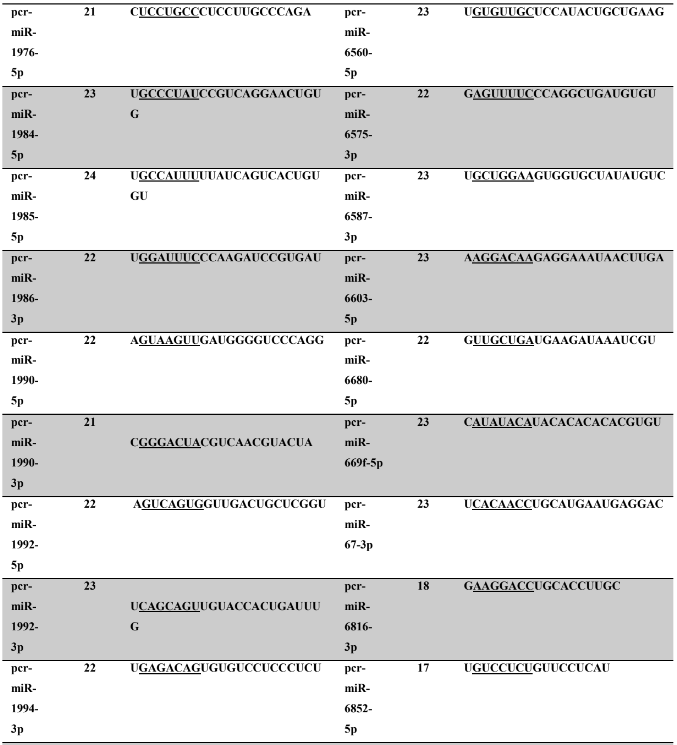

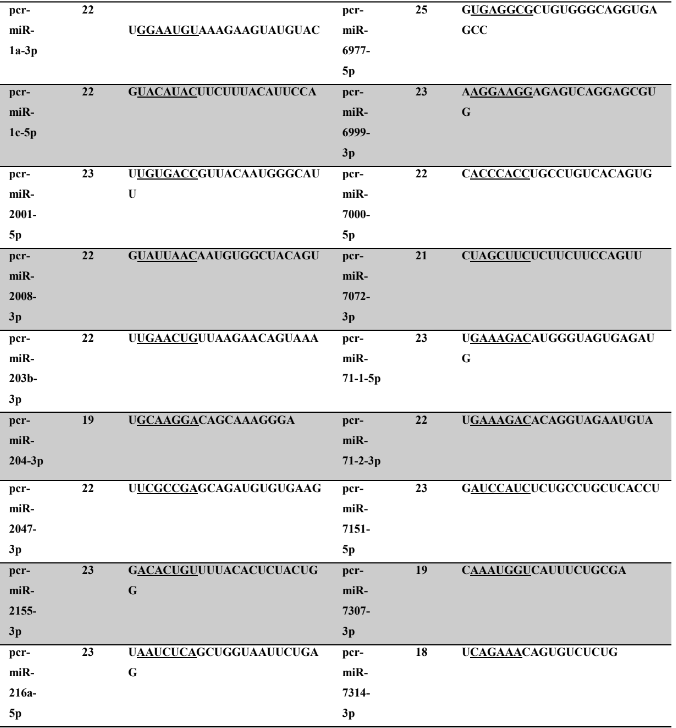

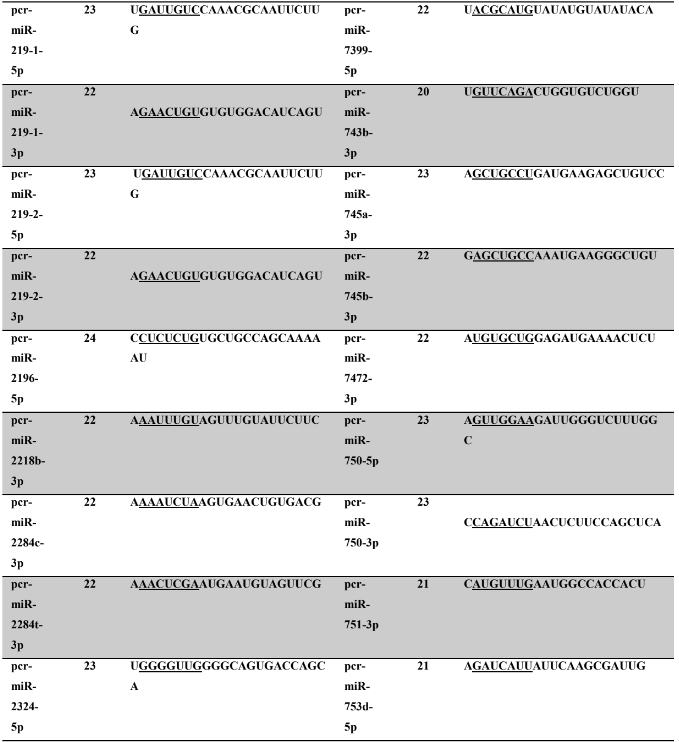
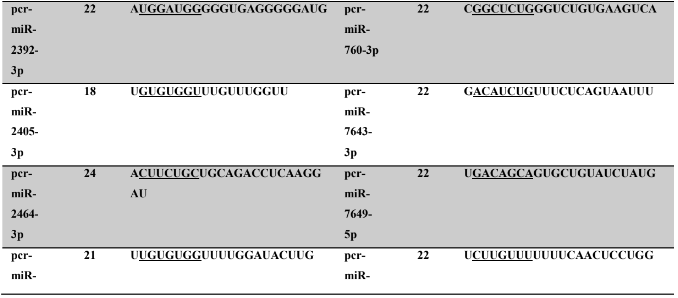
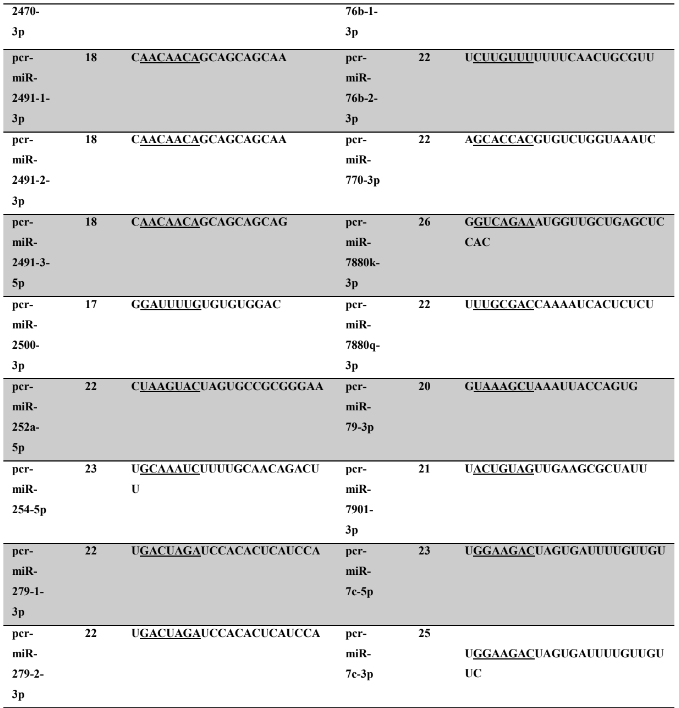
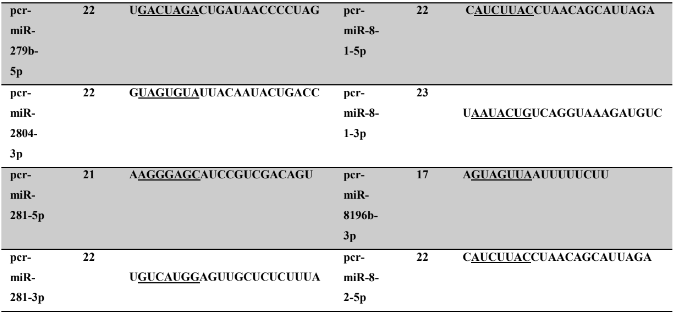
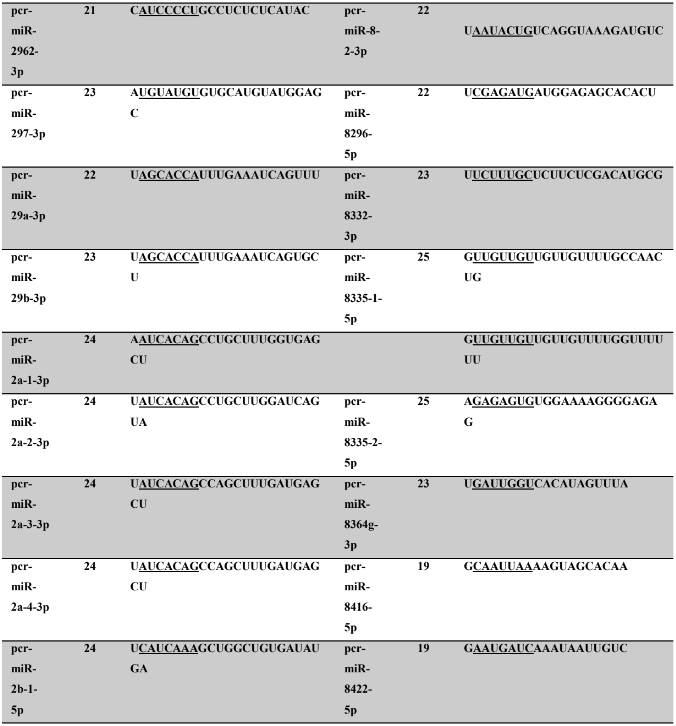
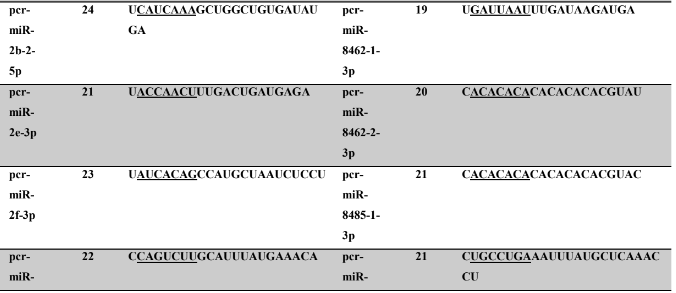
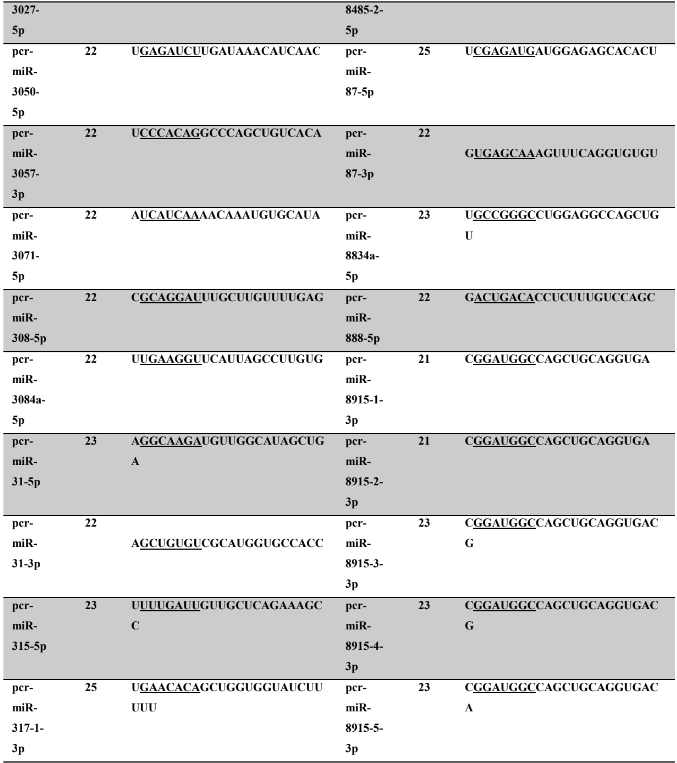

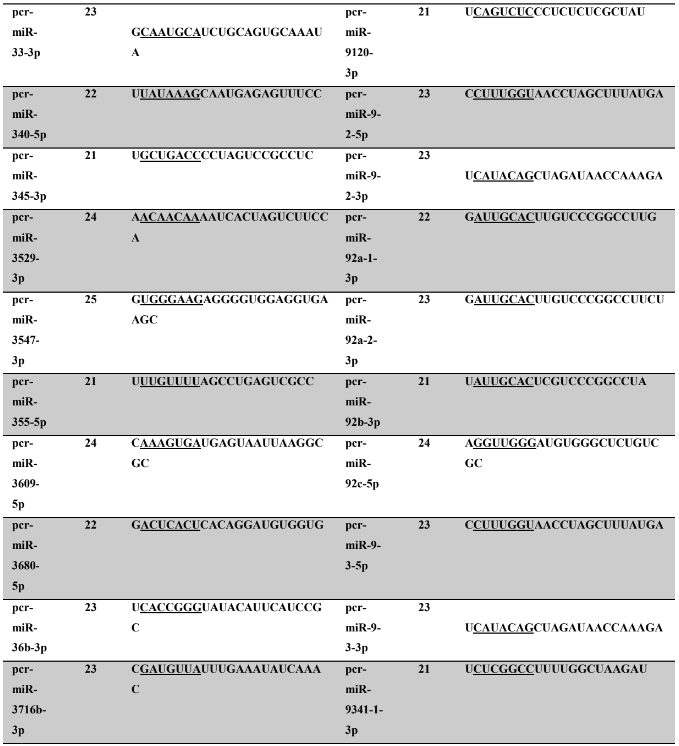

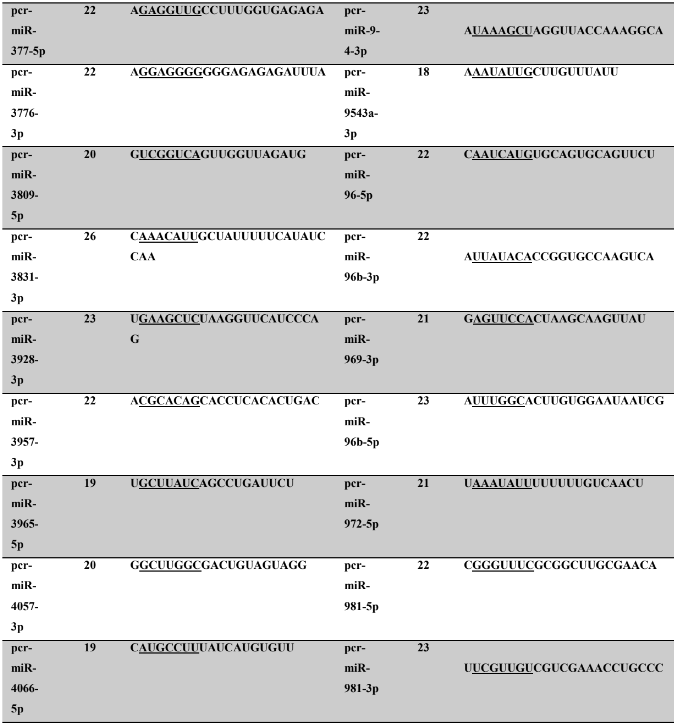
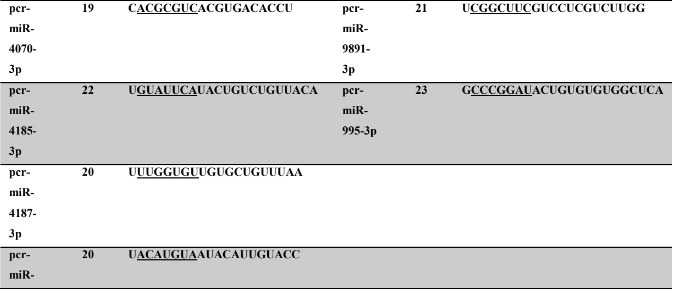

Tabela 8: Prováveis miRNAs maduros conservados identificados em P. maculata, tamanho dos nucleotídeos (nt), sequência nucleotídica do miRNA maduro, região seed em destaque.

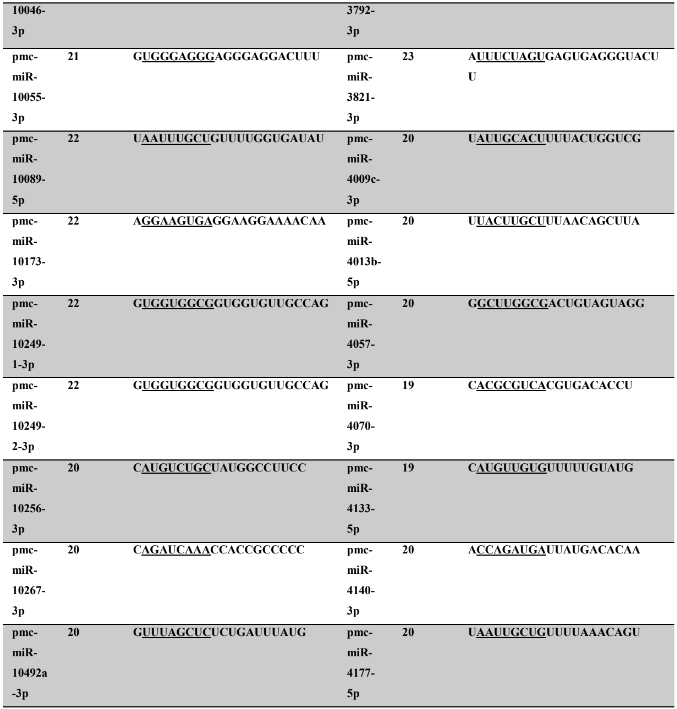
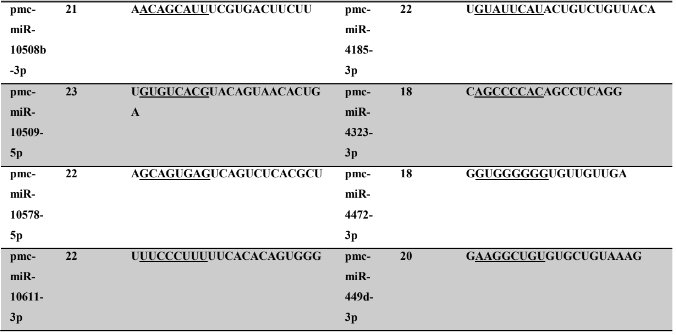
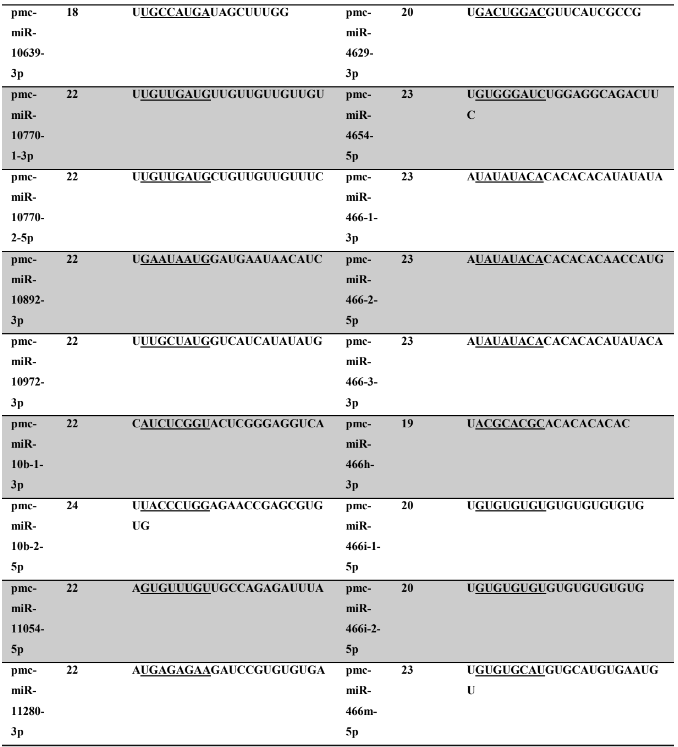









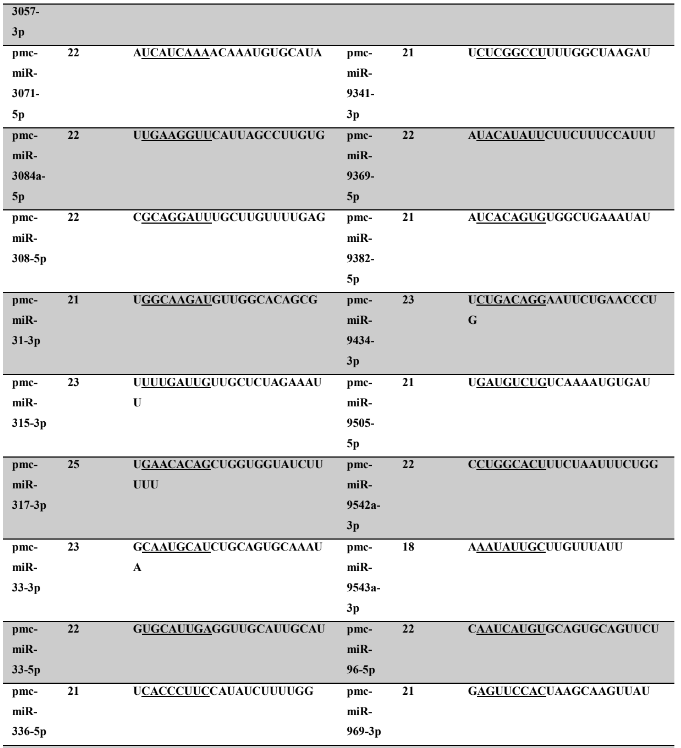


A partir do levantamento dos miRNAs das espécies com seus respectivos ortólogos, foram selecionados miRNAs que refletissem a seletividade por alguns táxons, corroborando assim para a ideia de conservação evolucional da estrutural dos precursores e miRNAs. Os táxons que foram utilizados como parâmetro de seleção de miRNAs específicos foram: Bilatéria, Protostômios, Lophotrocozoa e Mollusca.
Dentre estes miRNAs, buscou-se a caracterização de miRNAs bilatéria específicos, protostômio específicos, lophotrochozoa específicos e molusco específicos. As seguintes espécies foram utilizadas para análises; Lanistes nyassanus (lny), Marisa cornuarietis (mcr), Pomacea canaliculata (pcr), Pomacea maculata (pmc); animais do clado bilatéria, Melibe leonina (mle), Lottia gigantea (lgi), miRbase versão 22 (http://www.mirbase.org); todos os moluscos analisados com exceção dos acima citados (HUANG et al., 2021).
4.3 miRNAs BILATERIA ESPECÍFICOS
4.3.1 mir-9-1
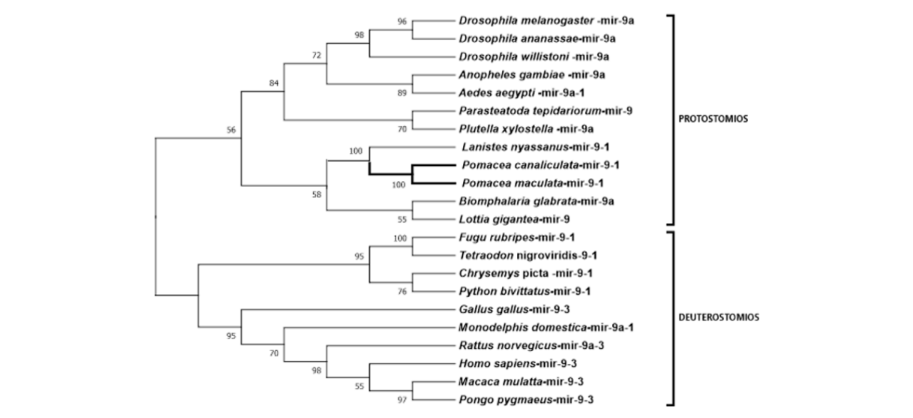
A família dos genes de mir-9 é responsável por regular a neurogênese, tendo papel no desenvolvimento cerebral, em linhagens de células neurais e não neurais. A sequência deste miRNA maduro é idêntico em insetos e humanos, revelando sua preservação nucleotídica entre protostômios e deuterostômios. Em deuterostômios há uma preferência para utilização da fita 5p no complexo RISC; no entanto, no molusco marinho gastrópode Aplysia californica ambas as fitas são igualmente utilizadas para induzir o silenciamento da expressão gênica (YUVA- AYDEMIR et al., 2011). Os precursores de mir-9-1 foram encontrados no genoma das espécies P. maculata e P. canaliculata, com a presença de dois miRNAs maduros por espécie (Figura 5). Observa-se alta similaridade da estrutura secundária dos precursores de P. canaliculata, P. maculata, L. nyassanus e M. cornuarietis (Figura 6). Foi verificado ampla distribuição entre os clados filogenéticos da família mir-9, com a presença de mir-9-1 de P. canaliculata e P. maculata agrupados com o ampularídeo M. cornuarietis no filo dos moluscos (Figura7). Estes dados corroboram para identificação de mir-9-1 nos dois ampularídeos em estudo.
Figura 5: Alinhamento de precursores de miRNA-9-1 com ortólogos.

Figura 6: Estrutura secundária de precursores de miRNA-9-1 e ortólogos; spu: Strongylocentrotus purpuratus, lgi: Lottia gigantea,lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata, pmc: Pomacea maculata
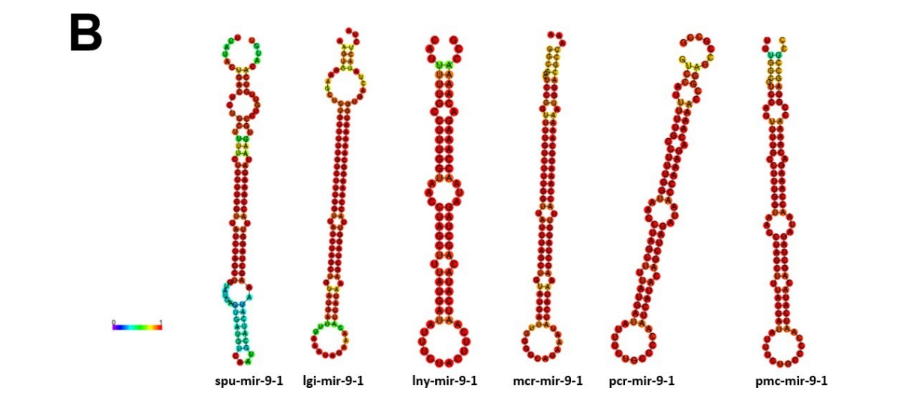
Figura 7: Distribuição filogenética de miR-9-1 (bilatéria-especifico).
4.3.2 mir-216a
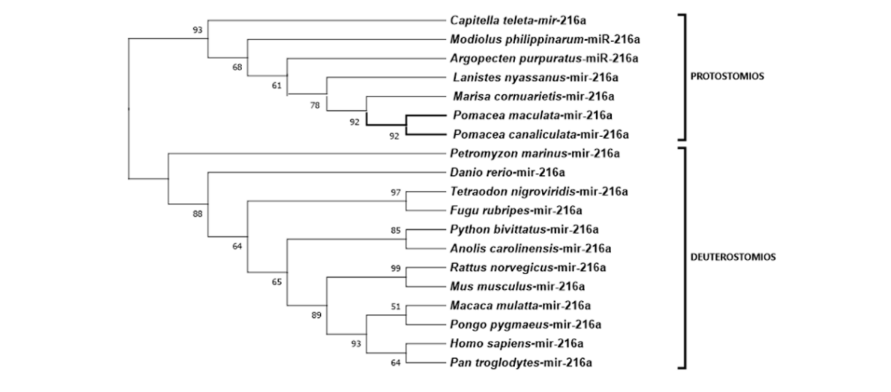
A família dos genes de mir-216a está relacionada a processos patológicos em deuterostômios, especificamente em humanos. O mir-216a-3p inibiu a viabilidade, migração, invasão e proliferação de células de câncer de pulmão em humanos (WANG et al., 2020). Já o mir-216a-5p foi reconhecido como um oncogene e está envolvido na progressão e metástase de vários subtipos de câncer (CHEN et al., 2018). Nos organismos protostômios, a família do mir- 216a foi encontrada no genoma de 18 espécies de moluscos, dentre estes 7 gastrópodes. (HUANG et al., 2021). Isso corrobora para a identificação dos precursores de mir-216 em P. canaliculata e P. maculata, com a presença de dois miRNAs maduros (cadeias 3p e 5p) por espécie (Figura 8). A estrutura secundária dos precursores de P. canaliculata e P. maculata apresentaram alta similaridade com seus ortólogos, principalmente com L. nyassanus e M. cornuarietis (Figura 9). Foi verificado ampla distribuição filogenética da família de mir-216a, com mir-216a de P. canaliculata e P. maculata apresentando-se agrupados (Figura 10).
Figura 8: Alinhamento de precursores de miRNA-216a com ortólogos.

Figura 9: Estrutura secundária de precursores de mir-216a e ortólogos; mle; Melibe leonina, lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata, pmc: Pomacea maculata, rno; Rattus norvegicus.
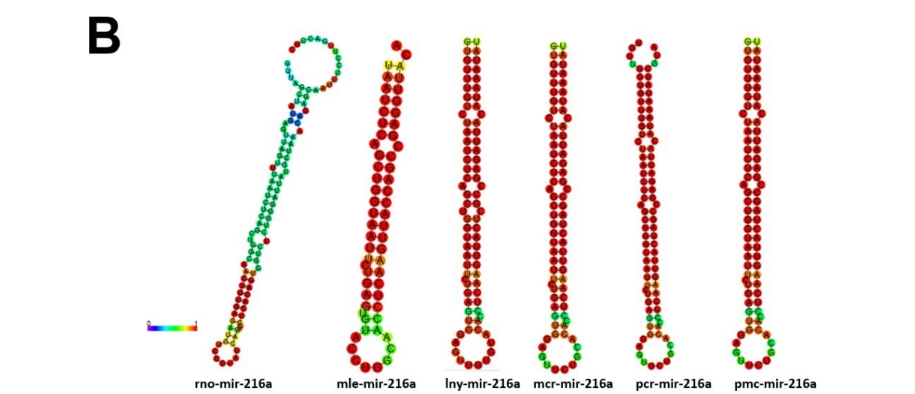
Figura 10: Distribuição filogenética de mir-216a (bilatéria-especifico).
4.3.3 mir-133
A família dos genes mir-133 é expressa em tecido muscular, sendo responsáveis pela remodelação cardíaca em humanos e diferenciação miocárdica em camundongos (LI; ZHOU; TANG, 2018; TAKAYA et al., 2009). Os homólogos de mir-133 foram identificados em diversas espécies, dentre estas os invertebrados como a Drosophila melanogaster (CHEN et al., 2006). O mir-133 têm ampla distribuição filogenética entre protostômios e deuterostômios, conforme verificado na Figura 13 deste trabalho. Em moluscos, na ostra Crassostrea gigas, o mir-133 mostrou-se ser responsivo ao estímulo de noradrenalina, sendo este miRNA responsável pela modulação do sistema imunológico nestes animais (CHEN et al., 2015). A família dos genes de mir-133 foi encontrada no genoma de P. maculata na isoforma mir-133c. Já para P. canaliculata foi verificado na espécie dois miRNAs maduros no precursor pcr-mir- 133 (Figura11). Observa-se extrema similaridade da estrutura secundária dos precursores de mir-133 entre organismos da família Ampullariidae (L. nyassanus, M. cornuarietis, P. canaliculata) e também destes com L. gigantea (Figura 12).
Figura 11: Alinhamento de precursores de miRNA-133 e ortólogos, em destaque miRNAs maduros.
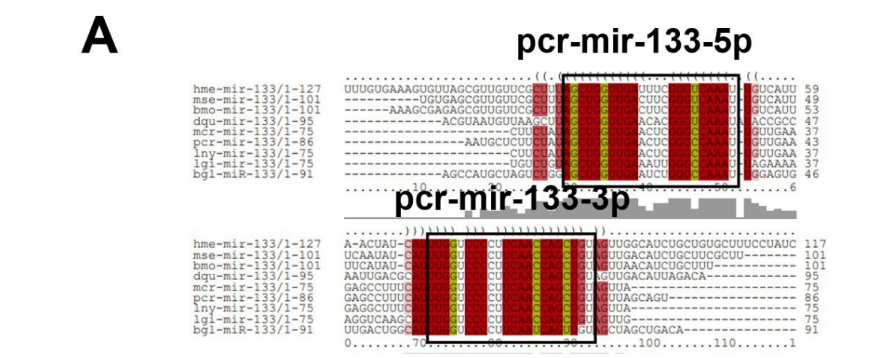
Figura 12: Estrutura secundária de precursores de miRNA-133 e ortólogos; hme: Heliconius melpomene, bmo: Bombyx mori, lgi: Lottia gigantea, lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata.
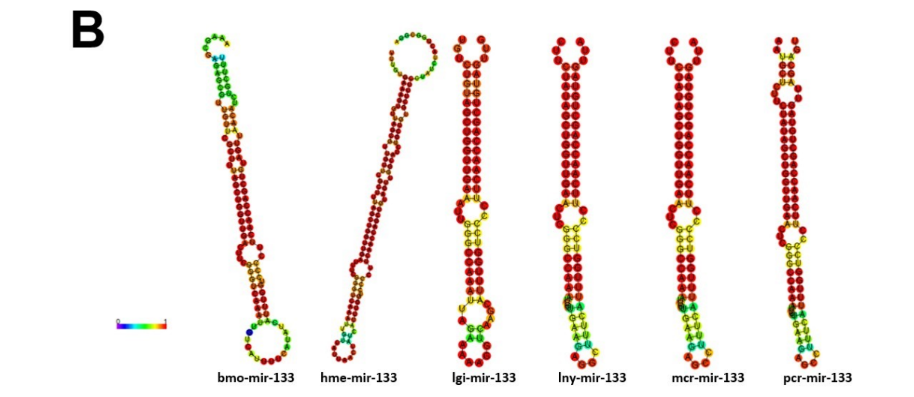
Figura 13: Distribuição filogenética de mir-133 (bilatéria-específico).
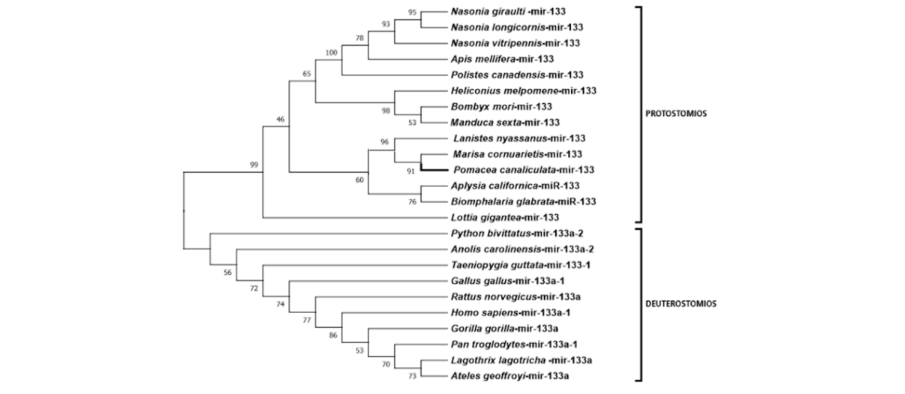
4.3.4 mir-33
Os precursores do mir-33 foram encontrados neste estudo: pcr-mir-33, pmc-mir-33 (Figura 14). Os precursores localizados nas 2 espécies apresentaram dois miRNAs maduros cada, 3p e 5p, corroborando estudos observados em moluscos bivalves e D. melanogaster (BAO et al., 2014; CLERBAUX et al., 2021). Foi realizada a predição das estruturas secundárias dos pré-miRNAs e seus ortólogos (Figura 15). O mir-33-5p supostamente regula os genes envolvidos na resposta ao estresse induzido por metais pesados no molusco Tegillarca granosa (BAO et al., 2014). Da mesma forma, P. canaliculata é capaz de sobreviver em ambientes com metais pesados, onde há alta concentração desses metais nas brânquias (KRUATRACHUE et al., 2011), provavelmente regulados por este micro-RNA. Além disso, miR-33 é um regulador pleiotrópico de processos metabólicos e de desenvolvimento em D. melanogaster. Sugere-se que o mir-33 seja um regulador conservado da homeostase lipídica (CLERBAUX et al., 2021). A árvore filogenética mostrou a ocorrência do miRNA em Deuterostômios (peixes e mamíferos), bem como em Protostômios (Ecdysozoa e Lophotrocozoa) (Figura 16).
Figura 14: Alinhamento de precursores de miRNA-33 com ortólogos.

Figura 15: Estrutura secundária de precursores de miRNA-133 e ortólogos; nvi: Nasonia vitripennis, lgi: Lottia gigantea, lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata, pmc: Pomacea maculata.
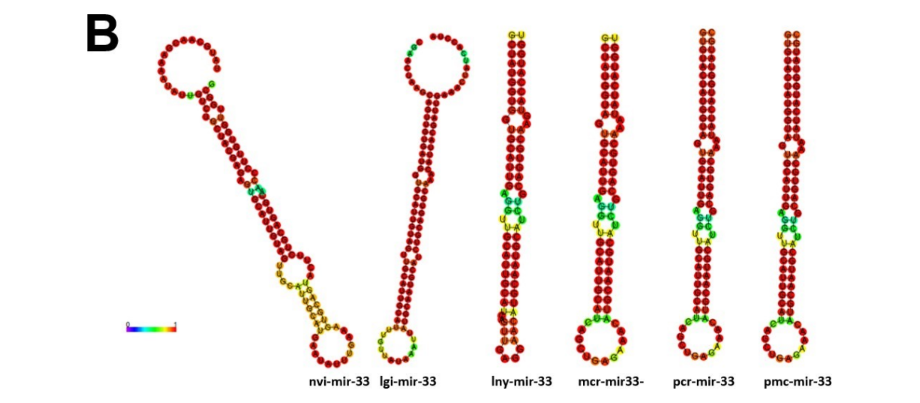
Figura 16: Distribuição filogenética de mir-33 (bilatéria-específico).

4.3.5 mir-153
A família dos genes de mir-153 foi encontrada no genoma de P. maculata. O precursor pmc-mir-153 apresentou o miRNA maduro na extremidade 3’ (Figura 17). Observa-se alta preservação nucleotídica na estrutura secundária e primária de P. maculata e seus ortólogos, principalmente na região do miRNA maduro (Figuras 17 e 18). Foi verificado ampla distribuição entre Deuterostômios e Protostômios, neste clado com presença do filo molusco sendo representado por bivalves e gastrópodes (Figura 20).
Figura 17: Alinhamento de precursores de miRNA-153 com ortólogos, em destaque miRNA maduro.

Figura 18: Estrutura secundária de precursores de miRNA-153 e ortólogos; cgi: Crassostrea gigas, cho: Crassostrea hongkongensis, bgl: Biomphalaria glabrata, rau: Radix auricularia lny: Lanistes nyassanus, pmc: Pomacea maculata.
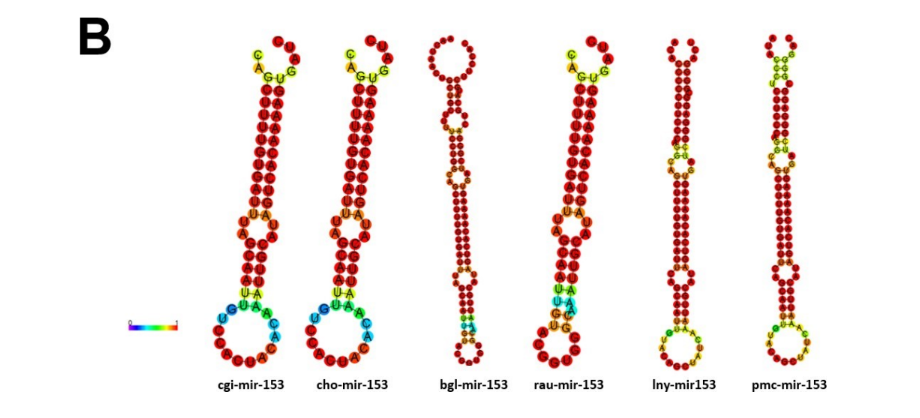
Figura 19: Distribuição filogenética de mir-153 (bilatéria-específico).

4.3.6 mir-190
A família dos precursores de mir-190 foi encontrada nos genomas de P. maculata e P. canaliculata. Observou-se um miRNA maduro por espécie na extremidade 5’ (Figura 20). Mais uma vez, verifica-se à similaridade das estruturas secundárias e primarias dos precursores entre os organismos da família Ampullariidae (Figuras 20 e 21). O mir-190 foi encontrado ao longo da cadeia evolutiva sendo classificado como um miRNA bilatéria específico. Desse modo, representantes dos grupos Lophotrocozoa, Ecdysozoa e Deuterostômios estão presentes na árvore filogenética (Figura 22).
Figura 20: Alinhamento de precursores de miRNA-190 com ortólogos.

Figura 21: Estrutura secundária de precursores de miRNA-190 e ortólogos; hdi: Haliotis discus hannai, afu: Achatina fulica, lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata, pmc: Pomacea maculata, bgl: Biomphalaria glabrata, mle: Melibe leonina
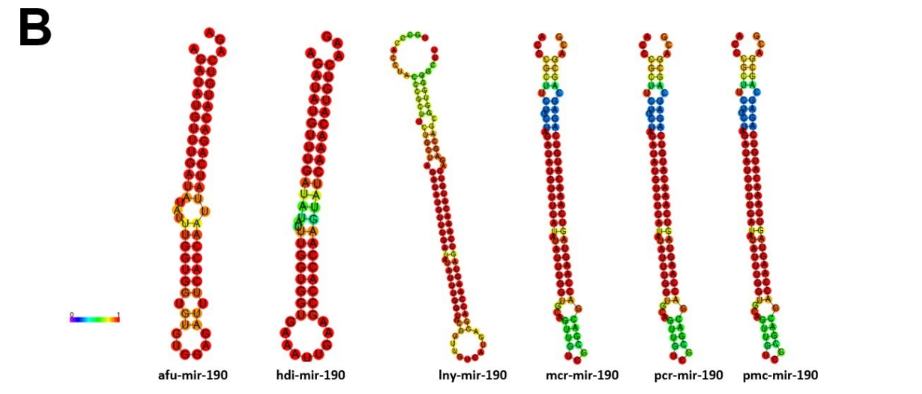
Figura 22: Distribuição filogenética de mir-190 (bilatéria-específico).
4.4 miRNA PROTÔSTOMA ESPECÍFICO
4.4.1 mir-bantam
O mir-bantam está relacionado ao crescimento de células cerebrais e homeostase (LAM et al., 2014; LI; PADGETT, 2012). Em Drosophila, mir-bantam funciona na manutenção do ritmo circadiano, estimulam a proliferação celular e reduzem a morte celular programada (BRENNECKE et al., 2003; KADENER et al., 2009). Na esquistossomose este miRNA de trematódeos pode estar envolvido em processos patológicos da doença, sendo mir-bantam detectado na corrente sanguínea de hospedeiros. Há possibilidade de utilizá-lo como possível biomarcador para diagnóstico desta doença (ZHU; LIU; CHENG, 2014). Em um estudo em B. glabrata foi possível identificar bgl-bantam, que é altamente conservado em metazoários. Foi sugerido que bgl-bantam atue na regulação do metabolismo de caramujos, uma vez que reconhece alvos com função redox e com ação regulatória sobre o complexo ubiquitina-proteína (QUEIROZ et al., 2020). O alinhamento com os precursores ortólogos de P. canaliculata e P. maculata revela alta similaridade com os miRNAs maduros, com 100% de identificação desta espécie com alguns dos seus miRNAs ortólogos (Figura 23). Além disso, há uma similaridade entre as estruturas secundárias dos precursores de miRNAs analisados e seus ortólogos (Figura 24). A árvore filogenética foi representada pelos clados Lophotrocozoa e Ecdysozoa (Figura 25). Estes dados corroboram para identificação de mir-bantam (5p e 3p) nas espécies em estudo.
Figura 23: Alinhamento de precursores miRNA-bantam e ortólogos.

Figura 24: B) Estrutura secundária de precursores de miRNA-bantam e ortólogos; dqu: Dinoponera quadriceps, nvi: Nasonia vitripennis, lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata, pmc: Pomacea maculata.
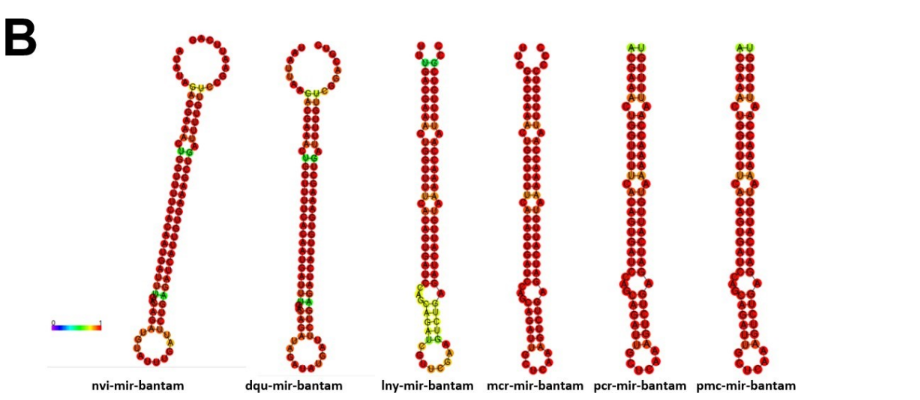
Figura 25: Distribuição filogenética de miR-bantam (protostômio específico).

4.4.2 mir-1175
Há registros de mir-1175 na literatura em mosquitos da ordem insecta como: Aedes aegypti, Anopheles stephensi, Anopheles gambiae, Tinissa amboinensis (LI et al., 2009), conforme observado na figura 28 deste trabalho. Diferentes níveis de expressão de miRNAs são observados em machos e fêmeas da espécie Anopheles anthropophagus. No caso do mir- 1175, ocorre uma diminuição de expressão de cerca de 2 vezes menor em machos quando comparado a fêmeas (LIU et al., 2014). Em moluscos, em um estudo no gastrópode marinho Rapana venosa, analisa os perfis de expressão de miRNAs na metamorfose da espécie. Um total de 195 miRNAs diferentemente expressos foram obtidos neste processo, dentre estes o mir-1175 foi regulado positivamente (SONG et al., 2017). Além disso, a família de mir-1175 foi encontrada no genoma de 34 espécies de moluscos (HUANG et al., 2021), dentre estes cefalópodes, bivalves e gastrópodes (Figura 28). Isso corrobora para a identificação do precursor de mir1175 em P. canaliculata, com a presença de dois miRNAs maduros, 5p e 3p (Figura 26). A estrutura secundária do precursor de P. canaliculata apresenta alta similaridade com os ortólogos, principalmente com representantes da família Ampullariidae, L. nyassanus e M. cornuarietis (Figura 27).
Figura 26: Alinhamento de precursores de miRNA-1175 e ortólogos.
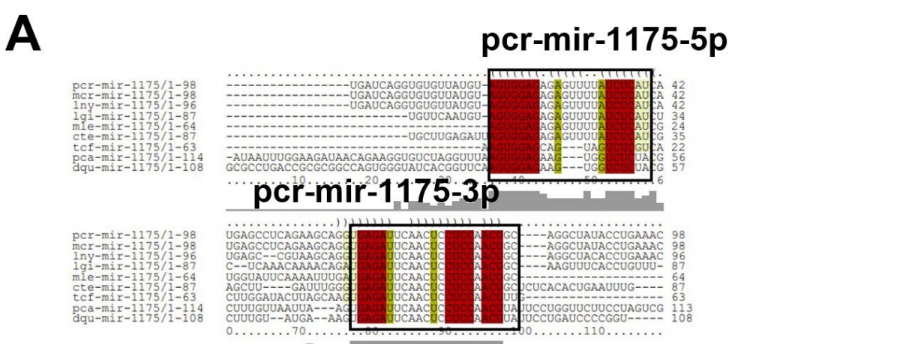
Figura 27: Estrutura secundária de precursores de miRNA-1175 e ortólogos; mle: Melibe leonina, cte: Capitella teleta, lgi: Lottia gigantea, lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata.
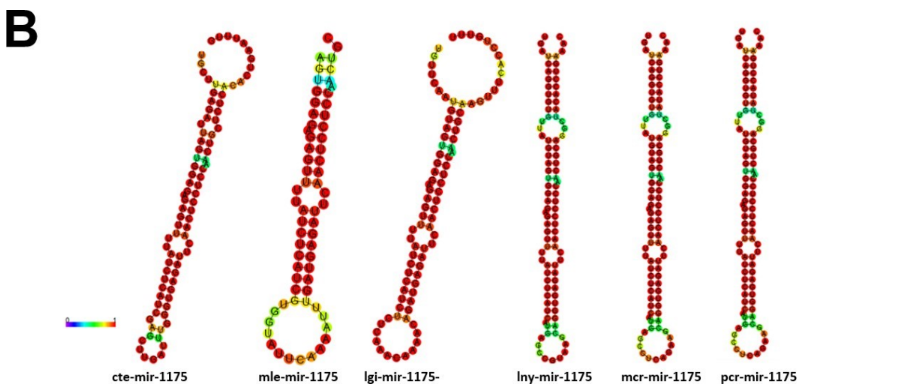
Figura 28: Distribuição filogenética de miR-1175 (protostômio específico).

4.4.3 mir-8
O mir-8 assim como o mir-33 está potencialmente associado à regulação gênica no bivalve T. granosa quando exposto a níveis tóxicos de cádmio (Cd) (BAO et al., 2014). A família de genes de mir-8 foi encontrada no genoma das 2 espécies Ampullariidae analisadas, com a presença das isoformas mir-8-1 e mir-8-2 em P. canaliculata. Para P. maculata foi observado o mir-8 com a presença de um miRNA maduro, 5p (Figura 29). A estrutura secundária do precursor de P. maculata apresentou alta similaridade com o precursor de M. cornuarietis (Figura 30). A árvore filogenética foi representada pelos clados Lophotrocozoa e Ecdysozoa (Figura 31). Estes dados corroboram para identificação de mir-8 (5p) em P. maculata.
Figura 29: Alinhamento de precursores de miRNA-8 e ortólogos.

Figura 30: Estrutura secundária: precursores de miRNA-8 e ortólogos;dgr: Drosophila grimshawi: dan: Drosophila ananassae, der: Drosophila erecta, hdi: Haliotis discus hannai, mcr: Marisa cornuarietis, pmc: Pomacea maculata.
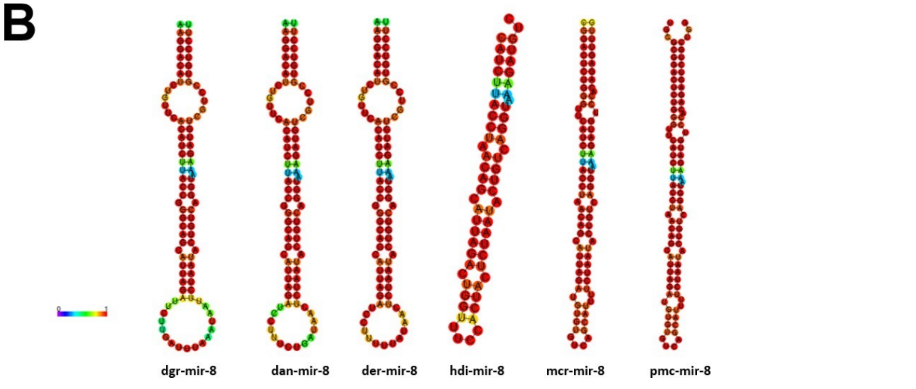
Figura 31: Análise filogenética de miR-8 (protostômio específico).

4.4.4 mir-12
Assim como o mir-8, o mir-12 está relacionado potencialmente ao desenvolvimento do crustáceo Daphnia pulex. A regulação positiva de mir-8 coincide com mudanças morfológicas e crescimento na espécie (CHEN et al., 2014b). Já em moluscos, a família dos precursores de mir-12 foi identificada no organismo B. glabrata (QUEIROZ et al., 2020), próximo evolutivamente aos organismos do gênero Pomacea analisados. Os precursores de B. glabrata, P. canaliculata e P. maculata (Figura 32) apresentaram o mir-12-5p para cada espécie. A estrutura secundária dos pré-miRNAs de P. canaliculata e P. maculata apresentaram formato canônico característico dos precursores, intrinsecamente semelhantes entre si (Figura 33). A distribuição filogenética foi verificada entre os clados Ecdysozoa e Lophotrocozoa, sendo este representado por moluscos das classes cefalópode, bivalve e gastrópode (Figura 34).
Figura 32: Alinhamento de precursores de miRNA-12 e ortólogos.
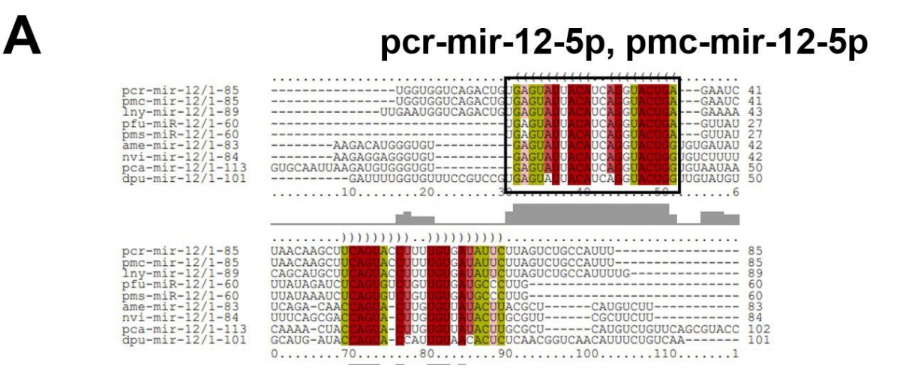
Figura 33: Estrutura secundária: precursores de miRNA-12 e ortólogos: ame: Apis melífera, pca: Polistes canadensis pfu: Pinctada fucata, lny: Lanistes nyassanus, pcr: Pomacea canaliculata, pmc: Pomacea maculata.
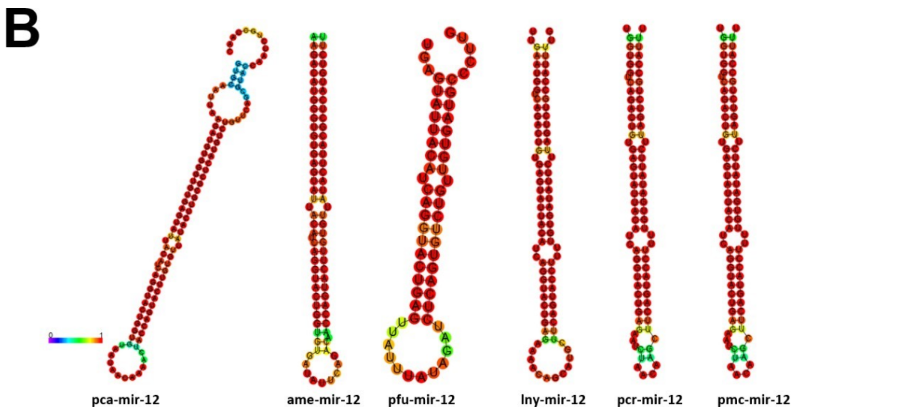
Figura 34: Análise filogenética de miR-12 (protostômio específico).
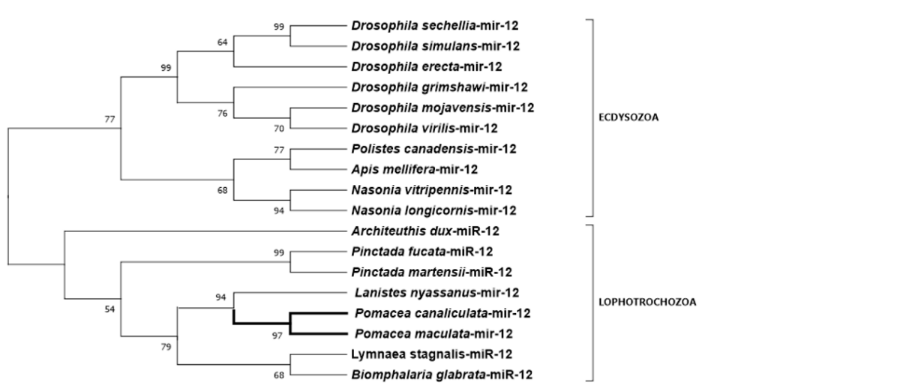
4.4.5 mir-750
A família do mir-750 apresentou o precursor mir-750 nos organismos P. canaliculata e P. maculata e os miRNAs maduros pcr-mir-750-3p e pmc-miR-750-3p (Figura 35). O alinhamento com ortólogos e estruturas secundárias revelaram alta preservação nucleotídica entre espécies do gênero Pomacea e ortólogos (Figuras 35 e 36). Na distribuição filogenética foi observado o clado Ecdysozoa, representado por artrópodes e nematódeo e o clado Lophotrocozoa, constituído exclusivamente por moluscos (Figura 37).
Figura 35: Alinhamento de precursores de miRNA-750 e ortólogos.

Figura 36: Estrutura secundária: precursores de miRNA-bantam e ortólogos; dqu: Dinoponera quadriceps, pca: Polistes canadensis, lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata, pmc: Pomacea maculata.
Figura 37: Análise filogenética de miR-750 (protostômio específico).

4.5 miRNA LOPHOTROCOZOA ESPECÍFICO
4.5.1 mir-745a
A família de mir-745a apresentou um miRNA maduro nas sequencias de cada precursor das espécies P. canaliculata e P. maculata, extremidade 3’ (Figura 38). Frente aos seus ortólogos houve conservação da sequência de nucleotídeos bem como da estrutura secundária (Figuras 38 e 39). Há registros da família mir-745a em 28 espécies de moluscos, dentre estes organismos próximos evolutivamente as espécies analisadas; B. glabrata, Aplysia californica e Lottia gigantea (HUANG et al., 2021). Foi verificado uma distribuição filogenética formada por animais dos filos dos moluscos, platelmintos e anelídeos (Figura 40).
Figura 38: Alinhamento de precursores de miRNA-745a com seus ortólogos.

Figura 39: Estrutura secundária dos precursores de miRNA-745a e ortólogos; lgi: Lottia gigantea, bgl: Biomphalaria glabrata, cte: Capitella teleta, lny: Lanistes nyassanus, pcr: Pomacea canaliculata, pmc: Pomacea maculata.
Figura 40: Distribuição filogenética de miR-754a (lophotrochozoa-específico).
4.5.2 mir-1992
A família dos genes de mir-1992 foi identificada apenas no genoma da espécie de P. canaliculata, com a presença de dois miRNAs maduros, 3p e 5p (Figura 41). A estrutura secundária do precursor de P. canaliculata revelou alta similaridade com seus ortólogos (Figura 42). O mir-1992 revelou expressão diferencial nos testículos e ovários de Crassostrea hongkongensis, sendo altamente expressos (WEI et al., 2019). Em gastrópodes, o mir-1992 foi identificado em 9 espécies de moluscos (HUANG et al., 2021). Assim, na distribuição filogenética foi representado em sua maior parte por moluscos gastrópodes e também por cefalópodes, além da presença de platelmintos (Figura 42).
Figura 41: Alinhamento de precursores miRNA-1992 com seus ortólogos, em destaque miRNAs maduros
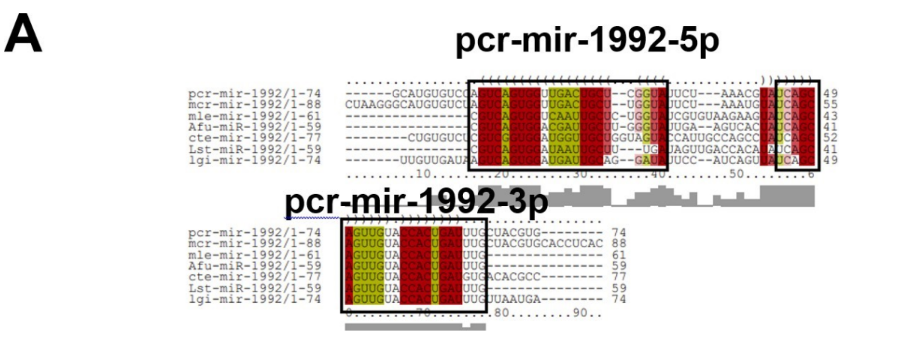
Figura 42: Estrutura secundária dos precursores de miRNA-1992 e ortólogos; lgi: Lottia gigantea, mle: Melibe leonina, cte: Capitella teleta, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata.
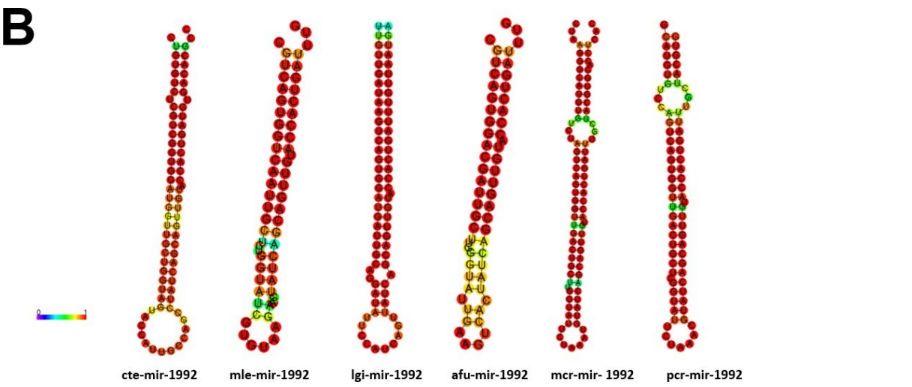
Figura 43: Distribuição filogenética de miR-1992 (lophotrochozoa-específico). Em destaque a espécie: Pomacea canaliculata.
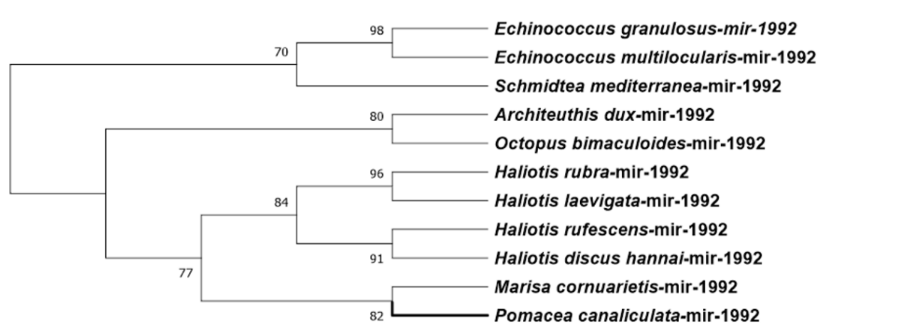
4.5.3 mir-1994
A família do mir-1994 é composta pelos precursores mir-1994 e mir-1994a, para ambas as espécies da família Ampullariidae. P. canaliculata e P. maculata, apresentam um miRNA maduro (3p) para cada precursor mir-1994 (Figura 44). Comparado aos seus ortólogos as espécies analisadas apresentaram conservação da sequência de nucleotídeos bem como da estrutura secundária (Figuras 44 e 45). A distribuição filogenética observou-se a presença de animais dos filos como Nemertea, Mollusca e Annelida (Figura 46).
Figura 44: Alinhamento de precursores de miRNA-1994 com ortólogos.
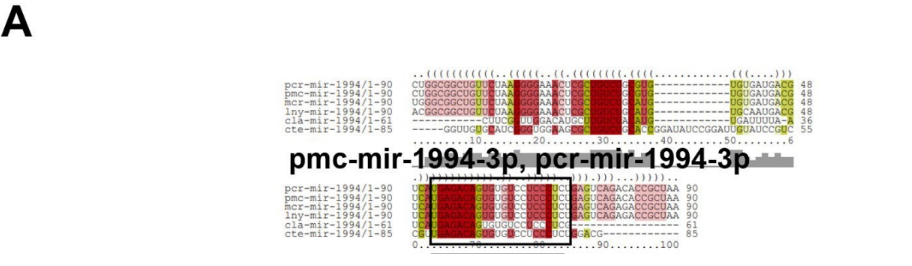
Figura 45: Estrutura secundária dos precursores de miRNA1994 com ortólogos; cte: Capitella teleta, cla: Cerebratulus lacteus, lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata, pmc: Pomacea maculata.
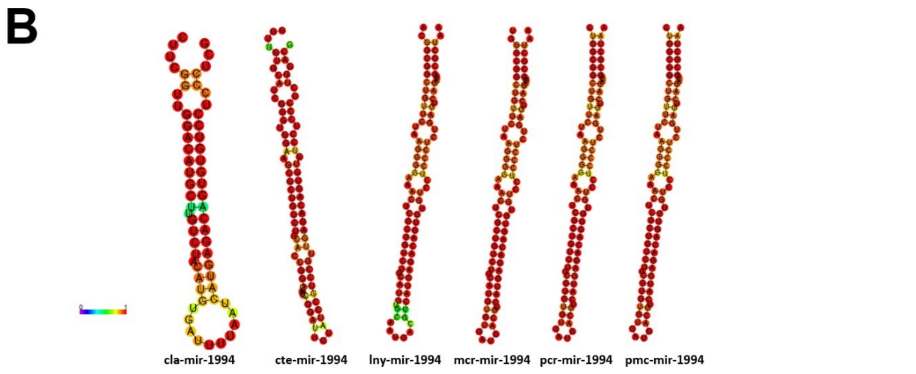
Figura 46: Distribuição filogenética de miR-1994 (lophotrochozoa-específico).
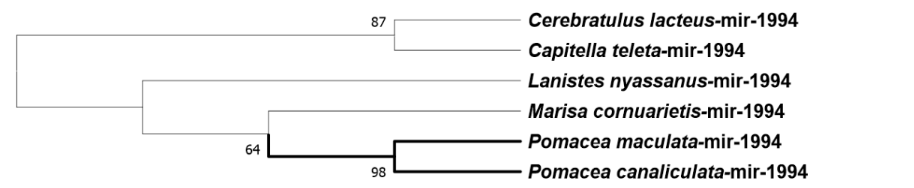
4.5.4 mir-96b
As sequências dos precursores do mir-96b apresentaram dois miRNAs maduros, 3p e 5p, nos genomas das espécies P. canaliculata e P. maculata (Figura 47). Os precursores de miR-96b para ambas as espécies demonstraram alta similaridade na estrutura secundária com os organismos gastrópodes (Figuras 48). A análise filogenética dos precursores do mir-96b foi verificada a distribuição entre as espécies em três filos como Moluscos, Anelídeos e Platelmintos (Figura 49). O mir-96b regula genes de forma pós-transcricional em Platelmintos e moluscos. No verme de água doce: Schmidtea mediterrânea pode ter funções na cicatrização de feridas, proliferação de neoblatos (principais responsáveis pela regeneração em planárias) e diferenciação de blastema (SASIDHARAN et al., 2013). Em moluscos, o mir-96b está envolvido junto com outros miRNAs na pigmentação das conchas de C. gigas, induzindo a síntese de melanina, carotenóide ou tetrapirrol (FENG et al., 2020). O mir-96b também foi encontrado no genoma de gastrópodes próximos evolutivamente às 2 espécies Ampullariidae, B. glabrata (QUEIROZ et al., 2020) e A. californica (HUANG et al., 2021). Achados na literatura científica corroboram os dados deste trabalho.
Figura 47: Alinhamento de precursores de miRNA-96b com seus ortólogos.

Figura 48: Estrutura secundária de precursores de miRNA-96b com seus ortólogos; mle: Melibe leonina, lgi: Lottia gigantea, lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata, pmc: Pomacea maculata.
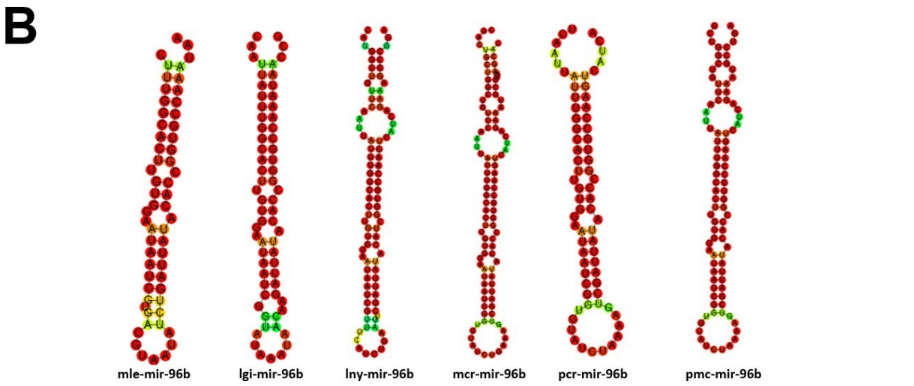
Figura 49: Análise filogenética de miR-96b (lophotrochozoa-específco).

4.5.5 mir-1990
A família mir-1990 predita para as espécies P. canaliculata e P. maculata é composta pelos precursores pcr-mir-1990 e pcm-mir-1990 e os miRNAs maduros, 3p e 5p, em cada espécie (Figura 50). Para esta família, observou-se grande conservação da estrutura primária, bem como da estrutura secundária dos precursores de P. canaliculata, P. maculata e seus ortólogos (Figuras 50 e 51). Verificou-se ampla distribuição de mir-1990 em moluscos principalmente em 15 animais bivalves, 10 gastrópodes e 1 cefalópode. Poucos estudos descreveram o papel dos miRNAs no desenvolvimento de moluscos. O mir-1990-3p pode desempenhar um papel vital no processo de biomineralização da concha em P. fucata, visto que é altamente expresso nos tecidos do manto (HUANG et al., 2021). O mir-1990 foi encontrado apenas em moluscos, mas a família mir-1990 é Lophotrochozoa específico pois cte-miR-1990c- 3p, cte-miR-1990a e cte-miR-1990b foram identificados em Capitella teleta na plataforma miRBase (http://www.mirbase.org/). A ampla distribuição do mir-1990 em moluscos e o provável mecanismo de controle gênico corroboram os dados deste estudo.
Figura 50: Alinhamento de precursores de miRNA-1990 com seus ortólogos.
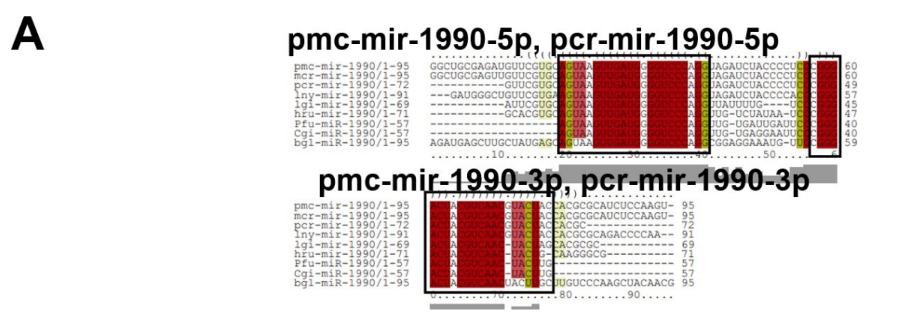
Figura 51: Estrutura secundária dos precursores miRNA-1990 ortólogos; cgi Crassostrea gigas, bgl: Biomphalaria glabrata, lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata, pmc: Pomacea maculata.
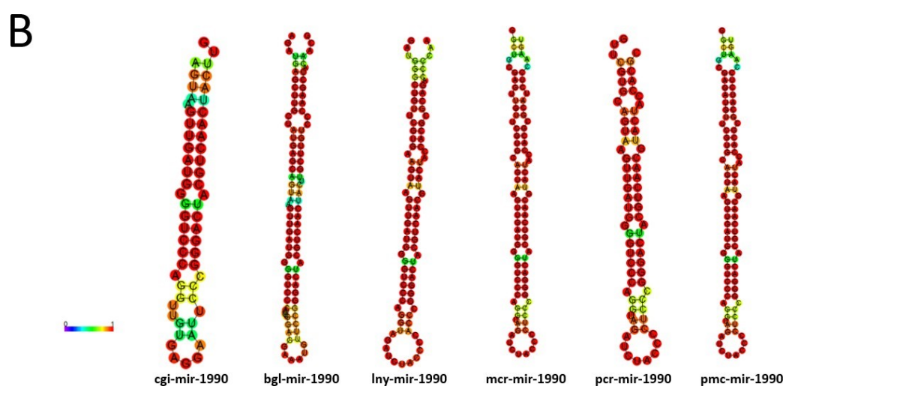
4.6 miRNA MOLLUSCA ESPECÍFICO
4.6.1 mir-1984
Em C. gigas, a expressão de mir-1984 aumentou significativamente em resposta aos estresses: baixa salinidade, calor e infecções bacterianas, sugerindo que este miRNA pode estar implicado em certas funções fisiológicas, como redução da oxidação e metabolismo energético (ZHAO et al., 2016). Já em gastrópodes este miRNA pode estar envolvido no crescimento muscular. Foi verificado que o gene BMP7 é alvo do hdl-miRNA-1984 no molusco gastrópode Haliotis discus hannai. A proteína morfogenética óssea 7 (BMP7) induz a formação da cadeia leve quinase de miosina e cadeia pesada de miosina. Desta forma, o mir-1984 pode nortear o crescimento muscular ao regular o gene BMP7 no abalone (HUANG et al., 2018). Achados na literatura científica corroboram os dados deste trabalho, na qual as sequencias dos precursores de P. canaliculata e P. maculata apresentaram conservação na estrutura primária e secundária em relação aos ortólogos (Figuras 52 e 53).
Figura 52: Alinhamento de precursores de miRNA-1984 com seus ortólogos.
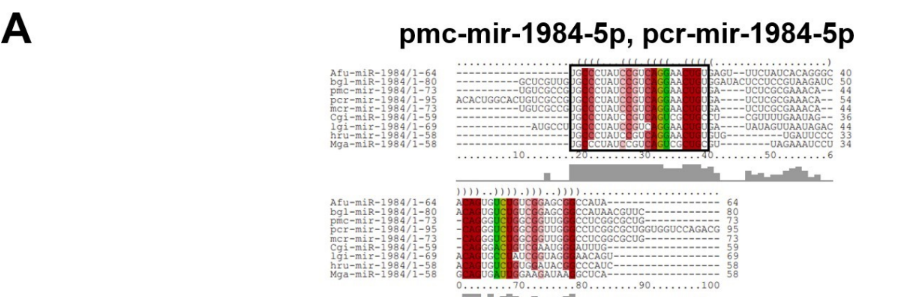
Figura 53: Estrutura secundária dos precursores de miRNA-1984 com seus ortólogos; cgi: Crassostrea gigas, afu: Achatina fulica, bgl: Biomphalaria glabrata, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata, pmc: Pomacea maculata.
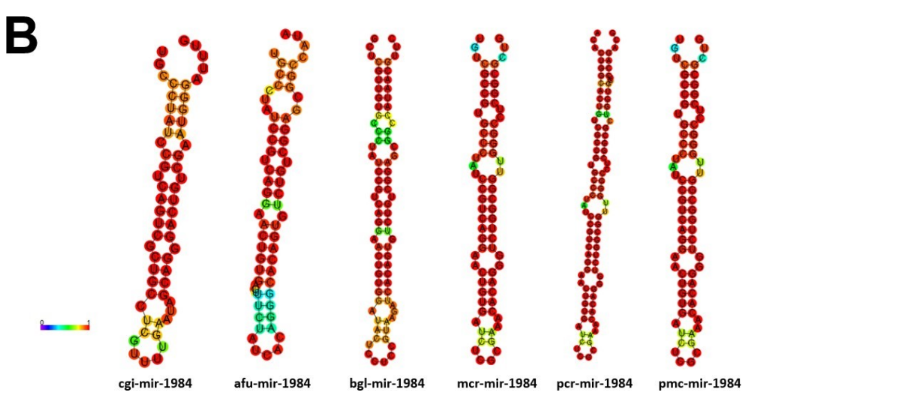
4.6.2 mir-1985
A família do mir-1985 predita para as espécies P. canaliculata e P. maculata é composta pelos precursores pcr-mir-1985 e pmc-mir-1985 e pelos miRNAs maduros pcr-miR-1985-5p e pmc-miR-1985-5p. Estes miRNAs demonstraram 100% de identidade com seus ortólogos (Figura 54). A estrutura secundária também se apresentou conservada entre os moluscos do gênero Pomacea e ortólogos (Figura 55).
Figura 54: Alinhamento de precursores de miRNA-1985 com seus ortólogos.

Figura 55: Estrutura secundária dos precursores miRNA-1985 e ortólogos; pfu: Pinctada fucata, bgl: Biomphalaria glabrata, mcr: Marisa cornuarietis, pcr: Pomacea canaliculata, pmc: Pomacea maculata.
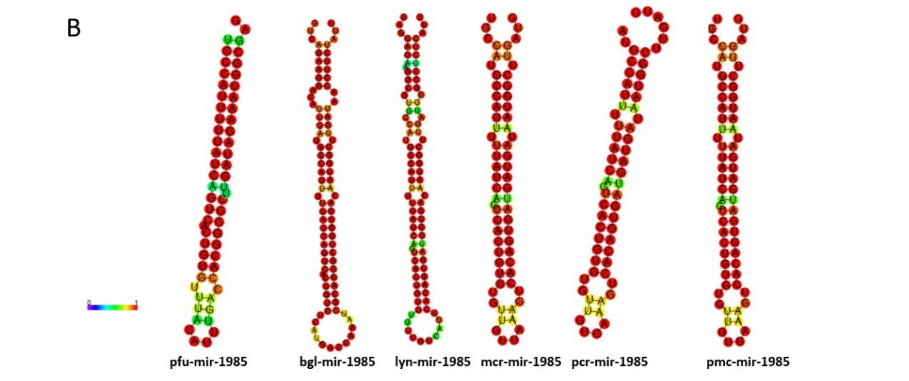
4.6.3 mir-12096b
A família de mir-12096b foi encontrado no genoma de P. maculata. O precursor predito pmc-mir-12096b apresentou dois miRNAs maduros, 3p e 5p (Figura 56). Estes miRNAs foram localizados no genoma de Melibe Leonina na plataforma miRBase. Além disso, a família mir- 12096b foi encontrada no genoma das espécies de moluscos; A. californica, Chlamys farreri, Haliotis discus hannai, Haliotis laevigata e Haliotis rubra (HUANG et al., 2021). No alinhamento das estruturas primárias de P. maculata e ortólogos foi verificado alta conservação de nucleotídeos, sobretudo nas regiões dos miRNAs maduros (Figura 56). A estrutura secundária de pmc-mir 12096b demonstrou alta similaridade estrutural com ortólogos, especialmente entre organismos da família Ampullariidae (Figura 57).
Figura 56: Alinhamento de precursores de miRNA-12096b com seus ortólogos.

Figura 57: Estrutura secundária dos precursores miRNA-12096b e ortólogos; hla: Haliotis laevigata, hdi: Haliotis discus hannai, mle: Melibe leonina, lny: Lanistes nyassanus, mcr: Marisa cornuarietis, pmc: Pomacea maculata.
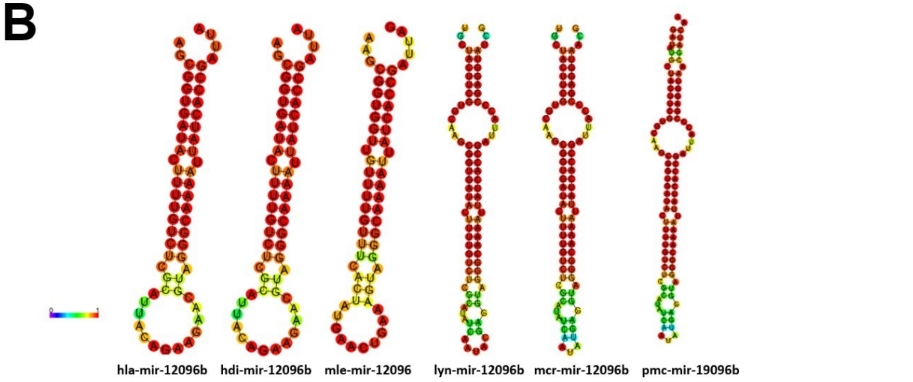
Os prováveis miRNAs maduros de P. canaliculata e P. maculata, bilatéria-específico, protostômio-específico, lophotrochozoa-específico e mollusca específico, caracterizados neste trabalho apresentaram uma grande conservação na região dos miRNAs maduros, quando comparados com seus respectivos ortólogos. Esta conservação se acentua nas regiões “seeds”, demonstrando alta preservação nucleotídica, com 100% de similaridade nas posições de 2 a 8 nucleotídeos. As estruturas secundárias dos pré-miRNAs apresentaram formato característico canônico dos precursores de miRNAs e revelaram alta similaridade estrutural entre os ortólogos. Na análise filogenética, foi verificada ampla distribuição entre os clados filogenéticos, verificando uma distribuição evolutiva que corrobora com a árvore da vida.
4.7 IDENTIFICAÇÃO E CARACTERIZAÇÃO DE PROTEÍNAS ENVOLVIDAS NA BIOGÊNESE DA VIA DE miRNAS EM P. canaliculata E P. maculata.
Os miRNAs são importantes reguladores da expressão gênica que necessitam ser processados no núcleo e citoplasma em pri-miRNA, pré-miRNA e, finalmente, na forma madura do miRNA. Para isso, várias proteínas estruturais e catalíticas executam funções pré- determinadas de forma a obter a forma ativa do regular gênico.
Neste trabalho, as proteínas responsáveis pela biogênese dos miRNAs das espécies P. canaliculata e P. maculata foram identificadas e recuperadas a partir do proteoma predito das espécies localizados no banco de dados NCBI (http://.ncbi.nlm.nih.gov/). A partir desses dados, através de um script próprio, foi utilizado como ferramenta o BLASTp, usando como sequências de busca (queries) as proteínas da via de miRNAs oriundas dos organismos modelo D. melanogaster e C. elegans.
Foram identificadas e caracterizadas as prováveis proteínas em P. canaliculata e P. maculata envolvidas na via de miRNA. A detecção foi realizada baseada na homologia, comprimento da proteína, análise de domínios conservados, pesquisa de sítios ativos e filogenia (CARDOSO et al., 2020; QUEIROZ et al., 2017). As proteínas preditas foram comparadas com suas ortólogas de organismos próximo evolutivamente como A. californica e B. glabrata (Tabelas 9 e 10).
Tabela 9: Similaridade das proteínas da via de miRNA de P. canaliculata e suas ortólogas.
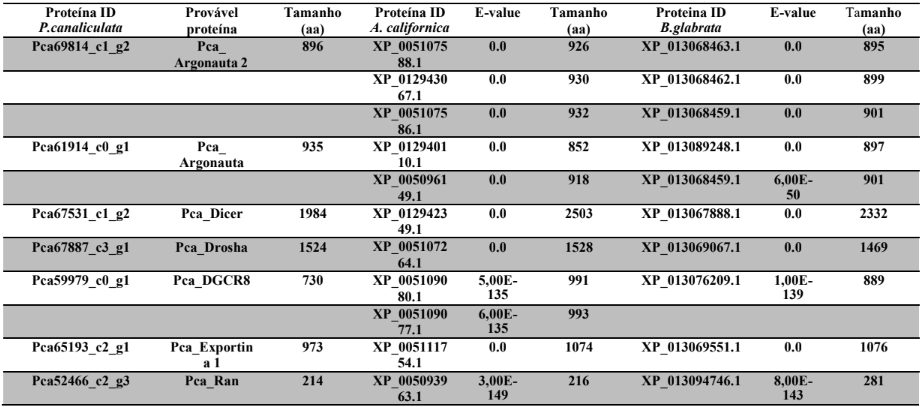
Tabela 10: Similaridade das proteínas da via de miRNA de P. maculata e suas ortólogas.
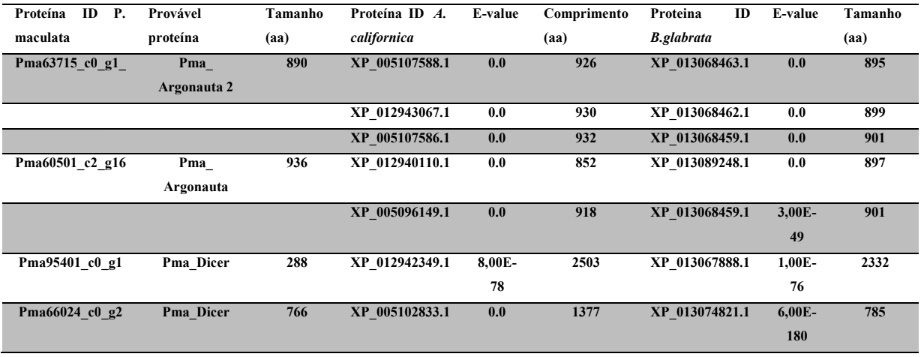
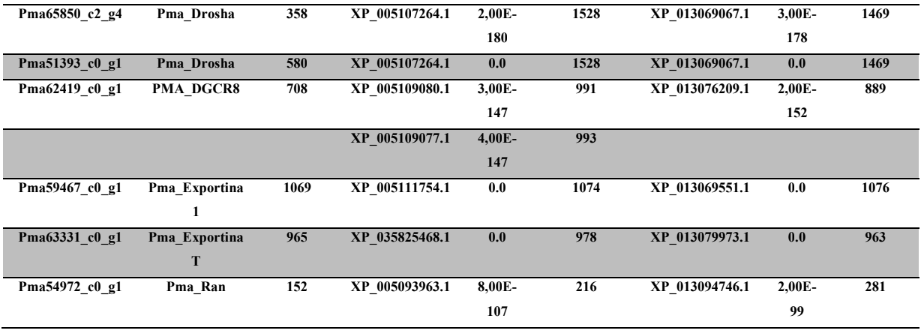
Os domínios conservados das proteínas foram analisados de forma a caracterizar as proteínas preditas. A partir das sequências de aminoácidos de P. canaliculata e P. maculata obtidas, foi utilizada a ferramenta Blastp no banco de dados RefSeq do NCBI (http://.ncbi.nlm.nih.gov/), para busca por proteínas ortólogas. Desta forma, os melhores hits foram aquelas que tinham maior similaridade e, então, recuperadas para pesquisa de domínios conservados, alinhamento e filogenia. As sequências escolhidas para pesquisa de sítios ativos basearam-se em espécies próximas evolutivamente, de organismos modelos (D. melanogaster, C. elegans, H. sapiens), além de protostômios e deuterostomios de forma geral. Tendo em vista a relação parasita hospedeiro entre P. canaliculata e A. cantonensis, procurou-se no banco de dados públicos proteínas do nematoide ortológas ao molusco, não obtendo sucesso na pesquisa.
4.7.1 Análise de domínios conservados, alinhamento global e filogenia das proteínas Argonauta
A proteína Argonauta é caracterizada pela presença dos domínios PIWI e PAZ (PIWI- Argonauta-Zwille) (SONG et al., 2004). As proteínas Argonauta de eucariotos podem apresentar além destes os seguintes domínios, N (N-terminal), e MID, junto com dois ligantes de domínio L1 e L2 (Figura 58) (SWARTS et al., 2014). O domínio PIWI em Argonauta contém três resíduos catalíticos conservados compostos por dois aspartatos e uma histidina, chamado de ‘DDH’ (YANG; STEITZ, 1995).
Figura 58: Arquiteturas de domínio das proteínas de eucariotos da superfamília PIWI.

Foram identificadas quatro prováveis proteínas Argonauta nas espécies em estudo, sendo duas em P. canaliculata (Pca69814_c1_g2, Pca61914_c0_g1) e duas em P. maculata (Pma63715_c0_g1_, Pma60501_c2_g16). A sequência proteica Argonauta 2 de P. canaliculata, (Pca69814_c1_g2) apresentou os mesmos e-values e ortólogos descritos para P. maculata, (Pma63715_c0_g1) para as espécies citadas na tabela 11.
Tabela 11: Similaridade de Argonauta 2 de P. maculata e ortólogos (protostômios e deuterostômios).
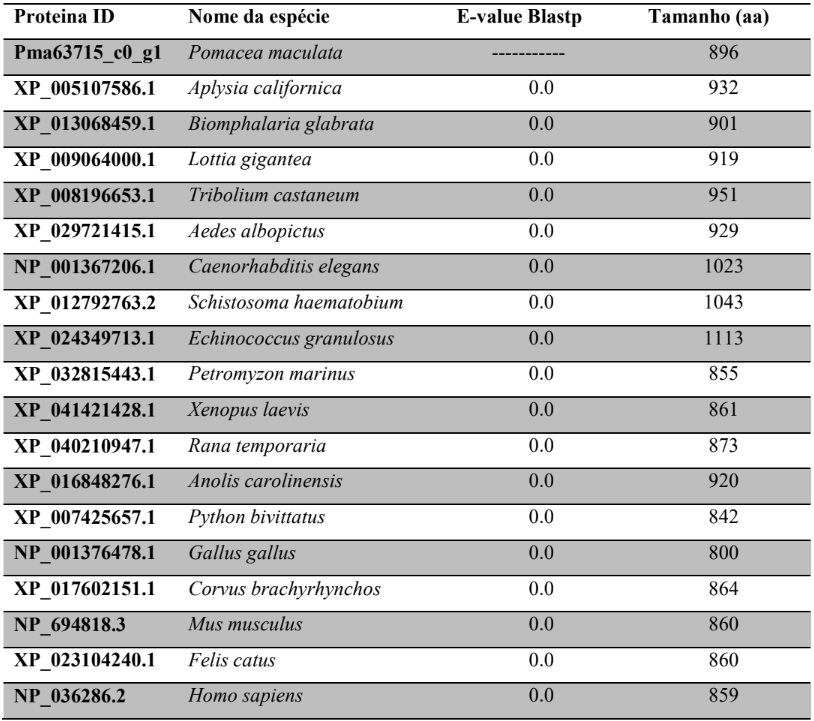
As sequências de aminoácidos Pca69814_c1_g2 e Pma63715_c0_g1 apresentaram seis domínios conservados (Tabela 12), enquanto as proteínas Argonauta (Pca61914_c0_g1 e Pma60501_c2_g16) apresentaram apenas os domínios PAZ E PWI (Tabela 13 e Figura 59). O comprimento das sequências para ambas as espécies, além da similaridade com suas ortólogas corroboram para identificação das prováveis proteínas Argonauta 2 e Argonauta (Tabelas 11,12,13 e 14, Figura 60).
Tabela 12: Análise de domínios conservados Argonauta 2, P. maculata, P. canaliculata e ortólogos.
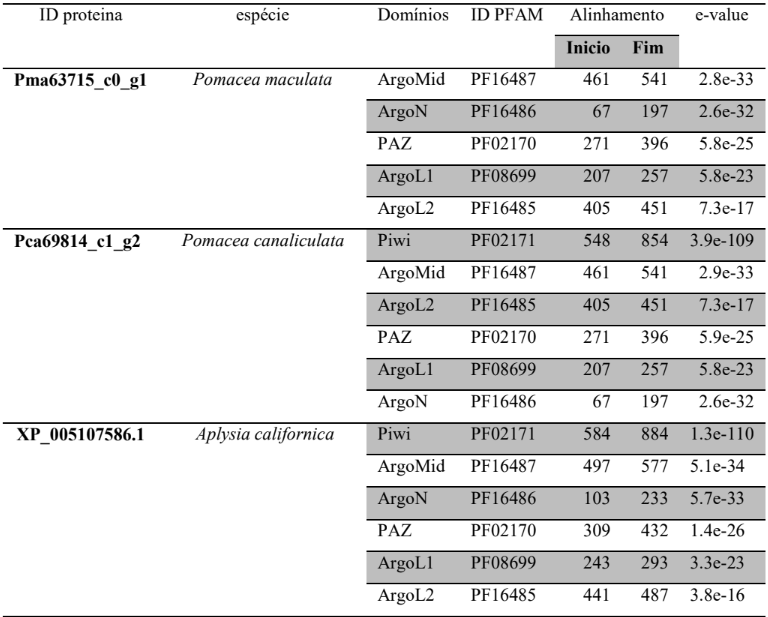
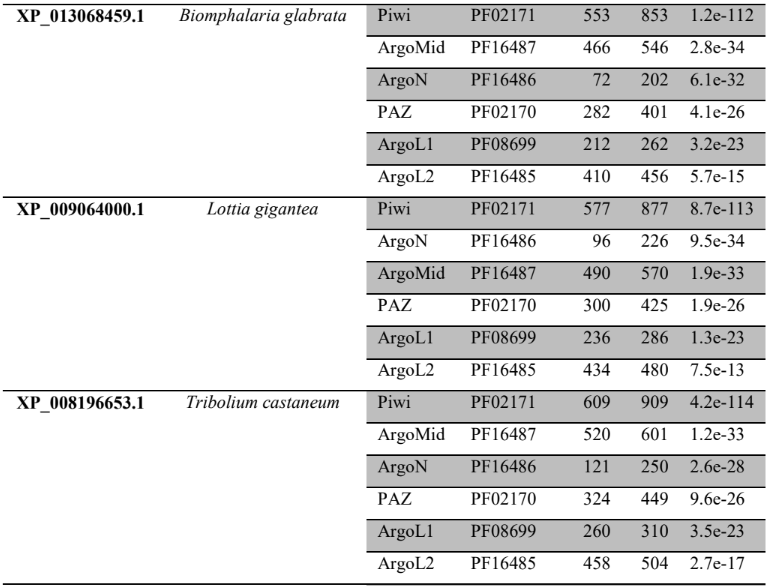

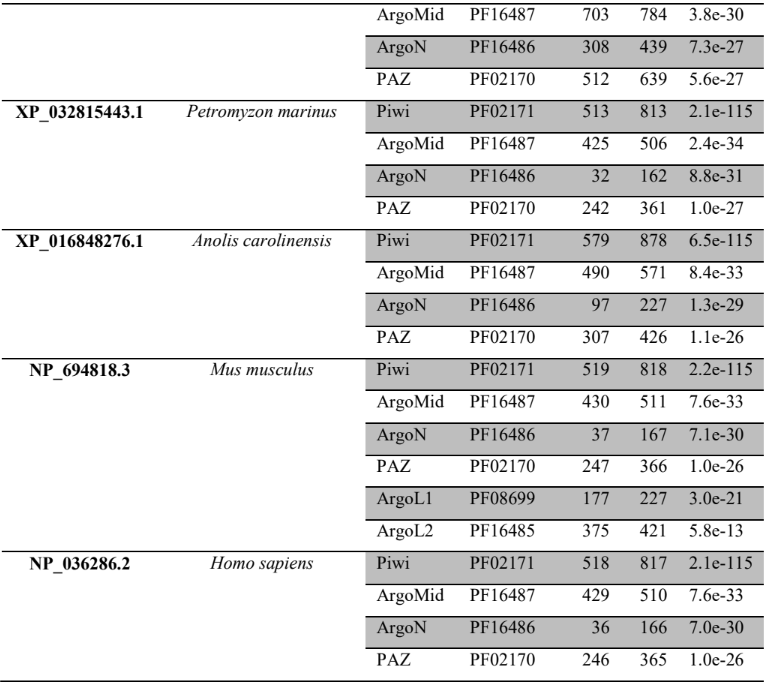
Tabela 13: Similaridade de Argonauta de P. maculata e ortólogos (protostômios e deuterostômios).
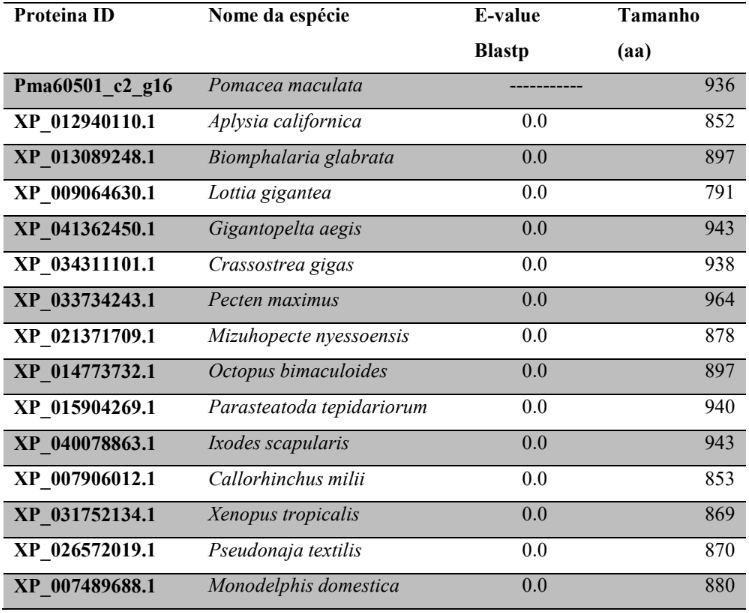

Tabela 14: Análise de domínios conservados Argonauta P. maculata, P. canaliculata e ortólogos.
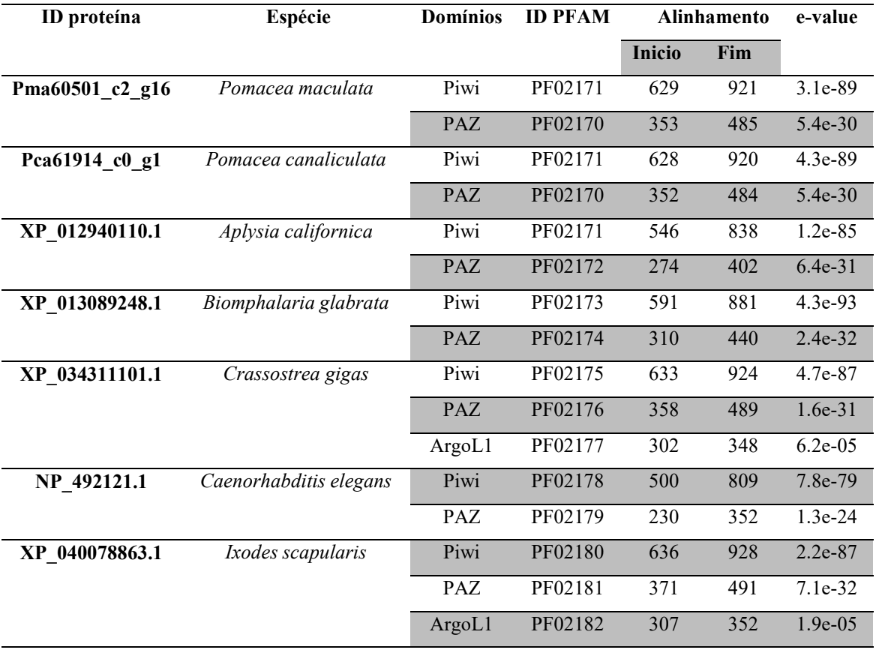
Figura 59: Distribuição dos domínios conservados, prováveis proteínas Argonauta.

Para P. canaliculata e P. maculata, foi realizado o alinhamento global de forma a demonstrar a similaridade de sequências peptídicas de organismos diversos das prováveis proteínas da via de miRNAs, além de mostrar os resíduos e as circunvizinhanças dos sítios catalíticos. O alinhamento global foi realizado utilizando o software ClustaX2.1 (Figura 60)
O domínio PIWI das prováveis proteínas de P. canaliculata e P. maculata apresentaram uma tríade catalítica formada por DDH (ácido aspártico, ácido aspártico e histidina), apresentando alta conservação em relação às proteínas ortólogas de Argonauta, principalmente nas imediações do resíduo catalítico (Figura 60).
Figura 60: Alinhamento global, análise do domínio PIWI das proteínas Argonauta de P. canaliculata e P. maculata.
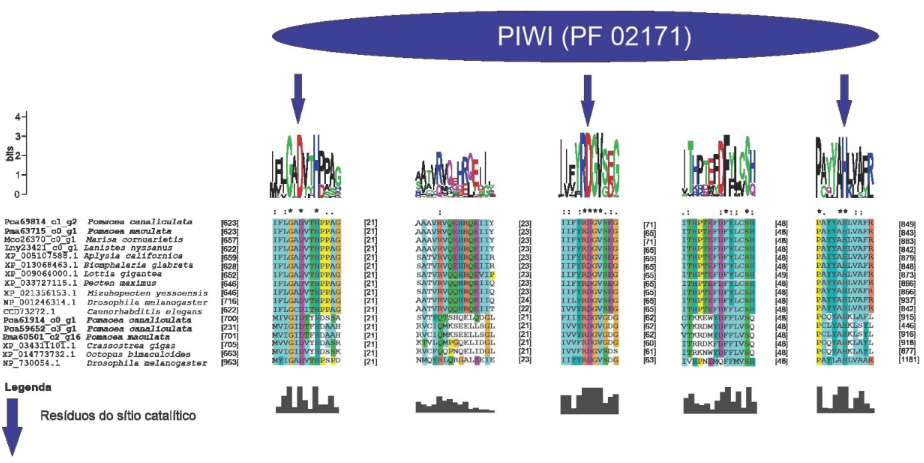
As sequências das proteínas preditas de P. canaliculata, P. maculata e ortólogos de referência (RefSeq) (organismos protostômios e deuterostômios) foram submetidas a análises filogenéticas comparativas utilizando o programa MEGA versão X (TAMURA et al., 2007). As árvores filogenéticas foram construídas utilizando o método Neighbor-joining (NJ) (SAITOU; NEI, 1987) e calculada com o modelo de substituição JTT. Na análise filogenética foi verificada a ampla distribuição entre os clados filogenéticos, verificando uma distribuição evolutiva que corrobora com a árvore da vida (Figura 61).
Figura 61: Análise filogenética das proteínas Argonauta, de P. maculata e P. canaliculata e seus ortólogos.

4.7.2 Análise de domínios conservados, alinhamento global e filogenia das proteínas Dicer e Drosha
A ribonuclease III (RNase III) pertence à família das endoribonucleases que apresentam especificidade para RNA de fita dupla (dsRNA). Três classes de enzimas RNase III foram relatadas. As enzimas da classe I são as mais simples, contendo um domínio de endonuclease e um domínio de ligação de dsRNA (dsRBD). As proteínas de classe II contêm dois domínios de endonuclease e um dsRBD. As moléculas de classe III consistem em dois domínios de endonuclease, um dsRBD e um domínio de helicase N-terminal seguido por um domínio PAZ (Figura 62). Funcionalmente, as enzimas de classe 3 produzem produtos de RNA de fita simples (ssRNA) de aproximadamente 22 nucleotídeos de comprimento a partir de substratos de dsRNA longos (BERNSTEIN et al., 2001; BLASZCZYK et al., 2001; FILIPPOV et al., 2000). A via canônica da biogênese de miRNAs é a via dominante no qual os miRNAs são processados pelas RNAases III de classe II (Drosha) e classe III (Dicer), consecutivamente.
Figura 62: Três classes de proteínas da família RNase III representadas por Dicer humano (classe III), Drosha humana (classe II) e RNase III bacteriana (classe I).
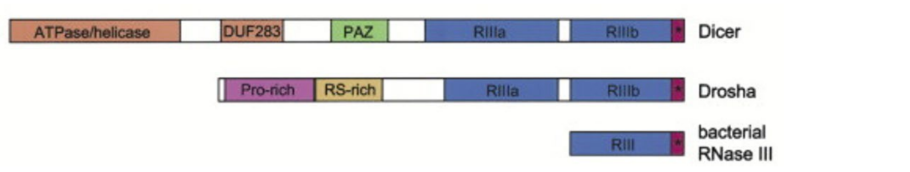
As RNases III (classes II e II) contém centros catalíticos com aminoácidos importantes para a função. A atividade das enzimas se concentra em domínios RIBOc, que possuem sítios ativos compostos pelos seguintes aminoácidos E (Ácido glutâmico), D (Ácido aspártico), D (Ácido aspártico), E (Ácido glutâmico) (Figura 64) (HAN et al., 2004; ZHANG et al., 2004)
Tabela 15: Similaridade de Drosha de P. canaliculata e ortólogos.
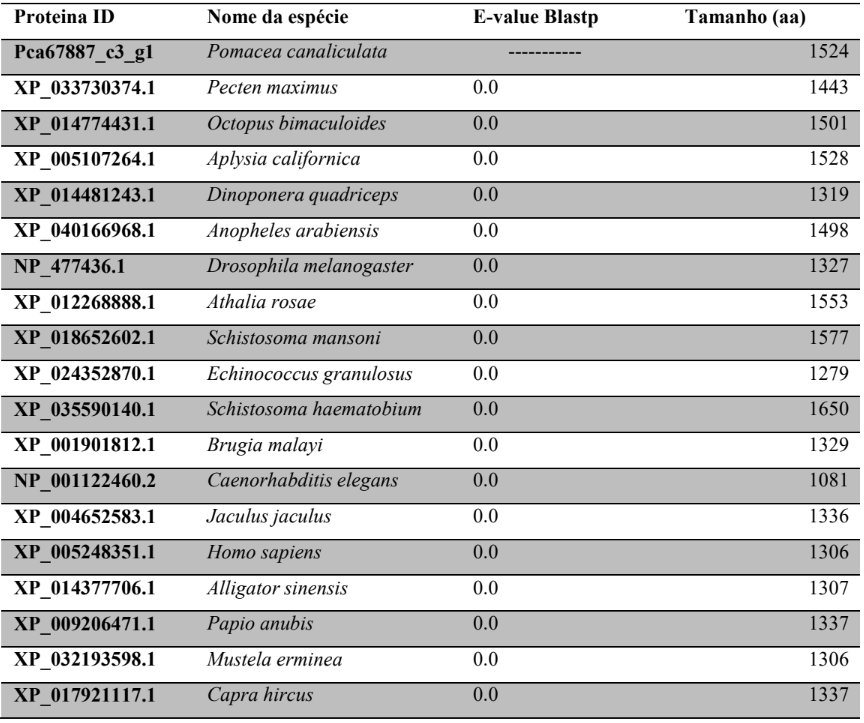
Tabela 16: Análise de domínios conservados de Drosha P. canaliculata e ortólogos.
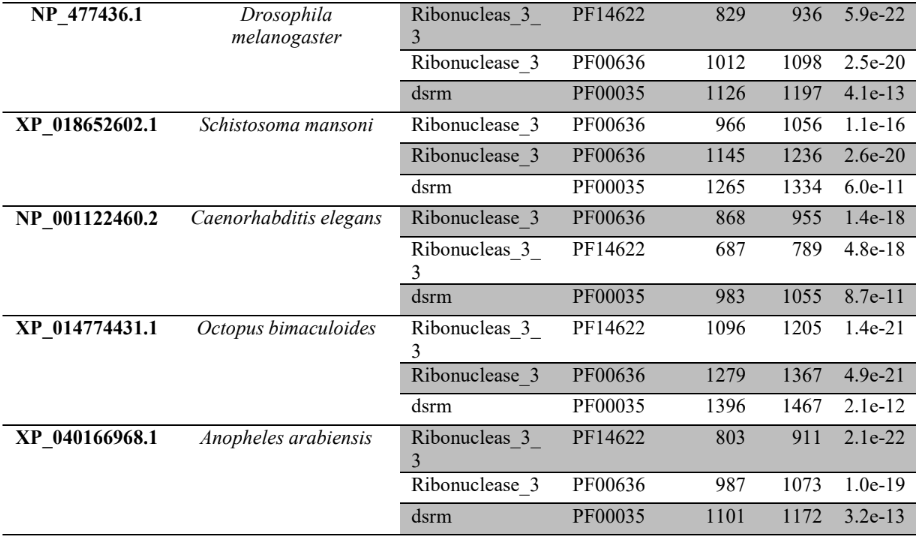


Figura 63 : Análise de domínios conservados de Drosha em P. canaliculata.
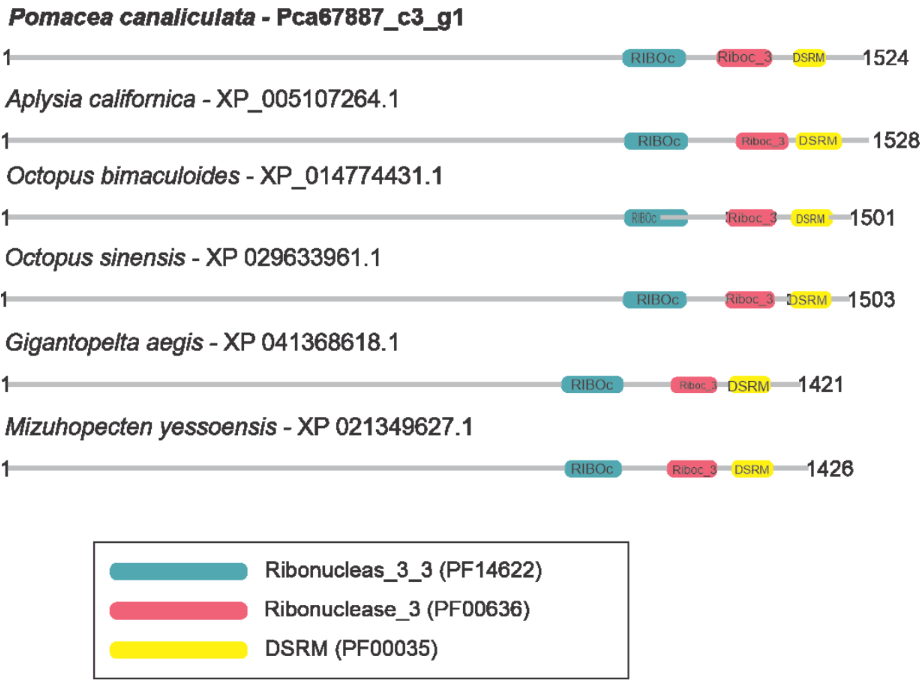
A provável proteína Pca67887_c3_g1 de P. canaliculata (Pca_drosha) apresentou comprimento compatível e similaridade com seus ortólogos, principalmente com relação aos clados Mollusca e Arthropoda (Tabela 15 e Figura 63). Na análise de domínios conservados, apresentou os dois domínios de endonuclease e um dsrm (importante para a ligação à dupla fita de RNA), característico das RNAases III de classe 2 (Tabela 16 e Figura 63). Na pesquisa por sítios catalíticos, apresentou apenas o primeiro conjunto composto pelos aminoácidos E-D-D- E no domínio Riboc (figura 64). O comprimento de 1524 aminoácidos é compatível com a proteína de P. canaliculata (ID: XP_025111754.1) de 1523 aminoácidos registrada no NCBI (National Centre for Biotechnology Information ― http://.ncbi.nlm.nih.gov/), com um percentual de identificação de 99,68% (1522/1524 aminoácidos) e um gap. Esses dados corroboram a identificação da provável proteína Drosha em P. canaliculata.
Para P. maculata, as proteínas Drosha apresentaram tamanho incompatível para os seus ortólogos em deuterostômios e protostômios (Tabela 10). No entanto, revelam indícios de serem prováveis proteínas. A sequências proteica (Pma51393_c0_g1) com 580 aminoácidos tem como ortólogo a proteína de P. canaliculata registrada no NCBI (ID: XP_025111754.1). Ao se realizar BLASTp no NCBI do peptídeo Pma51393_c0_g1 de P. maculata contra a proteína acima descrita, apresentou percentual de identificação de 99,68% (579/580) dos aminoácidos. Foi verificado parte dos aminoácidos catalíticos (E-D) presentes no primeiro domínio Riboc (Figura 64), apresentando apenas este domínio conservado para a provável proteína Pma51393_c0_g1. Em Drosha, provavelmente devido à falta de cobertura no sequenciamento desta região do genoma de P. maculata, não foi possível evidenciar a presença do segundo domínio catalítico Riboc. As regiões no genoma preditas para este gene provavelmente estão com erro de anotação levando ao truncamento das sequências proteicas. Consequentemente, o tamanho das sequências de aminoácidos para estes genes está com tamanho e estrutura subestimadas. Futuras versões do genoma podem resolver este problema (GOMES, 2012).
Tabela 17: Similaridade de Dicer de P. canaliculata e ortólogos.
Tabela 18: Domínios conservados de Dicer de P. canaliculata e ortólogos.
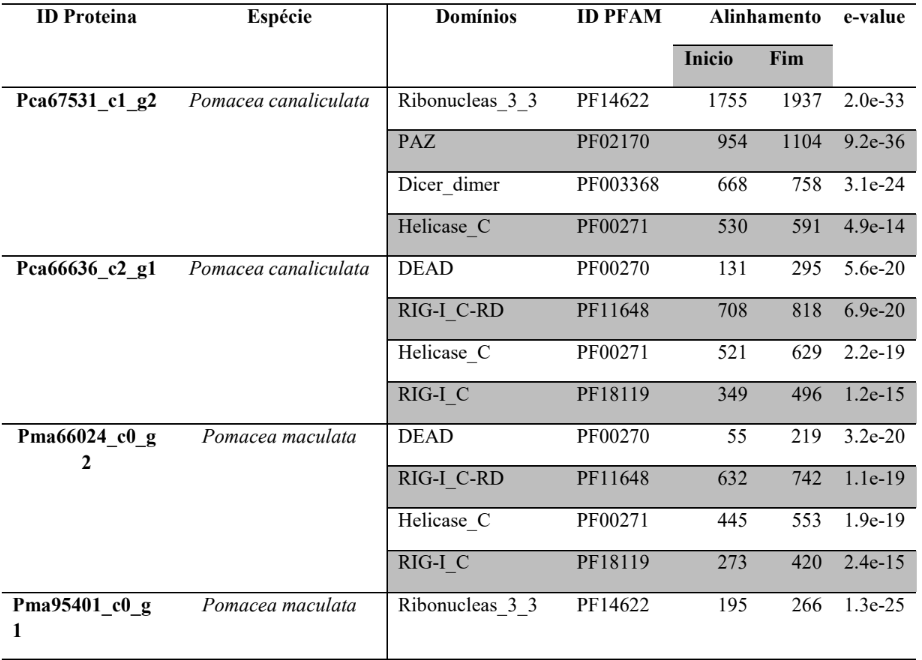
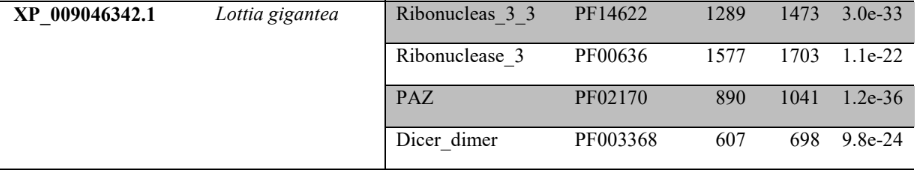
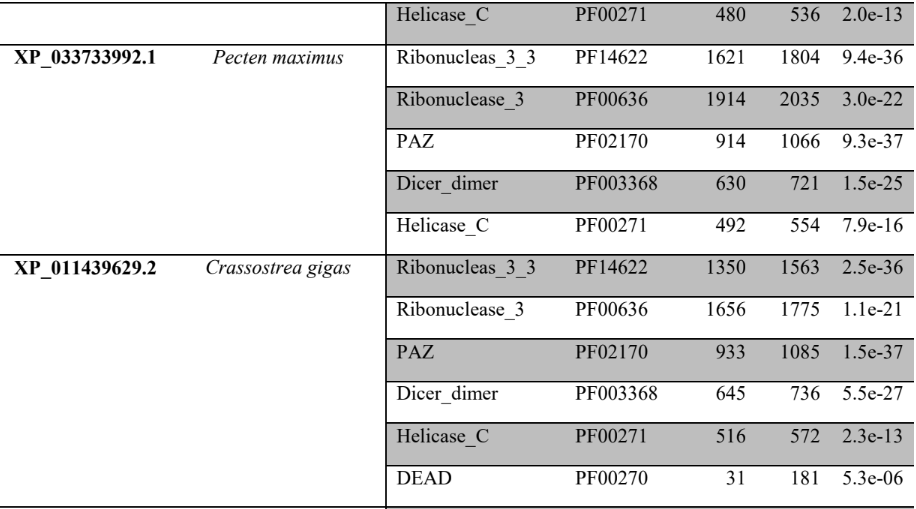
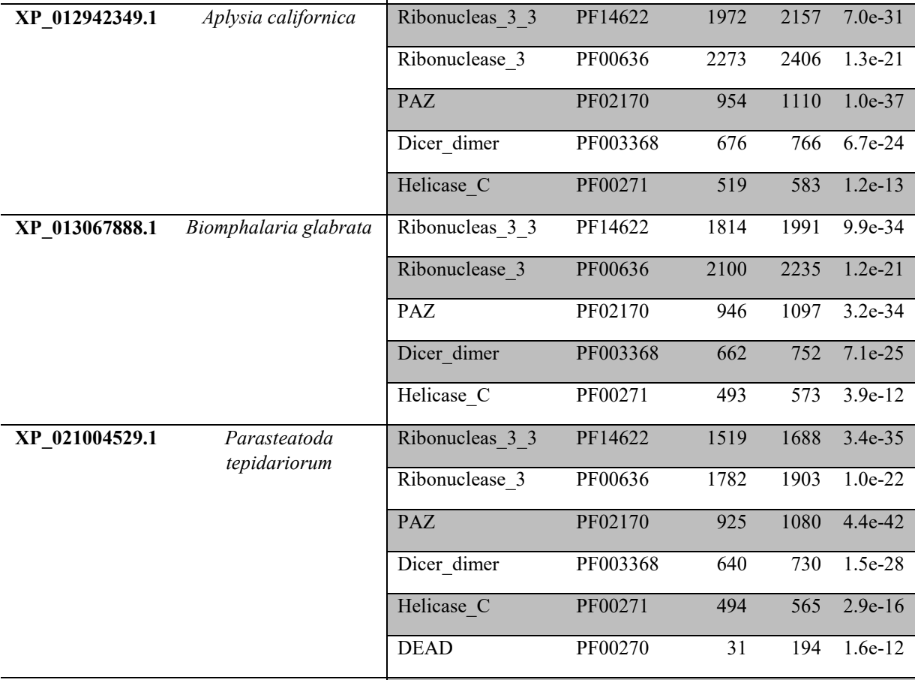
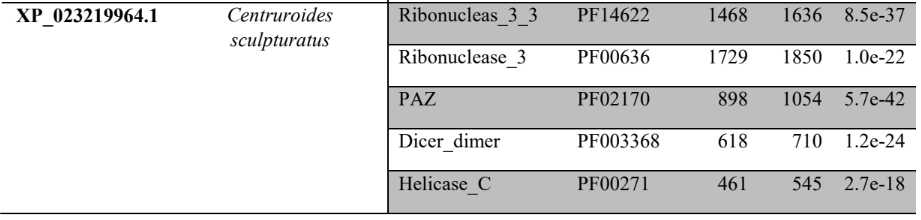
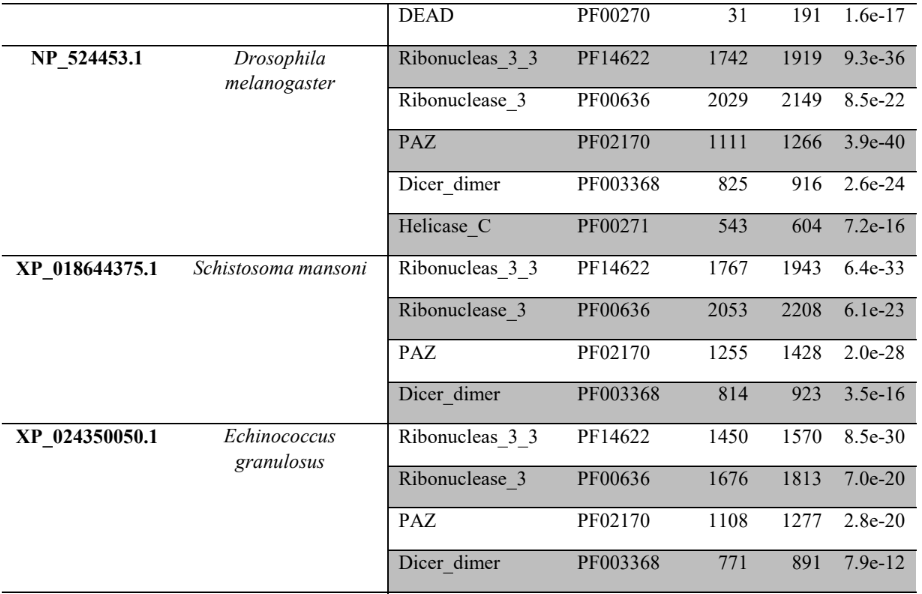
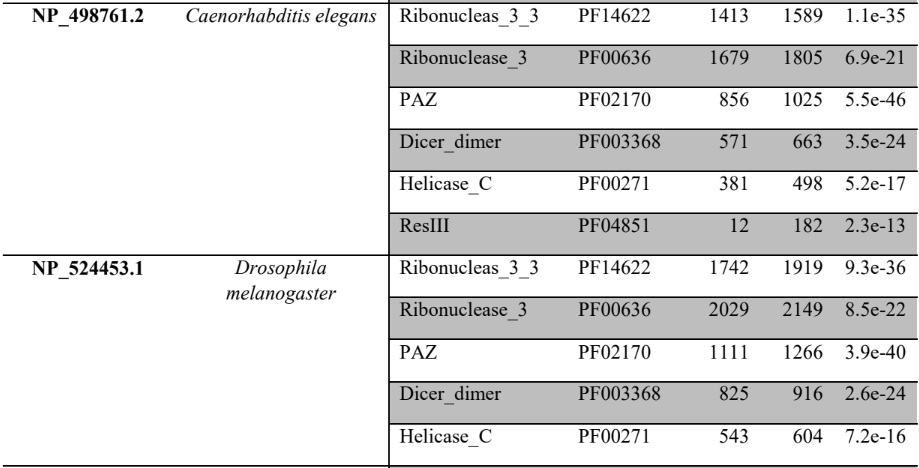

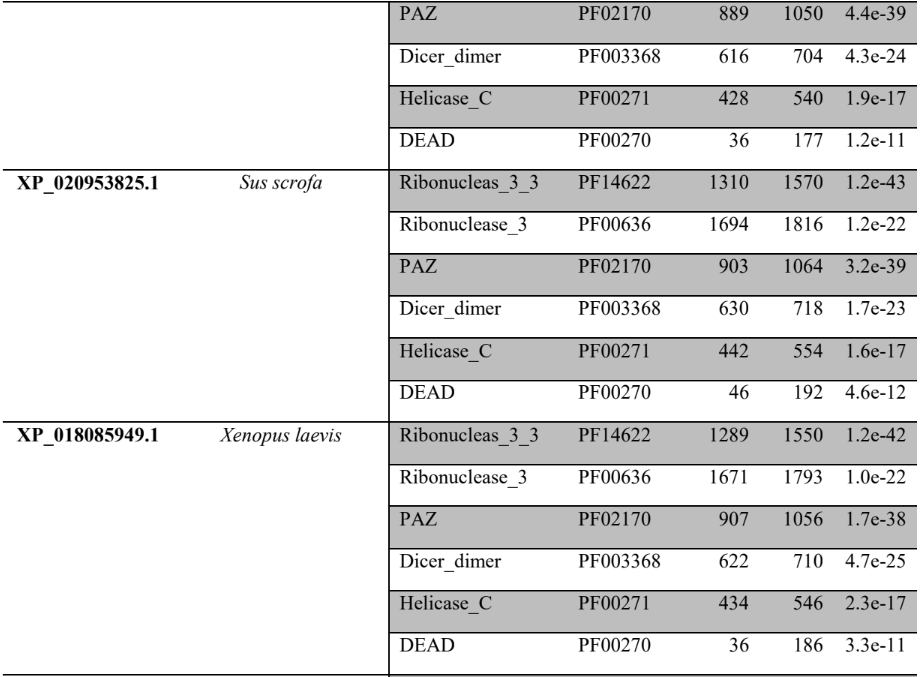
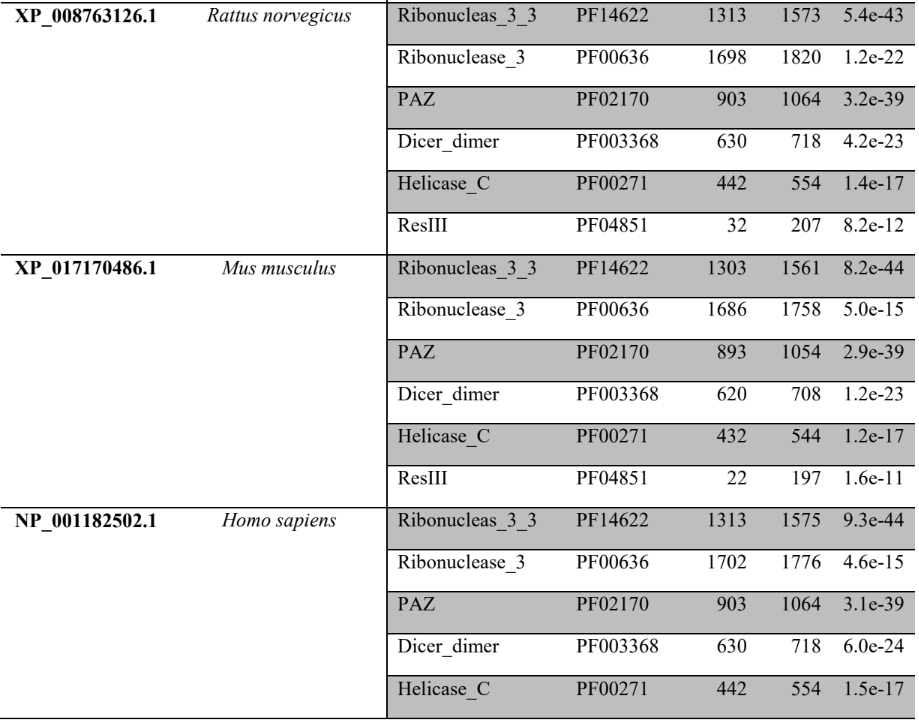

Figura 64: Alinhamento global, análise ribonuclease III (Riboc) das proteínas Dicer (acima representado) e Drosha (abaixo representado) de P. canaliculata e P. maculata e seus ortólogos
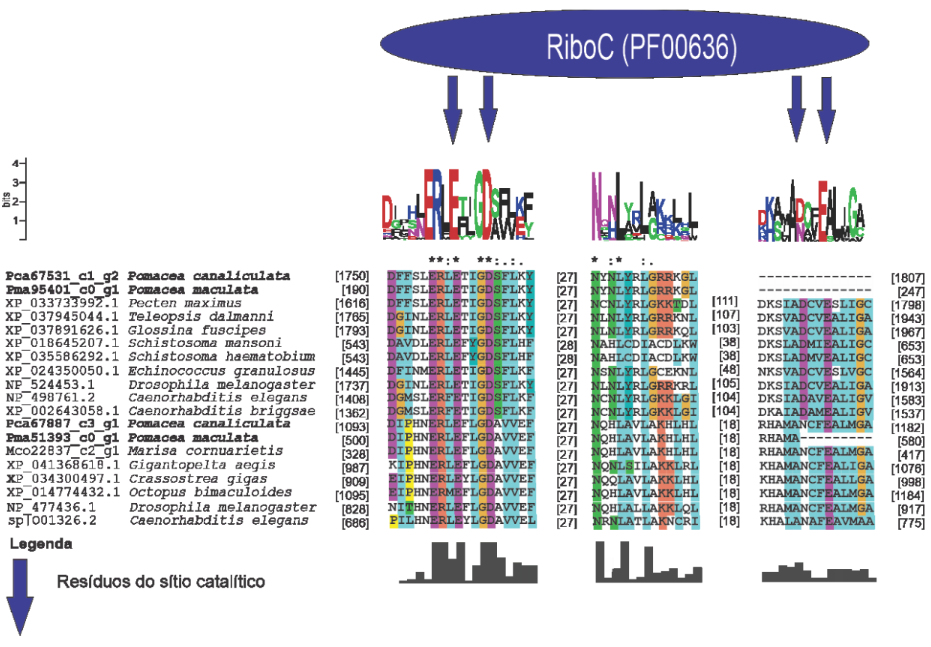
Figura 65: Análise filogenética das proteínas Dicer (acima representado) e Drosha (abaixo representado) de P. canaliculata e P. maculata e seus ortólogos.
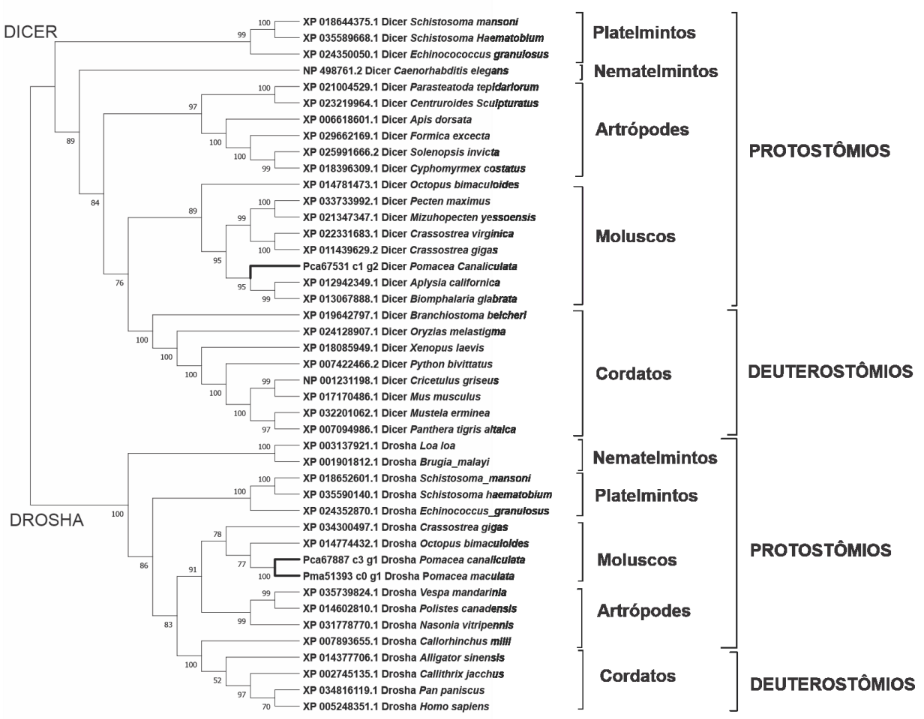
Figura 66: Domínios conservados de Dicer de P. canaliculata e ortólogos.

A candidata à sequência proteica Pca67531_c1_g2 de P. canaliculata (Pca_Dicer) apresentou comprimento similar (1984 aa) a organismos próximos evolutivamente, A. californica (2503 aa) e B. glabrata (2332 aa) (Tabelas 9 e 17). Foram identificados apenas os domínios conservados; Ribonucleas_3_3, PAZ, Dicer dimer e Helicase_C; não apresentando o segundo domínio Riboc (Tabela 18 e Figura 66). Ao verificar o posicionamento de início do segundo domínio Riboc de A. californica (2273 aa) e B. glabrata (2100 aa), corrobora o fato de não identificação da segunda ribonuclease na provável sequência proteica. Em Dicer, provavelmente devido à falta de cobertura no sequenciamento desta região do genoma de P. maculata, não foi possível evidenciar a presença do segundo domínio catalítico Riboc. A proteína Dicer de P. canaliculata (XP_025104500) identificada no NCBI, comprimento 2249 aminoácidos é ortologa a Pca67531_c1_g2, com uma cobertura da query de 99% e porcentagem de identificação de 98,83%. A sequência proteica Pca67531_c1_g2 ao utilizar a ferramenta BLASTp contra o genoma de P. canaliculata no banco de dados AmbuBase (Department of Computer Science HKBU – https://www.comp.hkbu.edu.hk/~db/AmpuBase/#&panel1-4) apresentou um percentual de identificação e cobertura de 100% (1984/1984 aminoácidos). Foi observado o primeiro sítio catalítico dos aminoácidos (E-D) na Riboc na proteína candidata citada (Pca67531_c1_g2) (Figura 64). Ao realizar a busca por resíduos catalíticos tendo como query NP_524453.1 Dicer-1 Drosophila melanogaster, os moluscos ortólogos apresentaram no primeiro domínio Riboc apenas os aminoácidos catalíticos (E-D), com exceção de Pectem Maximus (E-D-D-E) NCBI (http://.ncbi.nlm.nih.gov/). O alinhamento múltiplo das proteínas mostrou alta similaridade de sequências dos aminoácidos, particularmente em regiões de domínio funcional conservado (Figura 64). Dicer revelou ampla distribuição entre os clados filogenéticos na árvore filogenética, mostrando alta similaridade da proteína catalítica de P. canaliculata entre moluscos, artrópodes e cordados sobretudo (Figura 65). Os dados apresentados corroboram para a identificação da provável proteína.
Para P. maculata, as prováveis proteínas Dicer, Pma66024_c0_g2 (766 aa) e Pma95401_c0_g1 (288 aa) não apresentaram comprimento compatível com a dimensão média das proteínas ortólogas. As regiões no genoma preditas para este gene provavelmente estão com erro de anotação levando ao truncamento das sequências proteicas. Consequentemente, o tamanho das sequências de aminoácidos para estes genes está com tamanho e estrutura subestimadas. Futuras versões do genoma podem resolver esse problema (GOMES, 2012; QUEIROZ et al., 2017). A sequência proteica (Pma66024_c0_g2) não evidenciou os domínios de endonucleases, já a provável proteína (Pma95401_c0_g1) revelou apenas um domínico Riboc com resíduos catalíticos nos aminoácidos E-D (Tabelas 10 e 18, figura 66). A sequência proteica (Pma95401_c0_g1) de P. maculata é ortóloga as proteínas de P. canaliculata (XP_025104500) identificada no NCBI e Pca67531_c1_g2 (caracterizada neste trabalho), apresentado percentual de identidade de 97,22 % (280/288 aminoácidos) com a proteína de P. canaliculata identificada no NCBI.
4.7.3 Análise das proteínas DGCR8, Exportina 1 e T, ran
As proteínas Ras, Exportinas 1 e T, DGCR8 e TDRD1 em P. canaliculata e P. maculata foram analisadas através da homologia, tamanho da proteína e domínios conservados.
4.7.3.1 DGCR8
A sequência proteica DGCR8 (DiGeorge Syndrome Critical Region 8) faz parte do complexo de microprocessadores que realiza a clivagem do pri-miRNA (transcrito do DNA) em pré-miRNA. O DGCR8 se liga em uma estrutura no pri-miRNA; enquanto, Drosha realiza a cisão do duplex. Esta etapa realizada no núcleo celular é primordial para a biogênese dos miRNAs, reguladores pós-transcricionais da expressão gênica (HAN et al., 2004).
Tabela 19: Análise de similaridade de DGCR8 de P. maculata e ortólogos.
Tabela 20: Análise de similaridade de DGCR8 de P. canaliculata e ortólogos.
A proteína identificada no NCBI subunidade DGCR8 (XP_025088948.1) de P. canaliculata apresenta o mesmo conjunto de aminoácidos (730/730), 100% de percentual de identificação que a provável sequência proteica predita neste trabalho (Pca59979_c0_g1) de P. canaliculata. A sequência de aminoácidos (Pma62419_c0_g1) de P. maculata (708 aa) revelou um percentual de identificação de 99,71% com a proteína de P. canaliculata acima citada identificada no NCBI. As sequências proteicas preditas de P. maculata e P. canaliculata (DGCR8) revelam tamanho compatível e similaridade com ortólogos (Tabelas 19 e 20).
4.7.3.2 Exportina 1 e T
Neste trabalho, foram preditas 3 sequências proteicas pertencentes à família de proteínas exportinas (XPO1 e XPOT) no genoma dos caracóis; P. canaliculata (Pca65193_c2_g1) e P. maculata (Pma59467_c0_g1), Exportina 1; P. maculata (Pma63331_c0_g1) exportina T. Intrinsecamente, o transporte de precursores de miRNAs é executado pela proteína XPO5. Ocasionalmente, o transporte de pré-miRNAs pode ser realizado por XPO1 entrando na via de processamento não canônico de miRNAs sem a clivagem por Drosha. O deslocamento de pré- miRNAs também é executado pela exportina T (XPOT) de maneira dependente de Ran-GTP (RUBY; JAN; BARTEL, 2007; XIE et al., 2013).
Tabela 21 – Similaridade de XPO-1 de P. maculata e ortólogos.
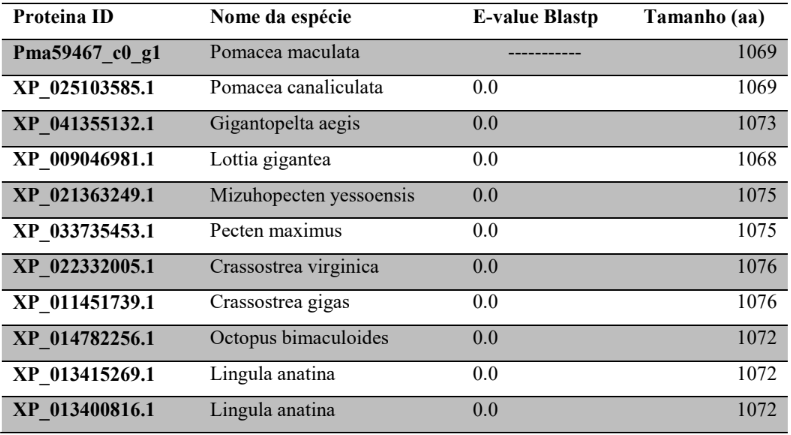
Tabela 22 – Similaridade de XPO-T de P. maculata e ortólogos.
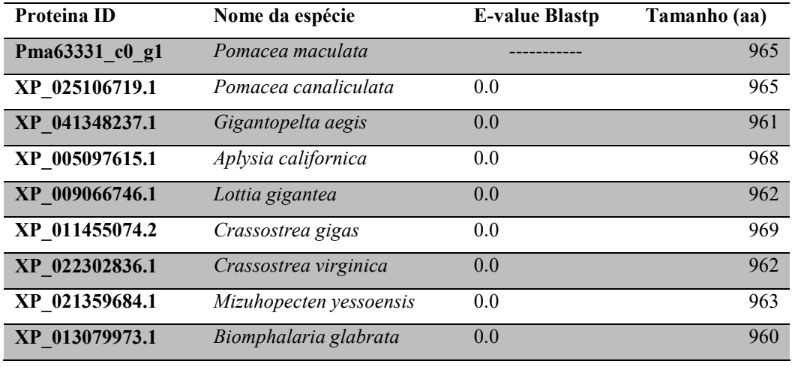
Figura 67: Árvore filogenética XPO-1 e XPO-T P. canaliculata e P. maculata.

Figura 68: Análise de domínios conservados de P. maculata e P. canaliculata – XPO-1.
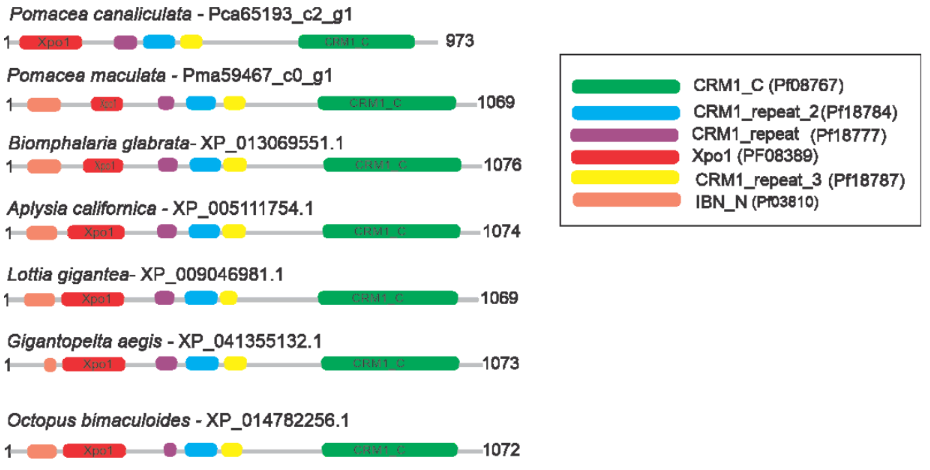
Com o objetivo de enfatizar a conservação destas prováveis proteínas identificados nas espécies em estudo, foi construído uma árvore filogenética e estudos de domínios conservados. A análise filogenética revelou uma distribuição das sequências de aminoácidos (Pca65193_c2_g1 e Pma59467_c0_g1) – XPO-1 e (Pma63331_c0_g1) – XPO-T, agrupadas próximas ao clado Deuterostômio. As prováveis proteínas de P. maculata e P. canaliculata agregaram-se com as respectivas ortólogas próximas ao filo Mollusca e ao clado Deuterostômio (Figura 67).
A similaridade das Exportinas identificadas do gênero Pomacea das espécies em estudo foi verificada junto às suas ortólogas observando nível de preservação dos domínios conservados e comprimento. A proteína predita de P. maculata (Pma59467_c0_g1) apresentou os mesmos domínios conservados (XPO-1) de suas ortólogas e comprimento compatível com organismos próximos evolutivamente (Tabela 21 e Figura 68). A sequência proteica de P. canaliculata (Pca65193_c2_g1) apresentou um domínio conservado a menos que seus ortólogos, revelando tamanho ligeiramente inferior (Figura 68). A falta de cobertura na predição da proteína prejudicou a predição de domínios conservados, uma vez que, ela se deu na porção N-terminal da proteína. A proteína predita (XPO-T) de P. maculata (Pma63331_c0_g1) apresentou comprimento compatível e similaridade com suas ortólogas, organismos próximos evolutivamente (Tabela 22).
4.7.3.3 Ran
A proteína Ran executa importante função na via de miRNAs. Na via canônica da biogênese do miRNAs, após a cisão dos pri-miRNAs em pré-miRNAs no núcleo celular os precursores precisam ser exportados para o citoplasma. Esta etapa é dependente complexo XPO5-Ran, que se ligam aos pré-miRNAs com dispêndio de GTP (YI et al., 2003).
Observou-se que a sequência proteica de P. canaliculata (Pca52466_c2_g3_Ran) apresentou o mesmo comprimento e 100% de identificação com os aminoácidos da proteína já identificada da espécie (XP_025090191.1) no NCBI com 214 aminoácidos. Da mesma forma, a sequência proteica de P. maculata (Pma54972_c0_g) de 152 aminoácidos, provavelmente truncadas no genoma, apresentou percentual de identificação de 100% com a proteína de P. canaliculata (XP_025090191.1) registrada no NCBI (National Centre for Biotechnology Information ― http://.ncbi.nlm.nih.gov/). As regiões no genoma preditas para o gene de Ran de P. maculata provavelmente estão com o erro de anotação levando ao truncamento da sequência proteica ocasionando o tamanho relativamente menor da proteína predita comparados com suas ortólogas (Tabela 23).
Tabela 23 – Similaridade de Ran de P. maculata e ortólogos.
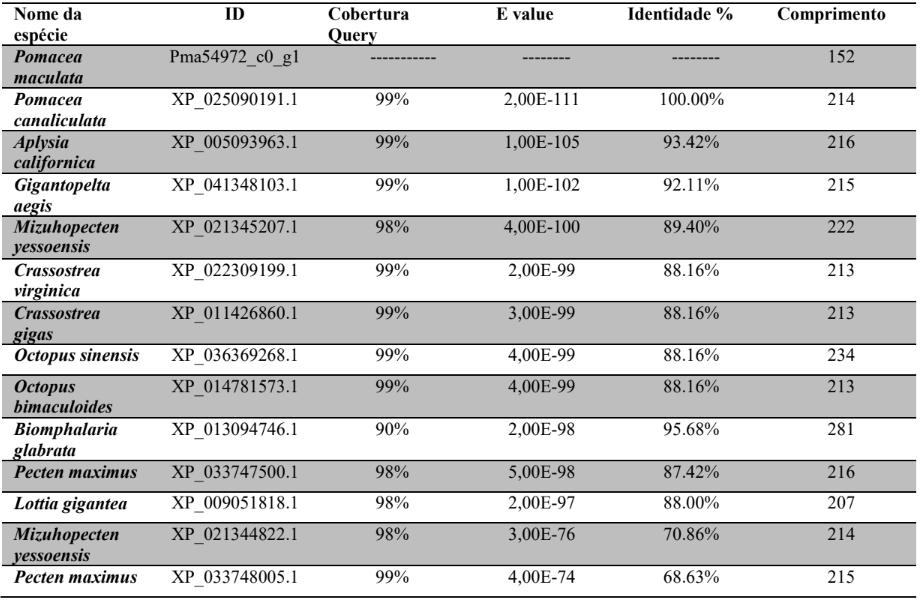
Conforme observado nas tabelas descritas acima, as proteínas Ran, Exportinas 1 e T, DGCR8 de P. canaliculata e P. maculata possuem alta similaridade com seus ortólogos, tamanho compatível com a dimensão média das proteínas ortólogas e domínios conservados característicos de acordo com a função da proteína. Desta forma, foi possível identificar estas prováveis proteínas nas espécies em estudo.
Assim, foi possível identificar 7 prováveis proteínas da via de miRNas em P. canaliculata, Pca69814_c1_g2 (Argonauta 2), Pca61914_c0_g1(Argonauta), Pca67531_c1_g2 (Dicer), Pca67887_c3_g1 (Drosha), Pca59979_c0_g1 (DGRC8), Pca65193_c2_g1 (Exportina 1), Pca52466_c2_g3 (Ran) e 8 prováveis proteínas em P. maculata, Pma63715_c0_g1 (Argonauta 2), Pma60501_c2_g16 (Argonauta), Pma62419_c0_g1 (DGRC8), Pma59467_c0_g1 (Exportina 1), Pma63331_c0_g1 (Exportina T), Pma54972_c0_g1 (Ran), Pma51393_c0_g1 (Drosha), Pma95401_c0_g1 (Dicer).
5 CONCLUSÃO
A pesquisa por miRNAs em P. canaliculata e P. maculata a partir de bancos de dados públicos atendeu as características estruturais e termodinâmicas de miRNAs identificados, para elucidação e predição de miRNAs conservados nos genomas das espécies. Foi possível predizer 296 formas maduras de miRNAs conservados em P. canaliculata e 316 miRNAs em P. maculata. A submissão de alguns pré-miRNAs ao alinhamento múltiplo de sequências revelou alta similaridade com seus ortólogos.
Como mostrado, os resultados indicam que algumas sequências analisadas de mir- bantam e mir-1990 de P. canaliculata e P. maculata mostraram 100% de identificação com alguns de seus ortólogos. A fim de evidenciar a conservação de miRNAs preditos em P. maculata e P. canaliculata foram construídas árvores filogenéticas, nas quais foram confirmadas a estreita proximidade entre as espécies estudadas e os moluscos da família Ampullariidae, Lanistes nyassanus e Marisa cornuarietis. A sequência madura de miRNAs se mostrou conservada evolutivamente e esta similaridade aumenta à medida que o grau de parentesco entre as espécies se acentua. Da mesma forma, foi verificada alta similaridade com a estrutura secundária dos precursores de miRNAs preditos e ortólogos, intrinsecamente semelhantes entre moluscos e a família Ampullariidae.
Uma das formas de demonstrar a presença de miRNAs no genoma das espécies: P. canaliculata e P. maculata é verificar a sua maquinaria de processamento. Dessa forma, foi possível identificar a via de processamento de miRNAs nas espécies: P. canaliculata e P. maculata, destacando várias prováveis proteínas da via, tais como Argonauta, Drosha, Dicer, DGCR8, XPO1, dentre outras. A caracterização das proteínas da via através de alinhamento múltiplo de sequências, análise de domínios conservados, analogia com similaridade e dimensionamento com ortólogos permitiu confirmar através de análise filogenéticas a conservação e a presença de prováveis proteínas detectas no proteoma das espécies.
As características inerentes de P. canaliculata e P. maculata referentes ao aspecto invasivo puderam ser ratificadas por mapeamento de características por lócus gênico em estudos anteriores. Estudos de expressão diferencial de miRNAs poderão ser conduzidos na região dos tentáculos cefálicos e palpos labiais, para ratificar estes dados, bem como estabelecer o gene GRL101 como alvo para os miRNAs descobertos neste trabalho. Dessa forma, poderá ser correlacionado o aspecto invasivo das espécies P. canaliculata e P. maculata a partir da regulação pós-transcricional de miRNAs em genes específicos das espécies.
Por fim, os nossos resultados ampliam o estudo dos miRNAs em moluscos, proporcionando novos desafios para compreensão de processos essenciais relacionados à invasividade das espécies P. canaliculata e P. maculata.
REFERÊNCIAS
ACCORSI, Alice; BUCCI, Laura; DE EGUILEOR, Magda; OTTAVIANI, Enzo; MALAGOLI, Davide. Comparative analysis of circulating hemocytes of the freshwater snail Pomacea canaliculata. Fish and Shellfish Immunology, [S. l.], v. 34, n. 5, p. 1260–1268, 2013. https://doi.org/10.1016/j.fsi.2013.02.008
ADEMA, Coen M. et al. Whole genome analysis of a schistosomiasis-transmitting freshwater snail. Nature Communications, [S. l.], v. 8, n. May, 2017. https://doi.org/10.1038/ncomms15451
ALARCÓN, Claudio R.; LEE, Hyeseung; GOODARZI, Hani; HALBERG, Nils; TAVAZOIE, Sohail F. N6-methyladenosine marks primary microRNAs for processing. Nature, [S. l.], v. 519, n. 7544, p. 482–485, 2015. https://doi.org/10.1038/nature14281
ALBERTIN, Caroline B.; SIMAKOV, Oleg; MITROS, Therese; WANG, Z. Yan; PUNGOR, Judit R.; EDSINGER-GONZALES, Eric; BRENNER, Sydney; RAGSDALE, Clifton W.; ROKHSAR, Daniel S. The octopus genome and the evolution of cephalopod neural and morphological novelties. Nature, [S. l.], v. 524, n. 7564, p. 220–224, 2015. https://doi.org/10.1038/nature14668
ANFOSSI, Simone; BABAYAN, Anna; PANTEL, Klaus; CALIN, George A. Clinical utility of circulating non-coding RNAs — an update. Nature Reviews Clinical Oncology, [S. l.], v. 15, n. 9, p. 541–563, 2017. https://doi.org/10.1038/s41571-018-0035-x
BABIARZ, Joshua E.; RUBY, J. Graham; WANG, Yangming; BARTEL, David P.; BLELLOCH, Robert. Mouse ES cells express endogenous shRNAs, siRNAs, and other microprocessor-independent, dicer-dependent small RNAs. Genes and Development, [S. l.], v. 22, n. 20, p. 2773–2785, 2008. https://doi.org/10.1101/gad.1705308
BAO, Yongbo; ZHANG, Lili; DONG, Yinghui; LIN, Zhihua. Identification and comparative analysis of the tegillarca granosa haemocytes MicroRNA transcriptome in response to Cd using a deep sequencing approach. PLoS ONE, [S. l.], v. 9, n. 4, 2014. https://doi.org/10.1371/journal.pone.0093619
BARTEL, David P. MicroRNAs: Target Recognition and Regulatory Functions. Cell, [S.l.], v. 136, n. 2, p. 215–233, 2009. https://doi.org/10.1016/j.cell.2009.01.002
BEHM-ANSMANT, Isabelle; REHWINKEL, Jan; DOERKS, Tobias; STARK, Alexander; BORK, Peer; IZAURRALDE, Elisa. mRNA degradation by miRNAs and GW182 requires both CCR4:NOT deadenylase and DCP1:DCP2 decapping complexes. Genes and Development, [S. l.], v. 20, n. 14, p. 1885–1898, 2006. https://doi.org/10.1101/gad.1424106
BERNSTEIN, Emily; CAUDY, Amy A.; HAMMOND, Scott M.; HANNON, Gregory J. Role for a bidentate ribonuclease in the initiation step of RNA interference. Nature, [S. l.], v. 409, n. 6818, p. 363–366, 2001. https://doi.org/10.1038/35053110
BIGGAR, Kyle K.; KORNFELD, Samantha F.; MAISTROVSKI, Yulia; STOREY, Kenneth B. MicroRNA Regulation in Extreme Environments: Differential Expression of MicroRNAs in the Intertidal Snail Littorina littorea During Extended Periods of Freezing and Anoxia. Genomics, Proteomics and Bioinformatics, [S. l.], v. 10, n. 5, p. 302–309, 2012. https://doi.org/10.1016/j.gpb.2012.09.002
BLASZCZYK, Jaroslaw; TROPEA, Joseph E.; BUBUNENKO, Mikhail; ROUTZAHN, Karen M.; WAUGH, David S.; COURT, Donald L.; JI, Xinhua. Crystallographic and modeling studies of RNase III suggest a mechanism for double-stranded RNA cleavage. Structure, [S. l.], v. 9, n. 12, p. 1225–1236, 2001. https://doi.org/10.1016/S0969-2126(01)00685-2
BRACKEN, C. P.; GREGORY, P. A.; KHEW-GOODALL, Y.; GOODALL, G. J. The role of microRNAs in metastasis and epithelial-mesenchymal transition. Cellular and Molecular Life Sciences, [S. l.], v. 66, n. 10, p. 1682–1699, 2009. https://doi.org/10.1007/s00018-009-8750-1
BREDY, Timothy W.; LIN, Quan; WEI, Wei; BAKER-ANDRESEN, Danay; MATTICK, John S. MicroRNA regulation of neural plasticity and memory. Neurobiology of Learning and Memory, [S. l.], v. 96, n. 1, p. 89–94, 2011. https://doi.org/10.1016/j.nlm.2011.04.004
BRENNECKE, Julius; HIPFNER, David R.; STARK, Alexander; RUSSELL, Robert B.; COHEN, Stephen M. bantam encodes a developmentally regulated microRNA that controls cell proliferation and regulates the proapoptotic gene hid in Drosophila. Cell, [S. l.], v. 113, n. 1, p. 25–36, 2003. https://doi.org/10.1016/S0092-8674(03)00231-9
BROUGHTON, James P.; LOVCI, Michael T.; HUANG, Jessica L.; YEO, Gene W.; PASQUINELLI, Amy E. Pairing beyond the Seed Supports MicroRNA Targeting Specificity. Molecular Cell, [S. l.], v. 64, n. 2, p. 320–333, 2016. https://doi.org/10.1016/j.molcel.2016.09.004.
BROWN, Kenneth M.; LYDEARD, Charles. Mollusca: Gastropoda. Third Edit ed. [s.l.]: Elsevier Ltd, 2010. DOI: 10.1016/B978-0-12-374855-3.00010-8. https://doi.org/10.1016/B978-0-12-374855-3.00010-8
BURGOS-ACEVES, Mario Alberto; COHEN, Amit; SMITH, Yoav; FAGGIO, Caterina. MicroRNAs and their role on fish oxidative stress during xenobiotic environmental exposures. Ecotoxicology and Environmental Safety, [S. l.], v. 148, n. November 2017, p. 995–1000, 2018. https://doi.org/10.1016/j.ecoenv.2017.12.001
CARDOSO, TCS; DE ARAUJO, CB; PORTILHO, LG; ET.AL. Computational Prediction and Characterisation of miRNAs and Their Pathway Genes in Human Schistosomiasis Caused by Schistosoma Haematobium. Men Inst Oswaldo Cruz., [S. l.], v. 115, 2020. https://doi.org/10.1590/0074-02760190378
CHELOUFI, Sihem; DOS SANTOS, Camila O.; CHONG, Mark M. W.; HANNON, Gregory J. A dicer-independent miRNA biogenesis pathway that requires Ago catalysis. Nature, [S. l.], v. 465, n. 7298, p. 584–589, 2010. https://doi.org/10.1038/nature09092
CHEN, C. Y.; KUO, C. C.; LO, Chung Ping; HUANG, M. Y.; WANG, Y. M.; WANG, W. Y. Eosinophilic meningoencephalitis caused by Angiostrongylus cantonesis. Qjm, [S. l.], v. 107, n. 7, p. 573–575, 2014. a. https://doi.org/10.1093/qjmed/hcr261
CHEN, Hao et al. The comprehensive immunomodulation of NeurimmiRs in haemocytes of oyster Crassostrea gigas after acetylcholine and norepinephrine stimulation. BMC Genomics, [S. l.], v. 16, n. 1, p. 1–14, 2015. https://doi.org/10.1186/s12864-015-2150-8
CHEN, Jian Fu; MANDEL, Elizabeth M.; THOMSON, J. Michael; WU, Qiulian; CALLIS, Thomas E.; HAMMOND, Scott M.; CONLON, Frank L.; WANG, Da Zhi. The role of microRNA-1 and microRNA-133 in skeletal muscle proliferation and differentiation. Nature Genetics, [S. l.], v. 38, n. 2, p. 228–233, 2006. https://doi.org/10.1038/ng1725
CHEN, Peijie et al. miR-216a-5p acts as an oncogene in renal cell carcinoma. Experimental and Therapeutic Medicine, [S. l.], v. 15, n. 4, p. 4039–4046, 2018. https://doi.org/10.3892/etm.2018.5881
CHEN, Shuai; MCKINNEY, Garrett J.; NICHOLS, Krista M.; SEPÚLVEDA, Maria S. In silico prediction and in vivo validation of Daphnia pulex micrornas. PLoS ONE, [S. l.], v. 9, n. 1, p. 1–5, 2014. b. https://doi.org/10.1371/journal.pone.0083708
CLERBAUX, Laure-Alix; SCHULTZ, H.; ROMAN-HOLBA, S.; RUAN, DF; YU, R.; LAMB, AM; BOMMER, GT; KENNELL, JA. The microRNA miR-33 is a pleiotropic regulator of metabolic and developmental processes in Drosophila melanogaster. Developmental Dynamics, [S. l.], 2021. https://doi.org/10.1002/dvdy.344
DE RIE, Derek et al. An integrated expression atlas of miRNAs and their promoters in human and mouse. Nature Biotechnology, [S. l.], v. 35, n. 9, p. 872–878, 2017. https://doi.org/10.1038/nbt.3947
DE SOUZA GOMES, Matheus. miRNAs em Schistosoma mansoni : biogênese, predição, validação e alvos potenciais. 2012. Universidade Federal de Ouro Preto, [S. l.], 2012. Disponível em: http://www.repositorio.ufop.br/jspui/handle/123456789/2662.
DE SOUZA GOMES, Matheus; MUNIYAPPA, Mohan Kumar; CARVALHO, Sávio Gonçalves; GUERRA-SÁ, Renata; SPILLANE, Charles. Genome-wide identification of novel microRNAs and their target genes in the human parasite Schistosoma mansoni. Genomics, [S. l.], v. 98, n. 2, p. 96–111, 2011. https://doi.org/10.1016/j.ygeno.2011.05.007
DE WIT, Elzo; LINSEN, Sam E. V.; CUPPEN, Edwin; BEREZIKOV, Eugene. Repertoire and evolution of miRNA genes in four divergent nematode species. Genome Research, [S. l.], v. 19, n. 11, p. 2064–2074, 2009. https://doi.org/10.1101/gr.093781.109
DENLI, Ahmet M.; TOPS, Bastiaan B. J.; PLASTERK, Ronald H. A.; KETTING, René F.; HANNON, Gregory J. Processing of primary microRNAs by the Microprocessor complex. Nature, [S. l.], v. 432, n. 7014, p. 231–235, 2004. https://doi.org/10.1038/nature03049
DO ESPÍRITO-SANTO, Maria Cristina Carvalho; PINTO, Pedro Luiz Silva; DA MOTA, Dan Jesse Gonçalves; GRYSCHEK, Ronaldo César Borges. Primeiro caso de meningite eosinofílica diagnosticado na cidade de Sáo Paulo, SP, Brasil. Revista do Instituto de Medicina Tropical de Sao Paulo, [S. l.], v. 55, n. 2, p. 129–132, 2013. https://doi.org/10.1590/S0036-46652013000200012
DREON, Marcos S.; SCHINELLA, Guillermo; HERAS, Horacio; POLLERO, Ricardo J. Antioxidant defense system in the apple snail eggs, the role of ovorubin. Archives of Biochemistry and Biophysics, [S. l.], v. 422, n. 1, p. 1–8, 2004. https://doi.org/10.1016/j.abb.2003.11.018.
DREON, Marcos Sebastián; FRASSA, María Victoria; CEOLÍN, Marcelo; ITUARTE, Santiago; QIU, Jian Wen; SUN, Jin; FERNÁNDEZ, Patricia E.; HERAS, Horacio. Novel Animal Defenses against Predation: A Snail Egg Neurotoxin Combining Lectin and Pore- Forming Chains That Resembles Plant Defense and Bacteria Attack Toxins. PLoS ONE, [S. l.], v. 8, n. 5, 2013. https://doi.org/10.1371/journal.pone.0063782
DU, Xiaodong et al. The pearl oyster Pinctada fucata martensii genome and multi-omic analyses provide insights into biomineralization. GigaScience, [S. l.], v. 6, n. 8, 2017. https://doi.org/10.1093/gigascience/gix059
EL-GEBALI, Sara et al. The Pfam protein families database in 2019. Nucleic Acids Research, [S. l.], v. 47, n. D1, p. D427–D432, 2019. https://doi.org/10.1093/nar/gky995
ELBASHIR, S. M.; LENDECKEL, W.; TUSCHL, T. RNA interference is mediated by 21- and 22-nucleotide RNAs. Genes and Development, [S. l.], v. 15, n. 2, p. 188–200, 2001. https://doi.org/10.1101/gad.862301
ESTEBENET, Alejandra L.; MARTIN, Pablo R. Shell interpopulation variation and its origin in Pomacea canaliculata (Gastropoda: Ampullariidae) from Southern Pampas, Argentina. Journal of Molluscan Studies, [S. l.], v. 69, n. 4, p. 301–310, 2003. https://doi.org/10.1093/mollus/69.4.301.
FABIAN, Marc Robert; SONENBERG, Nahum; FILIPOWICZ, Witold. Regulation of mRNA Translation and Stability by microRNAs. Annual Review of Biochemistry, [S. l.], v. 79, n. 1, p. 351–379, 2010. https://doi.org/10.1146/annurev-biochem-060308-103103
FENG, Dandan; LI, Qi; YU, Hong; LIU, Shikai; KONG, Lingfeng; DU, Shaojun. Integrated analysis of microRNA and mRNA expression profiles in Crassostrea gigas to reveal functional miRNA and miRNA-targets regulating shell pigmentation. [S. l.], 2020. https://doi.org/10.1038/s41598-020-77181-0
FERNANDEZ-MERCADO, Marta; MANTEROLA, Lorea; LARREA, Erika; GOICOECHEA, Ibai; ARESTIN, María; ARMESTO, María; OTAEGUI, David; LAWRIE, Charles H. The circulating transcriptome as a source of non-invasive cancer biomarkers: Concepts and controversies of non-coding and coding RNA in body fluids. Journal of Cellular and Molecular Medicine, [S. l.], v. 19, n. 10, p. 2307–2323, 2015. https://doi.org/10.1111/jcmm.12625
FIGUERAS, Antonio; MOREIRA, Rebeca; SENDRA, Marta; NOVOA, Beatriz. Genomics and immunity of the Mediterranean mussel Mytilus galloprovincialis in a changing environment. Fish and Shellfish Immunology, [S. l.], v. 90, n. April, p. 440–445, 2019. https://doi.org/10.1016/j.fsi.2019.04.064.
FILIPPOV, Valery; SOLOVYEV, Victor; FILIPPOVA, Maria; GILL, Sarjeet S. A novel type of RNase III family proteins in eukaryotes. Gene, [S. l.], v. 245, n. 1, p. 213–221, 2000. https://doi.org/10.1016/S0378-1119(99)00571-5
FINN, Robert D. et al. The Pfam protein families database. Nucleic Acids Research, [S.l.], v. 38, n. SUPPL.1, p. 211–222, 2010. https://doi.org/10.1093/nar/gkp985
FRIEDLÄNDER, Marc R. et al. Evidence for the biogenesis of more than 1,000 novel human microRNAs. Genome Biology, [S. l.], v. 15, n. 4, p. 1–17, 2014. https://doi.org/10.1186/gb-2014-15-4-r57
FRIEDMAN, Robin C.; FARH, Kyle Kai How; BURGE, Christopher B.; BARTEL, David P. Most mammalian mRNAs are conserved targets of microRNAs. Genome Research, [S. l.], v. 19, n. 1, p. 92–105, 2009. https://doi.org/10.1101/gr.082701.108
GHILDIYAL, Megha; XU, Jia; SEITZ, Hervé; WENG, Zhiping; ZAMORE, Phillip D. Sorting of drosophila small silencing RNAs partitions microRNA* strands into the RNA interference pathway. Rna, [S. l.], v. 16, n. 1, p. 43–56, 2010. https://doi.org/10.1261/rna.1972910
GHILDIYAL, Megha; ZAMORE, Phillip D. Small silencing RNAs: An expanding universe. Nature Reviews Genetics, [S. l.], v. 10, n. 2, p. 94–108, 2009. https://doi.org/10.1038/nrg2504
GOMES-DOS-SANTOS, André; LOPES-LIMA, Manuel; CASTRO, L. Filipe C.; FROUFE, Elsa. Molluscan genomics: the road so far and the way forward. Hydrobiologia, [S. l.], v. 847, n. 7, p. 1705–1726, 2020. https://doi.org/10.1007/s10750-019-04111-1
GUO, Yunhai et al. A chromosomal-level genome assembly for the giant African snail Achatina fulica. GigaScience, [S. l.], v. 8, n. 10, p. 1–8, 2019. https://doi.org/10.1093/gigascience/giz124
HA, Minju; KIM, V. Narry. Regulation of microRNA biogenesisNature Reviews Molecular Cell BiologyNature Publishing Group, , 2014. https://doi.org/10.1038/nrm3838
HALWART, Matthias. The golden apple snail Pomacea canaliculata in asian rice farming systems: Present impact and future threat. International Journal of Pest Management, [S. l.], v. 40, n. 2, p. 199–206, 1994. https://doi.org/10.1080/09670879409371882.
HAMMOND, Scott M.; BERNSTEIN, Emily; BEACH, David; HANNON, Gregory J. An RNA-directed nuclease mediates post-transcriptional gene silencing in Drosophila cells. Nature, [S. l.], v. 404, n. 6775, p. 293–296, 2000. https://doi.org/10.1038/35005107
HAN, Jinju; LEE, Yoontae; YEOM, Kyu Hyun; KIM, Young Kook; JIN, Hua; KIM, V. Narry. The Drosha-DGCR8 complex in primary microRNA processing. Genes and Development, [S. l.], v. 18, n. 24, p. 3016–3027, 2004. https://doi.org/10.1101/gad.1262504
HASZPRUNAR, Gerhard; WANNINGER, Andreas. Molluscs. Current Biology, [S. l.], v. 22, n. 13, p. R510–R514, 2012. https://doi.org/10.1016/j.cub.2012.05.039
HAYES, Kenneth A.; COWIE, Robert H.; JØRGENSEN, Aslak; SCHULTHEISS, Roland; ALBRECHT, Christian; THIENGO, Silvana C. Molluscan Models in Evolutionary Biology: Apple Snails (Gastropoda: Ampullariidae) as a System for Addressing Fundamental Questions*. American Malacological Bulletin, [S. l.], v. 27, n. 1–2, p. 47–58, 2009. https://doi.org/10.4003/006.027.0204
HAYES, Kenneth A.; COWIE, Robert H.; THIENGO, Silvana C.; STRONG, Ellen E. Comparing apples with apples: Clarifying the identities of two highly invasive Neotropical Ampullariidae (Caenogastropoda). Zoological Journal of the Linnean Society, [S. l.], v. 166, n. 4, p. 723–753, 2012. https://doi.org/10.1111/j.1096-3642.2012.00867.x
HERAS, Horacio; FRASSA, M. Victoria; FERNÁNDEZ, Patricia E.; GALOSI, Cecilia M.; GIMENO, Eduardo J.; DREON, Marcos S. First egg protein with a neurotoxic effect on mice. Toxicon, [S. l.], v. 52, n. 3, p. 481–488, 2008. https://doi.org/10.1016/j.toxicon.2008.06.022
HERAS, Horacio; GARIN, Claudia F.; POLLERO, Ricardo J. Biochemical composition and energy sources during embryo development and in early juveniles of the snail Pomacea canaliculata (Mollusca: Gastropoda). Journal of Experimental Zoology, [S. l.], v. 280, n. 6, p. 375–383, 1998. https://doi.org/10.1002/(SICI)1097-010X(19980415)280:6<375::AID- JEZ1>3.0.CO;2-K
HOLLENBECK, Christopher M.; JOHNSTON, Ian A. Genomic tools and selective breeding in molluscs. Frontiers in Genetics, [S. l.], v. 9, n. JUL, p. 1–15, 2018. https://doi.org/10.3389/fgene.2018.00253
HONG-GELLER, E.; MICHEVA-VITEVA, SN. Functional Gene Discovery Using RNA Interference-Based Genomic Screens to Combat Pathogen Infection. Current Drug Discovery Technologies, [S. l.], v. 7, n. 2, p. 86–94, 2010. https://doi.org/10.2174/157016310793180657
HORGAN, Finbarr G.; STUART, Alexander M.; KUDAVIDANAGE, Enoka P. Impact of invasive apple snails on the functioning and services of natural andmanaged wetlands. Acta Oecologica, [S. l.], v. 54, p. 90–100, 2014. https://doi.org/10.1016/j.actao.2012.10.002
HUANG, Jianfang; LUO, Xuan; HUANG, Miaoqin; LIU, Guangmou; YOU, Weiwei; KE, Caihuan. Identification and characteristics of muscle growth-related microRNA in the Pacific abalone, Haliotis discus hannai. BMC Genomics, [S. l.], v. 19, n. 1, p. 1–11, 2018. https://doi.org/10.1186/s12864-018-5347-9
HUANG, Songqian et al. Identification and characterization of microRNAs and their predicted functions in biomineralization in the pearl oyster (Pinctada fucata). Biology, [S. l.], v. 8, n. 2, 2019. https://doi.org/10.3390/biology8020047
HUANG, Songqian; YOSHITAKE, Kazutoshi; ASADUZZAMAN, Md; KINOSHITA, Shigeharu; WATABE, Shugo; ASAKAWA, Shuichi. Discovery and functional understanding of MiRNAs in molluscs: a genome-wide profiling approach. RNA Biology, [S. l.], n. January, 2021. https://doi.org/10.1080/15476286.2020.1867798
HUNTZINGER, Eric; IZAURRALDE, Elisa. Gene silencing by microRNAs: Contributions of translational repression and mRNA decay. Nature Reviews Genetics, [S. l.], v. 12, n. 2, p. 99–110, 2011. https://doi.org/10.1038/nrg2936
IORIO, Marilena V.; CROCE, Carlo M. MicroRNAs in cancer: Small molecules with a huge impact. Journal of Clinical Oncology, [S. l.], v. 27, n. 34, p. 5848–5856, 2009. https://doi.org/10.1200/JCO.2009.24.0317
IP, Jack C. H. et al. AmpuBase: A transcriptome database for eight species of apple snails (Gastropoda: Ampullariidae). BMC Genomics, [S. l.], v. 19, n. 1, p. 1–9, 2018. https://doi.org/10.1186/s12864-018-4553-9
IPSARO, Jonathan J.; HAASE, Astrid D.; KNOTT, Simon R.; JOSHUA-TOR, Leemor; HANNON, Gregory J. The structural biochemistry of Zucchini implicates it as a nuclease in piRNA biogenesis. Nature, [S. l.], v. 491, n. 7423, p. 279–282, 2012. https://doi.org/10.1038/nature11502
JO, Myung Hyun; SHIN, Soochul; JUNG, Seung Ryoung; KIM, Eunji; SONG, Ji Joon; HOHNG, Sungchul. Human Argonaute 2 Has Diverse Reaction Pathways on Target RNAs. Molecular Cell, [S. l.], v. 59, n. 1, p. 117–124, 2015. https://doi.org/10.1016/j.molcel.2015.04.027
JOKAT, Wilfried; BOEBEL, Tobias; KÖNIG, Matthias; MEYER, Uwe. Timing and geometry of early Gondwana breakup. Journal of Geophysical Research: Solid Earth, [S. l.], v. 108, n. B9, 2003. https://doi.org/10.1029/2002JB001802
JONAS, Stefanie; IZAURRALDE, Elisa. Towards a molecular understanding of microRNA-mediated gene silencing. Nature Reviews Genetics, [S. l.], v. 16, n. 7, p. 421–433, 2015. https://doi.org/10.1038/nrg3965
JOSHI, Ravindra C. Managing invasive alien mollusc species in rice. International Rice Research News, [S. l.], v. 30, n. 2, p. 5–13, 2005. https://doi.org/10.1016/j.ecolecon.2008.09.006
KADENER, Sebastian et al. A role for microRNAs in the Drosophila circadian clock. Genes and Development, [S. l.], v. 23, n. 18, p. 2179–2191, 2009. https://doi.org/10.1101/gad.1819509
KAWAMATA, Tomoko; TOMARI, Yukihide. Making RISC. Trends in Biochemical Sciences, [S. l.], v. 35, n. 7, p. 368–376, 2010. https://doi.org/10.1016/j.tibs.2010.03.009
KAWANO, Yoshihiko; IWATA, Seiko; KAWADA, Jun Ichi; GOTOH, Kensei; SUZUKI, Michio; TORII, Yuka; KOJIMA, Seiji; KIMURA, Hiroshi; ITO, Yoshinori. Plasma viral MicroRNA profiles reveal potential biomarkers for chronic active epstein-barr virus infection. Journal of Infectious Diseases, [S. l.], v. 208, n. 5, p. 771–779, 2013. https://doi.org/10.1093/infdis/jit222
KHVOROVA, Anastasia; REYNOLDS, Angela; JAYASENA, Sumedha D. Functional siRNAs and miRNAs exhibit strand bias. Cell, [S. l.], v. 115, n. 2, p. 209–216, 2003. https://doi.org/10.1016/S0092-8674(03)00801-8
KIM, V. Narry. MicroRNA biogenesis: Coordinated cropping and dicing. Nature Reviews Molecular Cell Biology, [S. l.], v. 6, n. 5, p. 376–385, 2005. https://doi.org/10.1038/nrm1644
KIM, Young Kook; KIM, V. Narry. Processing of intronic microRNAs. EMBO Journal, [S. l.], v. 26, n. 3, p. 775–783, 2007. https://doi.org/10.1038/sj.emboj.7601512
KRUATRACHUE, M.; SUMRITDEE, C.; POKETHITIYOOK, P.; SINGHAKAEW, S. Histopathological effects of contaminated sediments on golden apple snail (Pomacea canaliculata, Lamarck 1822). Bulletin of Environmental Contamination and Toxicology, [S. l.], v. 86, n. 6, p. 610–614, 2011. https://doi.org/10.1007/s00128-011-0265-4
LAGOS-QUINTANA, M.; RAUHUT, R.; LENDECKEL, W.; TUSCHL, T. Identification of novel genes coding for small expressed RNAs. Science, [S. l.], v. 294, n. 5543, p. 853–858, 2001. https://doi.org/10.1126/science.1064921
LAM, Victoria; TOKUSUMI, Tsuyoshi; TOKUSUMI, Yumiko; SCHULZ, Robert A. bantam miRNA is important for Drosophila blood cell homeostasis and a regulator of proliferation in the hematopoietic progenitor niche. Biochemical and Biophysical Research Communications, [S. l.], v. 453, n. 3, p. 467–472, 2014. https://doi.org/10.1016/j.bbrc.2014.09.109
LARKIN, M. A. et al. Clustal W and Clustal X version 2.0. Bioinformatics, [S. l.], v. 23, n. 21, p. 2947–2948, 2007. . https://doi.org/10.1093/bioinformatics/btm404
LAWRIE, Charles H. MicroRNA expression in. Expert Opinion, [S. l.], v. 7, n. 9, p. 1363–1374, 2007. https://doi.org/10.1517/14712598.7.9.1363
LAWRIE, Charles H. et al. Detection of elevated levels of tumour-associated microRNAs in serum of patients with diffuse large B-cell lymphoma. British Journal of Haematology, [S. l.], v. 141, n. 5, p. 672–675, 2008. https://doi.org/10.1111/j.1365-2141.2008.07077.x
LEE, Rosalind C.; FEINBAUM, Rhonda L.; AMBROS, Victor. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell, [S. l.], v. 75, n. 5, p. 843–854, 1993. https://doi.org/10.1016/0092-8674(93)90529-Y
LEE, Y. MicroRNA maturation: stepwise processing and subcellular localization. The EMBO Journal, [S. l.], v. 21, n. 17, p. 4663–4670, 2002. https://doi.org/10.1093/emboj/cdf476
LEONARDO, Trevor R.; SCHULTHEISZ, Heather L.; LORING, Jeanne F.; LAURENT, Louise C. The functions of microRNAs in pluripotency and reprogramming. Nature Cell Biology, [S. l.], v. 14, n. 11, p. 1114–1121, 2012. https://doi.org/10.1038/ncb2613
LEWIS, Benjamin P.; BURGE, Christopher B.; BARTEL, David P. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets. Cell, [S. l.], v. 120, n. 1, p. 15–20, 2005. https://doi.org/10.1016/j.cell.2004.12.035
LEWIS, BP; SHIH, I. U.; JONES-RHOADES, MW; BARTEL, Davi. ..; BURGE, CB. Prediction of mammalian microRNA targets. Cell, [S. l.], v. 115, p. 787–798, 2003. https://doi.org/10.1016/S0092-8674(03)01018-3
LI, Li; XU, Jianzhen; YANG, Deyin; TAN, Xiaorong; WANG, Hongfei. Computational approaches for microRNA studies: A review. Mammalian Genome, [S. l.], v. 21, n. 1–2, p. 1–12, 2010. https://doi.org/10.1007/s00335-009-9241-2
LI, Ning; ZHOU, Heng; TANG, Qizhu. miR-133: A suppressor of cardiac remodeling? Frontiers in Pharmacology, [S. l.], v. 9, n. AUG, 2018. https://doi.org/10.3389/fphar.2018.00903
LI, Song; MEAD, Edward A.; LIANG, Shaohui; TU, Zhijian. Direct sequencing and expression analysis of a large number of miRNAs in Aedes aegypti and a multi-species survey of novel mosquito miRNAs. BMC Genomics, [S. l.], v. 10, p. 1–17, 2009. https://doi.org/10.1186/1471-2164-10-581
LI, Ying; PADGETT, Richard W. Bantam is required for optic lobe development and glial cell proliferation. PLoS ONE, [S. l.], v. 7, n. 3, p. 1–12, 2012. https://doi.org/10.1371/journal.pone.0032910
LINDO, John F. et al. Enzootic Angiostrongylus cantonensis in rats and snails after an outbreak of human eosinophilic meningitis, Jamaica. Emerging Infectious Diseases, [S. l.], v. 8, n. 3, p. 324–326, 2002. https://doi.org/10.3201/eid0803.010316
LIU, Nghui et al. The genome of the golden apple snail Pomacea canaliculata provides insight into stress tolerance and invasive adaptation. GigaScience, [S. l.], v. 7, n. 9, p. 1–13, 2018. https://doi.org/10.1093/gigascience/giy101
LIU, Wenquan; HUANG, Huicong; XING, Cuicui; LI, Chunxiang; TAN, Feng; LIANG, Shaohui. Identification and characterization of the expression profile of microRNAs in Anopheles anthropophagus. Parasites and Vectors, [S. l.], v. 7, n. 1, p. 1–8, 2014. https://doi.org/10.1186/1756-3305-7-159
LIU, Xian Shuang et al. MicroRNA profiling in subventricular zone after stroke: MiR-124a regulates proliferation of neural progenitor cells through notch signaling pathway. PLoS ONE, [S. l.], v. 6, n. 8, p. 1–11, 2011. https://doi.org/10.1371/journal.pone.0023461
LUESSI, Felix; SOLLORS, Janina; TORZEWSKI, Michael; MÜLLER, Harald D.; SIEGEL, Ekkehard; BLUM, Johannes; SOMMER, Clemens; VOGT, Thomas; THÖMKE, Frank. Eosinophilic meningitis due to angiostrongylus cantonensis in Germany. Journal of Travel Medicine, [S. l.], v. 16, n. 4, p. 292–294, 2009. https://doi.org/10.1111/j.1708- 8305.2009.00337.x
LUQUE, Gloria M.; BELLARD, Céline; BERTELSMEIER, Cleo; BONNAUD, Elsa; GENOVESI, Piero; SIMBERLOFF, Daniel; COURCHAMP, Franck. The 100th of the world’s worst invasive alien species. Biological Invasions, [S. l.], v. 16, n. 5, p. 981–985, 2014. https://doi.org/10.1007/s10530-013-0561-5
MACRAE, Ian J.; ZHOU, Kaihong; DOUDNA, Jennifer A. Structural determinants of RNA recognition and cleavage by Dicer. Nature Structural and Molecular Biology, [S. l.], v. 14, n. 10, p. 934–940, 2007. https://doi.org/10.1038/nsmb1293
MACRAE, Ian J.; ZHOU, Kaihong; LI, Fei; REPIC, Adrian; BROOKS, Angela N.; CANDE, W. Zacheus; ADAMS, Paul D.; DOUDNA, Jennifer A. Structural basis for doublestranded RNA processing by Dicer. Science, [S. l.], v. 311, n. 5758, p. 195–198, 2006. https://doi.org/10.1126/science.1121638
MARTÍN-GÓMEZ, Laura; VILLALBA, Antonio; KERKHOVEN, Ron H.; ABOLLO, Elvira. Role of microRNAs in the immunity process of the flat oyster Ostrea edulis against bonamiosis. Infection, Genetics and Evolution, [S. l.], v. 27, p. 40–50, 2014. https://doi.org/10.1016/j.meegid.2014.06.026
MATSUKURA, Keiichiro; IZUMI, Yohei; YOSHIDA, Kazuhiro; WADA, Takashi. Cold tolerance of invasive freshwater snails, Pomacea canaliculata, P. maculata, and their hybrids helps explain their different distributions. Freshwater Biology, [S. l.], v. 61, n. 1, p. 80–87, 2016. https://doi.org/10.1111/fwb.12681
MATSUKURA, Keiichiro; OKUDA, Mitsuru; CAZZANIGA, Néstor Jorge; WADA, Takashi. Genetic exchange between two freshwater apple snails, Pomacea canaliculata and Pomacea maculata invading East and Southeast Asia. Biological Invasions, [S. l.], v. 15, n. 9, p. 2039–2048, 2013. https://doi.org/10.1007/s10530-013-0431-1
MATSUKURA, Keiichiro; TSUMUKI, Hisaaki; IZUMI, Yohei; WADA, Takashi. Physiological response to low temperature in the freshwater apple snail, Pomacea canaliculata (Gastropoda: Ampullariidae). Journal of Experimental Biology, [S. l.], v. 212, n. 16, p. 2558–2563, 2009. https://doi.org/10.1242/jeb.031500
MEIJER, Hedda A.; SMITH, Ewan M.; BUSHELL, Martin. Regulation of miRNA strand selection: Follow the leader? Biochemical Society Transactions, [S. l.], v. 42, n. 4, p. 1135– 1140, 2014. https://doi.org/10.1042/BST20140142
MONTE, Tainá Carneiro de Castro et Al. Estudo Filogenético, Biológico e Morfológico de isolados de Angiostrongylus cantonensis provenientes de diferentes áreas geográficas do Brasil. 2014. [S. l.], 2014. Disponível em: https://www.arca.fiocruz.br/bitstream/icict/12867/1/taina_monte_ioc_mest_2014.pdf
MUECK, Kristy; DEATON, Lewis E.; LEE, Andrea; GUILBEAUX, Trey. Physiology of the apple snail pomacea maculata: Aestivation and overland dispersal. Biological Bulletin, [S. l.], v. 235, n. 1, p. 43–51, 2018. https://doi.org/10.1086/698817
NG, T. H.; TAN, S. K.; YEO, D. C. J. South American apple snails, Pomacea spp. (Ampullariidae), in Singapore. Biology and Management of Invasive Apple Snails, [S. l.], n. October, p. 221–239, 2017. Disponível em: https://www.cabi.org/isc/FullTextPDF/2017/20173354471.pdf
NIELSEN, Finn Cilius; LUND, Anders H. MicroRNA-10a Binds the 5′UTR of Ribosomal Protein mRNAs and Enhances Their Translation. Molecular Cell, [S. l.], v. 30, n. 4, p. 460–471, 2008. https://doi.org/10.1016/j.molcel.2008.05.001
NISHIMASU, Hiroshi et al. Structure and function of Zucchini endoribonuclease in piRNA biogenesis. Nature, [S. l.], v. 491, n. 7423, p. 284–287, 2012. https://doi.org/10.1038/nature11509
NOVARINO, Aadrean Aadrean. Record of Small-Clawed Otters (Aonyx cinereus) Foraging on an Invasive Pest Species, Golden Apple Snails (Pomacea canaliculata) in a West Sumatra Rice Field. IUCN/SCC Otter Specialist Group Bulletin, [S. l.], v. 28, n. 2, p. 34– 38, 2011. Disponível em: https://www.iucnosgbull.org/Volume28/Aadrean_et_al_2011.html
OETJEN, E. MicroRNAs in β-Cell Biology, Insulin Resistance, Diabetes and Its Complications. Yearbook of Endocrinology, [S. l.], v. 2012, p. 11–12, 2012. https://doi.org/10.1016/j.yend.2012.05.011
OKADA, Chiman; YAMASHITA, Eiki; LEE, Soo Jae; SHIBATA, Satoshi; KATAHIRA, Jun; NAKAGAWA, Atsushi; YONEDA, Yoshihiro; TSUKIHARA, Tomitake. A high-Resolution structure of the pre-microrna nuclear export machinery. Science, [S. l.], v. 326, n. 5957, p. 1275–1279, 2009. https://doi.org/10.1126/science.1178705
OLIVEIRA, Dorlam’s da Silva; FRANÇA, Clícia Rosane; PAIXÃO, Dionísia Fernanda; ARAGÃO, Renato juvino; FILHO, Adalberto Alves Pereira; ROSA, Ivone Garros. ESQUISTOSSOMOSE: CONTROLE BIOLÓGICO DA Biomphalaria glabrata POR Pomacea canaliculata. In: 64a REUNIÃO ANUAL DA SBPC 2012, São Luis, Brazil. Anais […]. São Luis, Brazil p. 7–8. Disponível em: http://www.sbpcnet.org.br/livro/64ra/resumos/resumos/1516.htm.
OTTAVIANI, Enzo; ACCORSI, Alice; RIGILLO, Giovanna; MALAGOLI, Davide; BLOM, Joan M. C.; TASCEDDA, Fabio. Epigenetic modification in neurons of the mollusc Pomacea canaliculata after immune challenge. Brain Research, [S. l.], v. 1537, p. 18–26, 2013. https://doi.org/10.1016/j.brainres.2013.09.009
OWENS, Leigh; MALHAM, Shelagh. Review of the RNA interference pathway in molluscs including some possibilities for use in bivalves in aquaculture. Journal of Marine Science and Engineering, [S. l.], v. 3, n. 1, p. 87–99, 2015. https://doi.org/10.3390/jmse3010087
PARK, Jong Eun; HEO, Inha; TIAN, Yuan; SIMANSHU, Dhirendra K.; CHANG, Hyeshik; JEE, David; PATEL, Dinshaw J.; KIM, V. Narry. Dicer recognizes the 5′ end of RNA for efficient and accurate processing. Nature, [S. l.], v. 475, n. 7355, p. 201–205, 2011. https://doi.org/10.1038/nature10198
PICONE, Barbara; RHODE, Clint; ROODT-WILDING, Rouvay. Identification and characterization of miRNAs transcriptome in the South African abalone, Haliotis midae. Marine Genomics, [S. l.], v. 31, p. 9–12, 2017. DOI: 10.1016/j.margen.2016.10.005. https://doi.org/10.1016/j.margen.2016.10.005
PIMENTEL, David; ZUNIGA, Rodolfo; MORRISON, Doug. Update on the environmental and economic costs associated with alien-invasive species in the United States. Ecological Economics, [S. l.], v. 52, n. 3 SPEC. ISS., p. 273–288, 2005. https://doi.org/10.1016/j.ecolecon.2004.10.002
PLOUGH, Louis V.; HEDGECOCK, Dennis. Quantitative trait locus analysis of stage- specific inbreeding depression in the pacific oyster Crassostrea gigas. Genetics, [S. l.], v. 189, n. 4, p. 1473–1486, 2016. https://doi.org/10.1534/genetics.111.131854
QUEIROZ, Fabio Ribeiro; LG, Portilho; WJ, Jeremias; AL., Et. Deep sequencing of small RNAs reveals the repertoire of miRNAs and piRNAs in Biomphalaria glabrata. Memorias do Instituto Oswaldo Cruz, [S. l.], v. 115, n. e190498, 2020. https://doi.org/10.1590/0074-02760190498
QUEIROZ, Fábio Ribeiro; SILVA, Luciana Maria; DE JESUS JEREMIAS, Wander; HIDEO BABÁ, Élio; CALDEIRA, Roberta Lima; COELHO, Paulo Marcos Zech; DE SOUZA GOMES, Matheus. Differential expression of small RNA pathway genes associated with the Biomphalaria glabrata/Schistosoma mansoni interaction. PLoS ONE, [S. l.], v. 12, n. 7, p. 1– 20, 2017. https://doi.org/10.1371/journal.pone.0181483
RACHAGANI, Satyanarayana; KUMAR, Sushil; BATRA, Surinder K. MicroRNA in pancreatic cancer: Pathological, diagnostic and therapeutic implications. Cancer Letters, [S. l.], v. 292, n. 1, p. 8–16, 2010. https://doi.org/10.1016/j.canlet.2009.11.010.
RAKOCZY, J. et al. MicroRNAs-140-5p/140-3p Modulate Leydig Cell Numbers in the Developing Mouse Testis. Biology of Reproduction, [S. l.], v. 88, n. 6, p. 143–143, 2013. https://doi.org/10.1095/biolreprod.113.107607
RENAUT, Sébastien; GUERRA, Davide; HOEH, Walter R.; STEWART, Donald T.; BOGAN, Arthur E.; GHISELLI, Fabrizio; MILANI, Liliana; PASSAMONTI, Marco; BRETON, Sophie. Genome survey of the freshwater mussel Venustaconcha ellipsiformis (Bivalvia: Unionida) using a hybrid de novo assembly approach. Genome Biology and Evolution, [S. l.], v. 10, n. 7, p. 1637–1646, 2018. b. https://doi.org/10.1093/gbe/evy117
RUBY, J. Graham; JAN, Calvin H.; BARTEL, David P. Intronic microRNA precursors that bypass Drosha processing. Nature, [S. l.], v. 448, n. 7149, p. 83–86, 2007. https://doi.org/10.1038/nature05983
SAITOU, N.; NEI, Masatoshi. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular biology and evolution, [S. l.], v. 4, n. 4, p. 406- 25, 1987. a. https://doi.org/10.1093/oxfordjournals.molbev.a040454
SANGER, F.; COULSON, A. R. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. Journal of Molecular Biology, [S. l.], v. 94, n. 3, p. 441–448, 1975. https://doi.org/10.1016/0022-2836(75)90213-2
SASIDHARAN, Vidyanand; LU, Yi Chien; BANSAL, Dhiru; DASARI, Pranavi; PODUVAL, Deepak; SESHASAYEE, Aswin; RESCH, Alissa M.; GRAVELEY, Brenton R.; PALAKODETI, Dasaradhi. Identification of neoblast- And regeneration-specific miRNAs in the planarian Schmidtea mediterranea. Rna, [S. l.], v. 19, n. 10, p. 1394–1404, 2013. https://doi.org/10.1261/rna.038653.113
SCHELL, Tilman et al. An Annotated Draft Genome for Radix auricularia (Gastropoda, Mollusca). Genome Biology and Evolution, [S. l.], v. 9, n. 3, p. 585–592, 2017. https://doi.org/10.1093/gbe/evx032
SCHULTZ, Jonathan H.; ADEMA, Coen M. Comparative immunogenomics of molluscs. Developmental and Comparative Immunology, [S. l.], v. 75, p. 3–15, 2017. https://doi.org/10.1016/j.dci.2017.03.013.
SEITZ, Hervé; TUSHIR, Jogender S.; ZAMORE, Phillip D. A 5′-uridine amplifies miRNA/miRNA* asymmetry in Drosophila by promoting RNA-induced silencing complex formation. Silence, [S. l.], v. 2, n. 1, p. 1–10, 2011. https://doi.org/10.1186/1758-907X-2-4
SHENNAN LU ET AL. CDD: NCBI’s conserved domain database. 2020. Disponível em: https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi. Acesso em: 7 jun. 2020.
SIGWART, Julia D.; LINDBERG, David R. Consensus and confusion in molluscan trees: Evaluating morphological and molecular phylogenies. Systematic Biology, [S. l.], v. 64, n. 3, p. 384–395, 2015. https://doi.org/10.1093/sysbio/syu105
SILVANA THIENGO. Sistemática e biogeografia família pilidae connoly: gênero pomacea. [S. l.], 1993. Disponível em: http://books.scielo.org/id/npy7z/pdf/barbosa- 9788575414019-05.pdf
SILVANA THIENGO. Sistemática e Biogeografia. Ecologia, [S. l.], p. 1–10, 1995. Disponível em: http://books.scielo.org/id/npy7z/pdf/barbosa-9788575414019-04.pdf
SIMAKOV, Oleg et al. Insights into bilaterian evolution from three spiralian genomes. Nature, [S. l.], v. 493, n. 7433, p. 526–531, 2013. https://doi.org/10.1038/nature11696
SIOMI, Mikiko C.; SATO, Kaoru; PEZIC, Dubravka; ARAVIN, Alexei A. PIWI- interacting small RNAs: The vanguard of genome defence. Nature Reviews Molecular Cell Biology, [S. l.], v. 12, n. 4, p. 246–258, 2011. https://doi.org/10.1038/nrm3089
SONG, Hao; QI, Lu; ZHANG, Tao; WANG, Hai Yan. Understanding microRNA regulation involved in the metamorphosis of the veined rapa whelk (Rapana venosa). G3: Genes, Genomes, Genetics, [S. l.], v. 7, n. 12, p. 3999–4008, 2017. https://doi.org/10.1534/g3.117.300210
SONG, Ji Joon; SMITH, Stephanie K.; HANNON, Gregory J.; JOSHUA-TOR, Leemor. Crystal structure of argonaute and its implications for RISC slicer activity. Science, [S. l.], v. 305, n. 5689, p. 1434–1437, 2004. https://doi.org/10.1126/science.1102514
SUN, Jin et al. Signatures of divergence, invasiveness, and terrestrialization revealed by four apple snail genomes. Molecular Biology and Evolution, [S. l.], v. 36, n. 7, p. 1507–1520, 2019. https://doi.org/10.1093/molbev/msz084
SUN, Jin; ZHANG, Huoming; WANG, Hao; HERAS, Horacio; DREON, Marcos S.; ITUARTE, Santiago; RAVASI, Timothy; QIAN, Pei Yuan; QIU, Jian Wen. First proteome of the egg perivitelline fluid of a freshwater gastropod with aerial oviposition. Journal of Proteome Research, [S. l.], v. 11, n. 8, p. 4240–4248, 2012. https://doi.org/10.1021/pr3003613
SWARTS, Daan C.; MAKAROVA, Kira; WANG, Yanli; NAKANISHI, Kotaro; KETTING, René F.; KOONIN, Eugene V.; PATEL, Dinshaw J.; VAN DER OOST, John. The evolutionary journey of Argonaute proteins. Nature Structural and Molecular Biology, [S. l.], v. 21, n. 9, p. 743–753, 2014. https://doi.org/10.1038/nsmb.2879
TAKANE, Kahori; FUJISHIMA, Kosuke; WATANABE, Yuka; SATO, Asako; SAITO, Nobuto; TOMITA, Masaru; KANAI, Akio. Computational prediction and experimental validation of evolutionarily conserved microRNA target genes in bilaterian animals. BMC Genomics, [S. l.], v. 11, n. 1, 2010. https://doi.org/10.1186/1471-2164-11-101
TAKAYA, Tomohide et al. MicroRNA-1 and microRNA-133 in spontaneous myocardial differentiation of mouse embryonic stem cells. Circulation Journal, [S. l.], v. 73, n. 8, p. 1492– 1497, 2009. https://doi.org/10.1253/circj.CJ-08-1032
TAKEUCHI, Takeshi et al. Draft genome of the pearl oyster Pinctada fucata: A platform for understanding bivalve biology. DNA Research, [S. l.], v. 19, n. 2, p. 117–130, 2012. https://doi.org/10.1093/dnares/dss005
TAMURA, Koichiro; DUDLEY, Joel; NEI, Masatoshi; KUMAR, Sudhir. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Molecular Biology and Evolution, [S. l.], v. 24, n. 8, p. 1596–1599, 2007. https://doi.org/10.1093/molbev/msm092
TANZER, Andrea; STADLER, Peter F. Molecular evolution of a microRNA cluster. Journal of Molecular Biology, [S. l.], v. 339, n. 2, p. 327–335, 2004. https://doi.org/10.1016/j.jmb.2004.03.065
THOMPSON, Julie D.; GIBSON, Toby. J.; HIGGINS, Des G. Multiple Sequence Alignment Using ClustalW and ClustalX. Current Protocols in Bioinformatics, [S. l.], v. 00, n. 1, p. 2.3.1-2.3.22, 2002. https://doi.org/10.1002/0471250953.bi0203s00
TSAI, Hung Chin et al. Eosinophilic meningitis caused by Angiostrongylus cantonensis: Report of 17 cases. American Journal of Medicine, [S. l.], v. 111, n. 2, p. 109–114, 2001. https://doi.org/10.1016/S0002-9343(01)00766-5
TUDOR, S. et al. Cellular and Kaposi’s sarcoma-associated herpes virus microRNAs in sepsis and surgical trauma. Cell Death and Disease, [S. l.], v. 5, n. 12, 2014. https://doi.org/10.1038/cddis.2014.515
ULIANO, Marcela; DONDERO, Francesco; DAN OTTO, Thomas; COSTA, Igor; LIMA, Nicholas Costa Barroso; AMERICO, Juliana Alves; MAZZONI, Camila Junqueira; PROSDOCIMI, Francisco; REBELO, Mauro de Freitas. A hybrid-hierarchical genome assembly strategy to sequence the invasive golden mussel, Limnoperna fortunei. [s.l: s.n.]. v. 7. https://doi.org/10.1093/gigascience/gix128
VASUDEVAN, Shobha. Posttranscriptional Upregulation by MicroRNAs. Wiley Interdisciplinary Reviews: RNA, [S. l.], v. 3, n. 3, p. 311–330, 2012. https://doi.org/10.1002/wrna.121
VASUDEVAN, Shobha; STEITZ, Joan A. AU-Rich-Element-Mediated Upregulation of Translation by FXR1 and Argonaute 2. Cell, [S. l.], v. 128, n. 6, p. 1105–1118, 2007. https://doi.org/10.1016/j.cell.2007.01.038
VEENSTRA, Jan A. Neurohormones and neuropeptides encoded by the genome of Lottia gigantea, with reference to other mollusks and insects. General and Comparative Endocrinology, [S. l.], v. 167, n. 1, p. 86–103, 2010. https://doi.org/10.1016/j.ygcen.2010.02.010
VOOLSTRA, Christian R.; GIGA COMMUNITY OF SCIENTISTS (COS); WÖRHEIDE, Gert; LOPEZ, Jose V. Corrigendum to: Advancing genomics through the Global Invertebrate Genomics Alliance (GIGA). Invertebrate Systematics, [S. l.], v. 31, n. 2, p. 231, 2017. https://doi.org/10.1071/IS16059_CO
WALKER, Sarah E.; SPENCER, Gaynor E.; NECAKOV, Aleksandar; CARLONE, Robert L. Identification and characterization of microRNAs during retinoic acid-induced regeneration of a molluscan central nervous system. International Journal of Molecular Sciences, [S. l.], v. 19, n. 9, 2018. https://doi.org/10.3390/ijms19092741
WANG et al. Scallop genome provides insights into evolution of bilaterian karyotype and development. Nature Ecology and Evolution, [S. l.], v. 1, n. 5, p. 1–12, 2017. https://doi.org/10.1038/s41559-017-0120
WANG, Fengjun; ZHENG, Zhiguo; GUO, Jiangfeng; DING, Xianfeng. Correlation and quantitation of microRNA aberrant expression in tissues and sera from patients with breast tumor. Gynecologic Oncology, [S. l.], v. 119, n. 3, p. 586–593, 2010. https://doi.org/10.1016/j.ygyno.2010.07.021
WANG, Q. P.; WU, Z. D.; WEI, J.; OWEN, R. L.; LUN, Z. R. Human angiostrongylus cantonensis: An update. European Journal of Clinical Microbiology and Infectious Diseases, [S. l.], v. 31, n. 4, p. 389–395, 2012. https://doi.org/10.1007/s10096-011-1328-5
WANG, Qiao Ping; LAI, De Hua; ZHU, Xing Quan; CHEN, Xiao Guang; LUN, Zhao Rong. Human angiostrongyliasis. The Lancet Infectious Diseases, [S. l.], v. 8, n. 10, p. 621– 630, 2008. https://doi.org/10.1016/S1473-3099(08)70229-9
WANG, Xiaolin; SHI, Jialun; NIU, Zhigao; WANG, Jianwu; ZHANG, Wenping. MiR- 216a-3p regulates the proliferation, apoptosis, migration, and invasion of lung cancer cells via targeting COPB2. Bioscience, Biotechnology and Biochemistry, [S. l.], v. 00, n. 00, p. 2014– 2027, 2020. https://doi.org/10.1080/09168451.2020.1783197
WANG, Xiaowo; ZHANG, Jing; LI, Fei; GU, Jin; HE, Tao; ZHANG, Xuegong; LI, Yanda. MicroRNA identification based on sequence and structure alignment. Bioinformatics, [S. l.], v. 21, n. 18, p. 3610–3614, 2005. https://doi.org/10.1093/bioinformatics/bti562
WEI, P.; P, He; X, Zhang; ET. Identification and characterization of microRNAs in the gonads of Crassostrea hongkongensis using high-throughput sequencing. Comp Biochem Physiol Part D Genomics Proteomics., [S. l.], v. 31, 2019. https://doi.org/10.1016/j.cbd.2019.100606
XIE, Mingyi; LI, Mingfeng; VILBORG, Anna; LEE, Nara; SHU, Mei Di; YARTSEVA, Valeria; ŠESTAN, Nenad; STEITZ, Joan A. Mammalian 5′-capped microRNA precursors that generate a single microRNA. Cell, [S. l.], v. 155, n. 7, p. 1568–1580, 2013. https://doi.org/10.1016/j.cell.2013.11.027
XUE, Chenghai; LI, Fei; HE, Tao; LIU, Guo Ping; LI, Yanda; ZHANG, Xuegong. Classification of real and pseudo microRNA precursors using local structure-sequence features and support vector machine. BMC Bioinformatics, [S. l.], v. 6, p. 1–7, 2005. https://doi.org/10.1186/1471-2105-6-310
YANG, Shiuan et al. Conserved vertebrate mir-451 provides a platform for Dicer- independent, Ago2-mediated microRNA biogenesis. Proceedings of the National Academy of Sciences of the United States of America, [S. l.], v. 107, n. 34, p. 15163–15168, 2010. https://doi.org/10.1073/pnas.1006432107
YANG, Wei; STEITZ, Thomas A. Recombining the structures of HIV integrase, RuvC and RNase H. Structure, [S. l.], v. 3, n. 2, p. 131–134, 1995. https://doi.org/10.1016/S0969- 2126(01)00142-3
YODA, Mayuko; KAWAMATA, Tomoko; PAROO, Zain; YE, Xuecheng; IWASAKI, Shintaro; LIU, Qinghua; TOMARI, Yukihide. ATP-dependent human RISC assembly pathways. Nature Structural and Molecular Biology, [S. l.], v. 17, n. 1, p. 17–24, 2010. https://doi.org/10.1038/nsmb.1733
YU, Deliang; WU, Huifeng; PENG, Xiao; JI, Chenglong; ZHANG, Xiaoying; SONG, Jun; QU, Junle. Profiling of microRNAs and mRNAs in marine mussel Mytilus galloprovincialis. Comparative Biochemistry and Physiology Part – C: Toxicology and Pharmacology, [S. l.], v. 230, n. November 2019, p. 108697, 2020. https://doi.org/10.1016/j.cbpc.2019.108697
YUSA, Yoichi; WADA, Takashi; TAKAHASHI, Satoshi. Effects of dormant duration, body size, self-burial and water condition on the long-term survival of the apple snail, Pomacea canaliculata (Gastropoda: Ampullariidae). Applied Entomology and Zoology, [S. l.], v. 41, n. 4, p. 627–632, 2006. https://doi.org/10.1303/aez.2006.627
YUVA-AYDEMIR, Yeliz; SIMKIN, Alfred; GASCON, Eduardo; GAO, Fen Biao. MicroRNA-9: Functional evolution of a conserved small regulatory RNA. RNA Biology, [S. l.], v. 8, n. 4, p. 557–564, 2011. https://doi.org/10.4161/rna.8.4.16019
ZHANG, Baohong; STELLWAG, Edmund J.; PAN, Xiaoping. Large-scale genome analysis reveals unique features of microRNAs. Gene, [S. l.], v. 443, n. 1–2, p. 100–109, 2009. https://doi.org/10.1016/j.gene.2009.04.027
ZHANG, Haidi; KOLB, Fabrice A.; JASKIEWICZ, Lukasz; WESTHOF, Eric; FILIPOWICZ, Witold. Single Processing Center Models for Human Dicer and Bacterial RNase III. Cell, [S. l.], v. 118, n. 1, p. 57–68, 2004. https://doi.org/10.1016/j.cell.2004.06.017
ZHANG, Wenyu; CHEN, Jiajia; YANG, Yang; TANG, Yifei; SHANG, Jing; SHEN, Bairong. A practical comparison of De Novo genome assembly software tools for next- generation sequencing technologies. PLoS ONE, [S. l.], v. 6, n. 3, 2011. https://doi.org/10.1371/journal.pone.0017915
ZHANG, Yan Feng; ZHANG, Rui; SU, Bing. Diversity and evolution of MicroRNA gene clusters. Science in China, Series C: Life Sciences, [S. l.], v. 52, n. 3, p. 261–266, 2009. https://doi.org/10.1007/s11427-009-0032-5
ZHANG, Yan Qiong; CHEN, Dong Liang; TIAN, Hai Feng; ZHANG, Bao Hong; WEN, Jian Fan. Genome-wide computational identification of microRNAs and their targets in the deep-branching eukaryote Giardia lamblia. Computational Biology and Chemistry, [S. l.], v. 33, n. 5, p. 391–396, 2009. https://doi.org/10.1016/j.compbiolchem.2009.07.013
ZHAO, Xuelin; YU, Hong; KONG, Lingfeng; LIU, Shikai; LI, Qi. High throughput sequencing of small RNAs transcriptomes in two Crassostrea oysters identifies microRNAs involved in osmotic stress response. Scientific Reports, [S. l.], v. 6, n. March, p. 1–11, 2016. https://doi.org/10.1038/srep22687
ZHOU, Meng et al. In silico detection and characteristics of novel microRNA genes in the Equus caballus genome using an integrated ab initio and comparative genomic approach. Genomics, [S. l.], v. 94, n. 2, p. 125–131, 2009. https://doi.org/10.1016/j.ygeno.2009.04.006
ZHU, Lihui; LIU, Jinming; CHENG, Guofeng. Role of microRNAs in schistosomes and schistosomiasis. Frontiers in Cellular and Infection Microbiology, [S. l.], v. 4, n. NOV, p. 1–5, 2014. https://doi.org/10.3389/fcimb.2014.00165
