ANALYSIS OF THE MOST PREVALENT BACTERIAL PROFILE IN RESPIRATORY INFECTIONS DURING THE COVID-19 PANDEMIC PERIOD: AN INTEGRATIVE REVIEW
REGISTRO DOI: 10.5281/zenodo.12733048
Anna Victória Cardeal Simão Ribeiro1; Maria Clara Silva Nascimento1; Vitória de Almeida Ribeiro Alves1; Williana Ferreira dos Santos1; Jorge Messias Leal do Nascimento2; Lilian Filadelfa Lima Santos Leal2; Maria da Conceição Aquino de Sá2.
Resumo
As infecções causadas por microrganismos secundários ao SARS-CoV-2 tiveram uma relevância significativa nos pacientes hospitalizados com COVID-19. Sendo estas associadas à utilização de ventilação mecânica, à presença de comorbidades e multirresistência de bactérias patogênicas aos antibióticos disponíveis. O estudo tem como objetivo grupar e sintetizar estudos relacionados ao perfil bacteriano mais prevalente no sistema respiratório dos pacientes frente às infecções respiratórias durante o período da pandemia de COVID-19. Trata-se de uma revisão integrativa, a qual utilizou os seguintes descritores: BACTERIA AND INFECÇÃO RESPIRATÓRIA AND PANDEMIA OR COVID-19”nas bases de dados Medline via PubMed, Scielo, Google acadêmico e LILACS. Com a pergunta norteadora: “Qual o perfil bacteriano mais prevalente dos pacientes com infecção do sistema respiratório durante o período da pandemia de COVID-19? ”. Como resultado identificaram-se485 estudos inicialmente. Após análise dos critérios de inclusão e exclusão, permaneceram 72 publicações para leitura na íntegra, restando 17 artigos que compuseram a amostra. As evidências selecionadas dos artigos permitiram a identificação dos patógenos mais prevalentes frente as coinfecções: S. aureus, A. baumannii, K. pneumoniae e P. aeruginosa. Conclui-se que a compreensão desses perfis bacterianos não apenas contribui para a seleção mais precisa de estratégias terapêuticas, incluindo a administração de antibióticos específicos, mas também ressalta a importância de uma abordagem integrada no cuidado desses pacientes.
PALAVRAS-CHAVE: Bactéria. Coinfecções. Infecção Respiratória. Pandemia. COVID-19.
1. INTRODUÇÃO
As doenças respiratórias, agudas ou crônicas, encontram-se entre as patologias com maiores índices mundiais de morbidade e mortalidade, afetando idosos, adultos e crianças. De acordo com dados da Organização Mundial da Saúde, essas enfermidades são responsáveis por cerca de 14% das mortes no mundo (ASSUNÇÃO et al., 2018).
Nesse contexto, as infecções do trato respiratório podem ser categorizadas em superiores e inferiores, sendo a origem comumente associada a patógenos virais ou bacterianos. As infecções do trato respiratório inferior são consideradas mais graves, apresentando maiores taxas de morbimortalidade em comparação às do trato superior, pois o pulmão, um órgão vital, está entre os componentes do trato inferior (CHARLSON et al., 2011).
Uma infecção importante do trato respiratório inferior é a Pneumonia Adquirida em Ambiente Hospitalar por Ventilação Mecânica (PAV), sendo a principal causa de mortalidade em pacientes críticos. A PAV está associada à característica invasiva do procedimento de ventilação mecânica (TIMSIT et al., 2017). O diagnóstico das PAV ocorre com maior frequência após 48 horas do início do uso da ventilação artificial, um tratamento crucial em pacientes críticos com insuficiência respiratória. Entre as Infecções Relacionadas à Assistência à Saúde (IRAS), a pneumonia destaca-se, com 86% dos casos relacionados à ventilação mecânica (CABRAL et al., 2020).
No contexto da COVID-19 (Coronavírus Disease 2019), uma doença viral altamente transmissível, causada pelo coronavírus conhecido como Coronavírus de Síndrome Respiratória Aguda Grave 2 (SARS-CoV-2), a presença de infecções secundárias ao SARS-CoV-2 é de grande importância. Essas infecções foram especialmente relevantes para pacientes hospitalizados com COVID-19, que apresentaram taxas consideráveis de coinfecções, associadas ao uso de ventilação mecânica e presença de comorbidades, resultando em um aumento na mortalidade desses pacientes (MUSUUZA et al., 2021).
Devido à necessidade de tratamento rápido para casos graves de infecções associadas à COVID-19, a antibióticoterapia foi iniciada empiricamente ao longo da pandemia, antes de se estabelecer precisamente o diagnóstico bacteriológico. Esse uso indiscriminado de antibióticos durante o tratamento da COVID-19 acarreta preocupações significativas sobre a resistência bacteriana, um fenômeno que ocorre espontaneamente durante a evolução bacteriana, sendo acelerado nesse contexto, gerando grande preocupação (LUCIEN et al., 2021).
Assim, considerando a associação do vírus SARS-CoV-2 com a pneumonia adquirida por ventilação mecânica (PAV) e a multirresistência desenvolvida pelas bactérias ao longo do tempo, torna-se necessário identificar as cepas com maior perfil de resistência. O grupo “ESKAPE” é particularmente relevante, apresentando aumento significativo na resistência a uma ampla gama de fármacos e alta virulência. Os patógenos desse grupo, incluindo Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa e Enterobacter spp, estão majoritariamente associados a infecções do trato respiratório (ASSUNÇÃO et al., 2023).
Em Unidades de Terapia Intensiva (UTI), a multirresistência dessas bactérias torna-se cada vez mais uma ameaça à saúde pública, aumentando o risco de infecções, especialmente em relação aos agentes etiológicos da pneumonia, responsáveis por 30% das infecções hospitalares (GATO et al., 2021).
Além disso, a resistência aos fármacos é uma habilidade naturalmente desenvolvida pelos microrganismos patogênicos quando expostos a medicamentos ao longo do tempo. Isso permite que produzam enzimas que reduzem a eficácia dos medicamentos, dificultando o controle e a cura de infecções (TIAGO et al., 2020).
Dentre os microrganismos do grupo ESKAPE, a Klebsiella pneumoniae se destaca como produtora de enzimas capazes de aumentar sua resistência. Pertencente ao grupo das gram-negativas, a K. pneumoniae está presente tanto no ambiente hospitalar quanto fora dele, causando altas taxas de mortalidade devido à sua resistência, manifestando-se, em sua forma mais grave, por meio de septicemia, infecções respiratórias e do trato urinário (DELGADO et al., 2019).
Essa crescente multirresistência de bactérias patogênicas aos antibióticos disponíveis é preocupante, visto que as infecções hospitalares, no Brasil e em outros países, tornam-se mais prolongadas e muitas vezes de difícil tratamento, resultando no aumento das taxas de óbito entre os pacientes que desenvolvem esse tipo de infecção (TIAGO et al., 2020).
Assim, esta revisão tem como objetivo sintetizar estudos relacionados ao perfil bacteriano mais prevalente no sistema respiratório dos pacientes diante das infecções do trato superior durante o período da pandemia de COVID-19, publicados entre os anos de 2019 a 2023.
2. METODOLOGIA
Para a realização dessa revisão, foi elaborada uma pergunta norteadora: Qual é o perfil bacteriano mais prevalente nos pacientes com infecção do sistema respiratório durante o período da pandemia de COVID-19?
O estudo foi desenvolvido em 5 etapas, incluindo: 1) identificação do tema e definição da questão de pesquisa; 2) busca de literatura e coleta dos dados; 3) seleção dos estudos com base nos critérios de inclusão e exclusão; 4) análise crítica dos dados; 5) apresentação dos resultados da revisão.
As bases de dados selecionadas para a pesquisa dos estudos primários foram: Medical Literature Analysis and Retrieval System Online (MEDLINE), via PubMed, Scientific Electronic Library Online (Scielo), Google Acadêmico e Literatura Latino-Americana e do Caribe em Ciências da Saúde (LILACS).
Para a estratégia de busca, foram utilizados os operadores booleanos “AND” e “OR”, com as seguintes combinações de descritores: “BACTERIA AND INFECÇÃO RESPIRATÓRIA AND PANDEMIA OR COVID-19”. Diante disso, foram seguidos os seguintes critérios de inclusão: escritos em português, inglês e espanhol, data de publicação do estudo (2019-2023), tipo de estudo (artigo científico, revisão sistemática, metanálises, estudos observacionais, transversais, descritivos, coorte e retrospectivos), estudos baseados na temática proposta.
Os critérios de exclusão abrangeram: publicações que não respondiam à questão específica desta revisão, escritos fora da margem da data de publicação escolhida, os que se repetiam nas diversas bases pesquisadas, e aqueles enquadrados como teses, monografias, editais periódicos científicos e cartas.
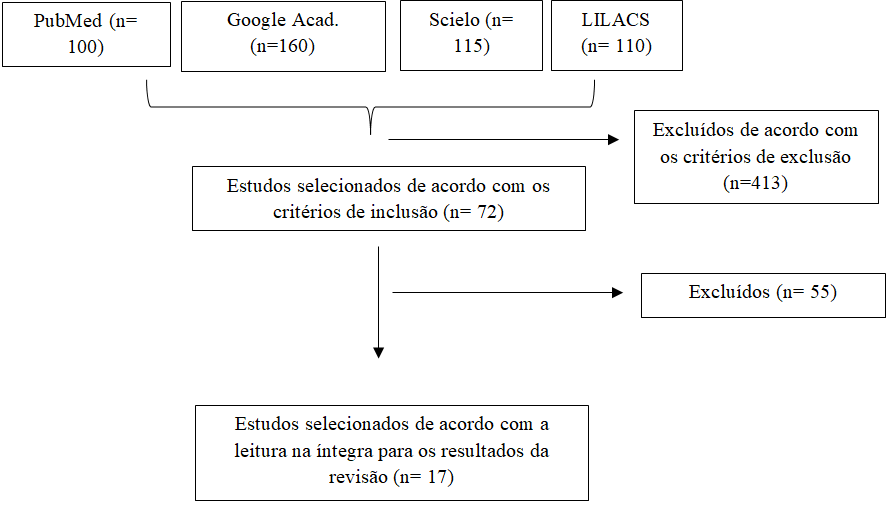
Dessa forma, a busca identificou inicialmente 485 estudos, dos quais 110 eram do LILACS, 160 do Google Acadêmico, 115 da Scielo e 100 no PubMed. Após a análise dos critérios de inclusão e exclusão, juntamente com a leitura do título e resumo, restaram 72 estudos para serem lidos na íntegra. Após a leitura desses, 17 artigos constituíram os resultados finais desta revisão. O desenvolvimento da busca está descrito na Figura 1.
Figura 1. Fluxograma da busca
3. RESULTADOS
Quadro 1- Quadro síntese dos estudos incluídos nos resultados da revisão.
Identificação Título Local Foco do Estudo Principais resultados Kariyawasam et al., (2022) Antimicrobial resistance (AMR) in COVID-19 patients: a systematic review and meta-analysis (November 2019–June 2021). Canadá Realizar uma revisão sistemática e uma metanálise durante os primeiros 18 meses da pandemia para quantificar a prevalência e os tipos de organismos coinfectantes resistentes em pacientes com COVID-19 e explorar as diferenças entre ambientes hospitalares e geográficos. A prevalência agrupada de coinfecção com organismos bacterianos e fúngicos resistentes foi de 24% (IC 95% 8–40%; n = 25 estudos: I = 99%) e 0,3% (IC 95% 0,1–0,6%; n = 8 estudos. I = 78%), respectivamente. Entre os organismos multirresistentes, Staphylococcus aureus resistente à meticilina, Acinetobacter baumannii resistente a carbapenem, Klebsiella pneumoniae, Pseudomonas aeruginosa e Candida auris multirresistente foram notificados com maior frequência. Suleiman et al., (2023) A meta-meta-analysis of co-infection, secondary infections, and antimicrobial resistance in COVID-19 patients. Banglash O objetivo desta metanálise secundária é analisar coinfecção, infecções secundárias e resistência antimicrobiana (RAM) em pacientes com COVID-19. Dados extraídos de 19 metanálises reuniram uma taxa resumida de coinfecção de 1,94% para Acinetobacter baumannii entre pacientes com COVID-19; 4,4% Chlamydia pneumoniae; 6,55% Staphylococcus coagulase negativo 2,72% Enterococcus faecalis 4,44% de Escherichia coli 1,85% de Estreptococos do Grupo A; 6,48% Haemophilus influenzae 5,16% Klebsiella oxytoca; 2,64% Klebsiella; 4,38% Legionella pneumophila; 4,29% Moraxella catarrhalis; 1,65% MRSA (Staphylococcus aureus resistente à meticilina); 2% MSSA (Staphylococcus aureus sensível à meticilina); 11,85% Mycoplasma pneumoniae; 1,87% Pseudomonas aeruginosa; 17,39% Staphylococcus aureus, 7,8% de Streptococcus pneumoniae. Musuuza et al., (2021) Prevalence and outcomes of co-infection and superinfection with SARS-CoV-2 and other pathogens: A systematic review and meta- analysis Estados Unidos O objetivo deste estudo foi examinar a ocorrência de coinfecções e superinfecções e seus resultados entre pacientes com infecção por SARS-CoV-2. Entre os pacientes com coinfecções, as três bactérias mais frequentemente identificadas foram Klebsiella pneumoniae (9,9%), Streptococcus pneumoniae (8,2%) e Staphylococcus aureus (7,7%). Entre aqueles com superinfecções, as três bactérias mais frequentemente identificadas foram Acinetobacter spp. (22,0%), Pseudomonas (10,8%) e Escherichia coli (6,9%). Yang et al., (2021) Molecular epidemiology of Pseudomonas aeruginosa isolated from lower respiratory tract of ICU patients China O presente estudo determina a epidemiologia molecular através do método de impressão digital de DNA e a resistência a medicamentos de P. aeruginosa isolada de pacientes com LTRIs internados em UTI. Dos 137 Pseudomonas sp isolados de pacientes de UTI, 57,7% dos isolados foram relatados como P. aeruginosa. A prevalência global de P. aeruginosa entre todos os pacientes incluídos foi de 34,5%. Montilla-Velásquez et al., (2021) Respiratory pathogens in children with acute lower respiratory infection detected through multiplex real-time PCR Colômbia Caracterizar os patógenos identificados em amostras de swab nasofaríngeo por meio de multiplex real- reação em cadeia da polimerase temporal (RT-PCR), bem como variáveis clínicas e achados laboratoriais em crianças <5 anos com diagnóstico de infecção aguda do trato respiratório inferior (ALRTI) e hospitalizadas em Bogotá D.C., Colômbia. As bactérias mais frequentes foram H. influenzae (20%) e S. pneumoniae (17,93%). Aydemir et al., (2021) Secondary bacterial infections in patients with coronavirus disease 2019-associated pneumonia Turquia Mostrar a frequência de infecções secundárias e condições de resistência em pacientes com doença coronavírus 2019 internados em unidade de terapia intensiva. A taxa de infecção do trato respiratório e/ou infecção da corrente sanguínea foi de 21%. Acinetobacter baumannii, Klebsiella pneumoniae e Pseudomonas aeruginosa em cultura de aspirado traqueal; Estafilococos coagulase-negativos, K. pneumoniae e A. baumannii em hemocultura; e Escherichia coli, K. pneumoniae e Enterococcus faecalis na urocultura foram os microrganismos mais comuns. Pourajam et al., (2022) Secondary Bacterial Infection and Clinical Characteristics in Patients With COVID-19 Admitted to Two Intensive Care Units of an Academic Hospital in Iran During the First Wave of the Pandemic Irã Determinar a frequência de infecção bacteriana secundária, uso de antibióticos e características clínicas em pacientes internados na UTI com pneumonia grave por SARS-CoV-2. Os agentes mais prevalentes causadores da infecção bacteriana secundária foram Klebsiella pneumoniae (n = 44) e Acinetobacter baumannii (n = 33). A maioria dos pacientes com infecção bacteriana secundária apresentou extensa resistência aos medicamentos. Saini et al., (2021) Paradigm Shift in Antimicrobial Resistance Pattern of Bacterial Isolates during the COVID-19 Pandemic India Este estudo teve como objetivo observar os pacientes com COVID-19 hospitalizados de 1º de março de 2019 até 31 de dezembro de 2020 e para avaliar o padrão de RAM de agentes bacterianos isolados Foram coletados 494 isolados bacterianos (sangue e urina) obtidos obtidos de pacientes com SARS-CoV-2 internado em UTI. Do total de isolados bacterianos, 55,46% eram gram negativos e 44,53% eram patógenos gram-positivos. Das amostras de sangue processadas, os isolados mais comuns foram CoNS (Staphylococcus coagulase negativa) e Staphylococcus aureus. Entre os urinários isolados, os patógenos mais comuns foram Escherichia coli e Staphylococcus aureus. Um total de 60% MRSA foi observado em amostras de urina e sangue. Singh et al., (2021) SARS-CoV-2 respiratory co-infections: Incidence of viral and bacterial co-pathogens Estados Unidos Analisar os padrões de coinfecção da infecção por SARS-CoV-2 nos Estados Unidos. Uma análise detalhada dos patógenos coinfectantes mostraram S. aureus, H. influenzae, S. pneumoniae e K. pneumoniae para ser as coinfecções bacterianas mais prevalentes tanto no SARS-CoV-2- pacientes positivos e negativos. Bezerra et al., (2020) SARS-CoV-2 como agente causador da COVID-19: Epidemiologia, características genéticas, manifestações clínicas, diagnóstico e possíveis tratamentos Brasil Realizar uma revisão integrativa das principais características da SARS-CoV-2 e dos aspectos epidemiológicos, clínicos e diagnósticos da COVID-19, bem como seus possíveis tratamentos. Para confirmação laboratorial, predominou o uso do teste de reação em cadeia da polimerase com transcriptase reversa (RT-PCR). Os sintomas mais recorrentes nos estudos de observação do paciente incluem febre, tosse, mialgia, fadiga e dispnéia. Syandrez Prima Putra, Taufik Hidayat e Rahma Tsania Zhuhra. (2022) SARS‑CoV‑2 persistence and infectivity in COVID‑19 corpses: a systematic review Indonésia Compreender a persistência e infecciosidade do SARS-CoV-2 em cadáveres de COVID-19. O RNA do SARS-CoV-2 no trato respiratório inferior foi observado em tecido pulmonar (138/155, 89,0%) e swab traqueal (9/20, 45,0%). O tempo máximo de persistência do RNA viral foi detectado com RT-qPCR após 4 meses em amostras de tecido pulmonar de cadáveres exumados (4/4, 100%). O sepultamento dos descendentes no inverno com baixas temperaturas constantes deve ser considerado um fator influenciador da persistência. Sharifipour et al., (2020) Evaluation of bacterial co-infections of the respiratory tract in COVID-19 patients admitted to ICU Irã Investigar superinfecções bacterianas em pacientes com COVID-19 em UTIs. Dos dezenove pacientes com COVID-19 foram identificadas dezessete cepas de Acinetobacter baumannii (90%) e duas de Staphylococcus aureus (10%). Provenzano et al., (2021) The impact of healthcare-associated infections on COVID-19 mortality: a cohort study from a Brazilian public hospital Brasil Analisar os fatores de risco para mortalidade hospitalar em uma coorte de pacientes internados em uma unidade de terapia intensiva recém-adaptada de um hospital público do Rio de Janeiro. A sepse na admissão aumentou a chance de morte hospitalar em 21 vezes. O fator de risco mais forte para morte foi o desenvolvimento de choque séptico durante a hospitalização e um em cada quatro pacientes tinha bactérias multirresistentes Karataş et al., (2021) Secondary bacterial infections and antimicrobial resistance in COVID-19: comparative evaluation of pre-pandemic and pandemic-era, a retrospective single center study Suécia Avaliar os padrões epidemiológicos e de resistência antimicrobiana (RAM) de patógenos bacterianos em pacientes com COVID-19 e comparar os resultados com grupos de controle da era pré-pandêmica e pandêmica. Observou-se que houve uma diminuição significativa nas Enterobacterales produtoras de beta-lactamase de espectro estendido e um aumento significativo da Acinetobacter baumannii, sendo considerada o principal patógeno nas infecções respiratórias de pacientes com COVID-19 (9,76%). Dhanusha Yesudhas, Ambuj Srivastava e M. Michael Gromiha. (2021) COVID‐19 outbreak: history, mechanism, transmission, structural studies and therapeutics. Alemanha Examinar a história e o mecanismo dos coronavírus e as características estruturais da proteína spike e seus principais resíduos pelas transmissoões humanas Os coronavírus podem cruzar a barreira das espécies e infectar humanos com consequências inesperadas para a saúde pública. A taxa de transmissão da infecção por SARS-CoV-2 é maior em comparação com as infecções estreitamente relacionadas por SARS-CoV.. Os principais resíduos da proteína spike têm afinidade de ligação mais forte com ACE2. Estas podem ser razões prováveis para a maior taxa de transmissão do SARS-CoV-2. Frangioti et al., (2021) Análise do impacto da pandemia da COVID-19 no consumo hospitalar de antimicrobianos. Brasil Analisar o aumento do consumo de antimicrobianos no período de janeiro a maio dos anos de 2019 a 2021 através da DDD e descrever ações para seu uso racional durante este período. Os resultados da DDD pré-pandemia (2019) e durante a pandemia (2020 e 2021) mostram um aumento importante do consumo de meropenem, vancomicina, polimixina B e tigeciclina. Os achados deste estudo corroboram outros relatos de aumento de microrganismos multirresistentes e do uso de antibióticos de amplo espectro durante a pandemia da COVID-19. O’ Toole. (2021) The interface between COVID-19 and bacterial healthcare-associated infections. Austrália Revisar dados recentes que indicam a ocorrência de infecções bacterianas de início hospitalar, inclusive com isolados resistentes a antibióticos, em pacientes com COVID-19. Patógenos resistentes a antibióticos que causam infecções associadas à assistência à saúde (IRAS) em pacientes com COVID-19 incluem Staphylococcus aureus resistente à meticilina, Enterobacterales resistentes a carbapenêmicos produtores de metalo-b-lactamase de Nova Delhi, Acinetobacter baumannii resistente a carbapenem, Acinetobacter baumannii resistente a antibióticos, espectro b-lactamase Klebsiella pneumoniae e enterococos resistentes à vancomicina.
Foram examinados 17 artigos na íntegra, sendo 5 provenientes da Scielo, 1 do Google Acadêmico, 6 do PubMed e 5 do Lilacs. A síntese dos resultados da pesquisa encontra-se no quadro 01. Quanto ao ano de publicação, foram identificados estudos abrangendo o período de 2019 a 2023, distribuídos da seguinte forma: 2 em 2020, 11 em 2021, 3 em 2022 e 1 em 2023.
Em relação ao tipo de estudo, 2 revisões sistemáticas e metanálises, 2 revisões sistemáticas, 3 metanálises, 1 revisões narrativas, 2 estudos transversais, 3 estudos retrospectivos, 1 estudo de coorte retrospectivo, 1 revisão integrativa e 2 estudos observacionais descritivos. Já ao local da pesquisa, 3 estudos ocorreram no Brasil, 1 na Alemanha, 1 na Austrália, 1 na Suécia, 2 no Irã. 1 na Indonésia, 2 nos Estados Unidos, 1 na índia, 1 na Turquia, 1 na China, 1 no Canadá, 1 no Bangladesh e 1 Colômbia.
As análises realizadas revelam que, durante a pandemia de COVID-19, os pacientes infectados pelo vírus SARS-CoV-2 frequentemente apresentaram coinfecções. Dos artigos examinados, 10 citam pelo menos um dos seguintes patógenos como prevalentes em infecções secundárias: Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii e Pseudomonas aeruginosa (KARIYAWASAM et al., 2022, SULEIMAN et al., 2023, MUSUUZA et al., 2021, YANG et al., 2021, VELÁSQUEZ et al., 2021, AYDEMIR et al., 2021, POURAJAM et al., 2022, SAINI et al., 2021, SINGH et al., 2021, SHARIFIPOUR et al., 2020, O’ TOOLE, 2021 e KARATAŞ et al., 2021).
Os perfis bacterianos predominantes nas coinfecções eram majoritariamente gram-negativos em comparação com os gram-positivos (SINGH et al., 2021 e SAINI et al., 2021). Entre os organismos multirresistentes, destaca-se a notificação mais frequente de S. aureus resistente à meticilina (gram positivo), A. baumannii resistente a carbapenem, K. pneumoniae e P. aeruginosa multirresistentes em pacientes com COVID-19 (KARIYAWASAM et al., 2022).
A resistência significativa aos medicamentos foi observada na maioria dos pacientes com infecção bacteriana secundária, contribuindo para o desenvolvimento de bactérias multirresistentes (POURAJAM et al., 2022). Em pacientes com coinfecções, as bactérias mais frequentemente identificadas foram Klebsiella pneumoniae, Streptococcus pneumoniae e Staphylococcus aureus. Já em casos de superinfecções, Acinetobacter spp., Pseudomonas e Escherichia coli foram as bactérias mais comumente identificadas (MUSUUZA et al., 2021).
Em análises de aspirado traqueal, hemoculturas e uroculturas de pacientes com COVID-19, foram identificados patógenos como Acinetobacter baumannii, Klebsiella pneumoniae e Pseudomonas aeruginosa em culturas de aspirado traqueal; estafilococos coagulase-negativos, K. pneumoniae, A. baumannii e Staphylococcus coagulase negativa, além de Staphylococcus aureus em hemoculturas; e Escherichia coli, K. pneumoniae, Enterococcus faecalis e S. aureus foram os microrganismos mais comuns em uroculturas (AYDEMIR et al., 2021e SAINI et al., 2021).
Além disso, outros patógenos, como Haemophilus influenzae e Mycoplasma pneumoniae (SINGH et al., 2021 e MONTILLA-VELÁSQUEZ et al., 2021 e SULEIMAN et al., 2023) foram encontrados em casos de coinfecção, mas não se destacaram em prevalência quando comparados aos patógenos mencionados acima.
4. DISCUSSÃO
Sabe-se que a Pandemia de COVID-19 foi responsável por 674,89 milhões de casos positivos e 6,75 milhões de mortes em todo o mundo, causando uma das mais significativas destruições em âmbito social, econômico e de saúde (SULEIMAN et al., 2023). Isso porque os coronavírus podem causar doenças respiratórias, gastrointestinais e do sistema nervoso central em humanos e animais além de possuírem a capacidade de se adaptar a um novo ambiente através de mutações, sendo uma ameaça constante uma vez que são programados para modificar o tropismo do hospedeiro (YESUDHAS et al., 2021).
Outrossim, é sabido que o vírus pode persistir meses após a morte e permanecer viável e infeccioso durante semanas, sob certas condições. Exemplo disso se dá através de um caso post-mortem que foi imediatamente levado para a câmara de resfriamento, onde a infectividade do SARS-CoV-2 ainda foi detectada até 17 dias após a morte, principalmente no pulmão, traqueia e perioral. Esta descoberta pode ser explicada pelo aumento da estabilidade do vírus a uma temperatura mais baixa, o que também pode aumentar a probabilidade de cultura celular positiva; sendo necessária maior precaução no manuseio de autópsias de cadáveres pré-resfriados (PUTRA et al., 2023).
Diante disso, o desafio da pandemia global em 2019, inicialmente foi diagnosticar com agilidade e precisão a doença COVID-19 diante da ampla variedade de manifestações clínicas, e em se tratando de um agente imunossupressor com alto potencial de levar a sepse, é necessário identificar a bactéria causadora da co-infecção para iniciar antibioticoterapia direcionada (SAINI, et al., 2021).
A análise de elementos clínicos e sociodemográficos que ampliam a probabilidade de um indivíduo contrair tais coinfecções pode abranger fatores como ambiente de saúde, condição socioeconômica, histórico de uso de antibióticos e período de internação hospitalar. Conforme evidenciado pelo estudo de Musuuza et al. (2021), até 19% dos pacientes com COVID-19 manifestam coinfecções, enquanto 24% apresentam superinfecções, resultando em um aumento significativo nas taxas de mortalidade.
De modo errôneo, as doenças infecciosas costumam ser atribuídas a um único patógeno, cuja correlação sindrômica fornece uma estimativa da probabilidade de diagnóstico e, como resultado, permite decisões sobre a terapia antimicrobiana empírica inicial. No entanto, hoje sabe-se que múltiplos agentes infecciosos podem estar envolvidos, mas as implicações clínicas, diagnósticas e terapêuticas desta situação ainda são desconhecidas. Isto ficou evidente pelo desenvolvimento de técnicas moleculares de multidetecção, que se caracterizam por um melhor desempenho operacional. O ensaio RT-PCR, por exemplo, busca genomas virais e bacterianos em uma única reação e relata a presença de múltiplos agentes causadores em menos de 2 horas (MONTILLA-VELÁSQUEZ et al., 2023).
Nesse sentido, dentre as bactérias identificadas nos pacientes co-infectados, destaca-se Klebsiella pneumonie, Streptococcus pneumoniae e Staphylococcus aureus, sendo essa uma causa frequente de superinfecção em outras doenças respiratórias, como a gripe. No que se refere a bactéria mais frequentemente identificada em pacientes superinfectados, tem-se destaque a Acinetobacter spp., sendo uma infecção comumente encontrada em pacientes com suporte ventilatório (MUSUUZA et al., 2021).
Ademais, a literatura descreve que as infecções virais são atenuantes das co-infecções bacterianas simultâneas ou secundárias, sendo os vírus mais encontrados o Influenza tipo A, Influenza tipo B e o vírus sincicial respiratório. Segundo Gupta (2008), estas são as principais causas de mortalidade em pandemias de gripe. O tropismo do vírus SARS-CoV-2, agente etiológico da doença COVID-19, pelo trato respiratório e o desenvolvimento da patogênese, corroboram com esses achados.
No contexto de elevada mortalidade, a OMS aconselha o início do tratamento empírico com antibióticos de amplo espectro em pacientes com suspeita ou risco de infecções bacterianas que poderiam levar à falência orgânica (POURAJAM et al., 2022). No entanto, o uso de antibióticos de amplo espectro sem a realização do antibiograma no período pandêmico acelerou o processo de resistência bacteriana levando ao aumento de organismos multirresistentes em todo o mundo (FRANGIOTI et al., 2021).
Segundo Musuuza et al. (2021) sabe-se que durante os estágios iniciais da pandemia de COVID-19, até 60% dos pacientes receberam prescrição de antimicrobianossendo para a maioria deles receitado azitromicina, que é um macrolídeo com resistência crescente a infecções Gram-positivas e Gram-negativas. Um estudo relatado por Kariyawasam et al. (2022) sugeriu um aumento na resistência à eritromicina em S. aureus, e este pode estar associado ao uso elevado de azitromicina. A resistência mais comum documentada foi em CRAB, MRSA, Klebsiella pneumoniae e Pseudomonas aeruginosa, embora alguns isolados de C. auris também tenham sido identificados.
Nesse contexto, uma análise transversal multicêntrica dos Estados Unidos relatou que 36,2% dos organismos Gram negativos isolados de pacientes com infecções do trato inferior internados em UTI foram identificados como P. aeruginosa.
No estudo de Aydemir et al. (2021) foi investigado a frequência de infecções bacterianas secundárias e o status de resistência em pacientes internados em UTI com diagnóstico de COVID-19, comprovando-se uma taxa de infecção do sistema urinário de 15,6%, infecção da corrente sanguínea de 14% e infecção do sistema respiratório de 8,4%. Os organismos mais comumente encontrados foram Acinetobacter baumannii, Klebsiella pneumoniae, e Pseudomonas aeruginosa na cultura de aspirado traqueal; Estafilococos coagulase negativos, K. pneumoniae, e A. baumannii em hemocultura; e Escherichia coli, K. pneumoniae, e Enterococcus faecalis na urocultura.
Neste mesmo estudo, ao se observar as taxas de resistência, A.baumannii era resistente à maioria dos antibióticos, exceto à colistina; as cepas de P. aeruginosas eram resistentes à maioria dos antibióticos, exceto amicacina, colistina, cefepim e imipenem. Por último, na K. pneumoniae, foi observada a maior sensibilidade ao meropenem (73%) e houve resistência à maioria dos antibióticos restantes.
Outrossim, segundo O’Telle (2021), reconhecer os organismos descritos como causadores das IRAS é ferramenta decisiva para identificar o perfil bacteriano das co-infecções relacionadas ao trato respiratório na COVID-19. O entendimento da origem bacteriana; hospitalar ou comunitária, representa o risco de desenvolver complicações hospitalares. No que diz respeito às SKAPES, a S. aureus resistente à meticilina (MRSA) têm maior probabilidade de ser derivada do uso de ventilador mecânico usado em pacientes críticos com pneumonia.
Em consonância com os estudos do período pandêmico, Yang et al. (2021) relaciona a bactéria P. aeruginosa como prevalente em 57,7% dos isolados de pacientes com infecção do trato respiratório em UTI, sendo encontrada grande variabilidade genética na relação de padrões de resistência. Em Sharifipour et al. (2020) há destaque A. baumanni e S. aureus em cultura de aspirado traqueal em pacientes com COVID-19. Em Karatas et al. (2021)A. baumanni apresentou novamente prevalência das co-infecções de trato respiratório.
Por fim, no estudo de Provenzano et al. (2021) 50% dos pacientes hospitalizados com COVID-19 tiveram comprometimento pulmonar que atingiram o parênquima pulmonar com alto risco de choque séptico. A presença de organismos multirresistentes em 1 a cada 4 pacientes aumentou o risco de morte.
5. CONCLUSÃO
Portanto, conclui-se que o perfil bacteriano mais prevalente entre os pacientes com coinfecções durante a pandemia de COVID-19 envolveu pelo menos um desses seguintes patógenos: Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii e Pseudomonas aeruginosa. Logo, a compreensão desses perfis bacterianos não apenas contribui para a seleção mais precisa de estratégias terapêuticas, incluindo a administração de antibióticos específicos, mas também ressalta a importância de uma abordagem integrada no cuidado desses pacientes. Isso porque a coexistência de infecções virais e bacterianas durante a pandemia implica em desafios únicos, demandando uma vigilância clínica mais atenta e uma resposta adaptativa dos profissionais de saúde.
REFERÊNCIAS
ASSUNÇÃO, R.G; PEREIRA, C.D.S; ABREU, A.G. Ventilation-Associated Pneumonia and its relation to ESKAPE pathogens. Revista Saúde e Desenvolvimento Humano. V. 11, n.3, p. 01-15, 2023. Disponível em: https://revistas.unilasalle.edu.br/index.php/saude_desenvolvimento/article/view/9536/4194. Acesso em janeiro de 2023.
ASSUNÇÃO, R.G; ABREU, A.G; PEREIRA, W.A. Pneumonia bacteriana: aspectos epidemiológicos, fisiopatologia e avanços no diagnóstico. Revista de Investigação Biomédica. V.10, p. 83-92, 2018.Disponível em: https://www.researchgate.net/publication/330548733_PNEUMONIA_BACTERIANA_ASPECTOS_EPIDEMIOLOGICOS_FISIOPATOLOGIA_E_AVANCOS_NO_DIAGNOSTICO. Acesso em janeiro de 2023.
AYDEMIR, O. et al. Secundary bacterial infections in patients with coronavirus disease 2019-associated pneumonia. Revista da Associação Médica Brasileira, V. 68, n. 2, p. 142-146, 2022. Disponível em: https://www.scielo.br/j/ramb/a/H9K7zRBBsPNPbgMR8KYJpqQ/. Acesso em janeiro 2023.
BEZERRA, V. et al. SARS-CoV-2 como agente causador da COVID-19: Epidemiologia, características genéticas, manifestações clínicas, diagnóstico e possíveis tratamentos. Brazilian Journal of health Review, v. 3, n. 4, p. 8452-8467, 2020. Disponível: https://ojs.brazilianjournals.com.br/index.php/BJHR/article/view/13364. Acesso em janeiro de 2023.
CABRAL, B.G. et al. Preventive Care for Pneumonia Mechanical Ventilation Associated: Integrative Review. Revista Enfermagem Atual in Derme,v.21, p. 131-141, 2020. Disponível em: https://revistaenfermagematual.com.br/index.php/revista/article/view/542. Acesso em janeiro de 2023.
CHALRSON, E.S. et al. Topographical Continuity of Bacterial Populations in the healthy Human Respiratory Tract. American Journal of Respiratory and Critical Care Medicine. V. 184, p. 957-963, 2011. Disponível em: https://pubmed.ncbi.nlm.nih.gov/21680950/. Acesso em janeiro de 2023.
DELGADO, L.A. et al. Atividade Antibacteriana do Extrato Etanólico Bruto da Gossypium hirsutum L.contra Cepas de Klebsiella pneumoniae e Escherichia coli. Revista Eletrônica Acervo Saúde. v. 11, p. 1-6, 2019. Disponível em: https://acervomais.com.br/index.php/saude/article/view/227. Acesso em janeiro de 2023.
FRANGIOTI, M. et al. Análise do impacto da pandemia da COVID-19 no consumo hospitalar de antimicrobianos. Revista Qualidade HC, p. 40-46, 2021. Disponível em: https://repositorio.usp.br/item/003059153. Acesso em janeiro de 2023.
GATO, P.C. et al. Klebsiella Pneumoniae Bacterial Resistance Profile in The Intensive Care unit in a Teaching Hospital in West Pará During 2018 to 2019. Brazilian Journal of Development. V.7, p. 115549-115566, 2021. Disponível em: https://ojs.brazilianjournals.com.br/index.php/BRJD/article/view/41091. Acesso em janeiro de 2023.
KARATAŞ, M. et al. Secondary bacterial infections and antimicrobial resistance in COVID-19: comparative evaluation of pre-pandemic and pandemic-era, a retrospective single center study. Annals of Clinical Microbiology and Antimicrobials, 2021. Disponível: https://pubmed.ncbi.nlm.nih.gov/34353332/. Acesso em janeiro de 2023.
KARIYAWASAM, R.M. et al. Antimicrobial resistance (AMR) in COVID-19 patients: a systematic review and meta-analysis (November 2019-June 2021). Antimicrobial resistance and infection control. V. 11, p. 45, 2022. Disponível em: https://pubmed.ncbi.nlm.nih.gov/35255988/. Acesso em janeiro de 2023.
KHAN, M. et al. COVID-19: A Global Challenge with Old History, Epidemiology and Progress So Far. Moleculares. V.26, p. 01-25, 2021. Disponível em: https://pubmed.ncbi.nlm.nih.gov/33374759/. Acesso em janeiro de 2023.
LUCIEN, M. A. et al. Antibiotics and antimicrobial resistance in the COVID-19 era: Perspective from resource-limited settings. International Journal of Infectious Diseases. V. 104, p. 250-254, 2021. Disponível em: https://pubmed.ncbi.nlm.nih.gov/33434666/. Acesso em janeiro de 2023.
MUSUUZA, J.S; et al. Prevalence and outcomes of co-infection and superinfection with SARS-CoV-2 and other pathogens: A systematic review and meta-analysis. PLoS One. V. 16, n. 5, May 6, 2021. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8101968/. Acesso em janeiro de 2023.
MONTILLA-VELÁSQUEZ, M.D. et al. Respiratory pathogens in children with acute lower respiratory infection detected through multiplex real-time PCR. Revista de la Facultad de Medicina. V. 60, n. 3, p. 207, 2023. Disponível em: http://www.scielo.org.co/scielo.php?pid=S0120-00112022000300207&script=sci_abstract. Acesso em janeiro de 2023.
O’TOOLE, R. The interface between COVID-19 and bacterial healthcare-associated infections. Clinical Microbiology and Infection, n. 27 p. 1772-1776, 2021. Disponível em: https://pubmed.ncbi.nlm.nih.gov/34111586/. Acesso em janeiro de 2023.
POURAJAM, S. et al. Secondary Bacterial Infection and Clinical Characteristics in Patients With COVID-19 Admitted to Two Intensive Care Units of na Academic Hospital in Iran During the First Wave of the Pandemic. Frontiers in Cellular and Infection Microbiology, v. 12, n. 784130, 2022. Disponível em: https://pubmed.ncbi.nlm.nih.gov/35281440/. Acesso em janeiro de 2023.
PROVENZANO, B. et al. The impact of healthcare associated infections on COVID-19 mortality: a cohort study from a Brazilian public hospital. Rev Assoc Med Bras, v. 67, n. 7, p. 997-1002, 2021. Disponível em: https://www.scielo.br/j/ramb/a/yq3CssBMCN6QyWrxfS7C7jC/?lang=en. Acesso em janeiro de 2023.
PUTRA, S.P; HIDAYAT, T; ZHUHRA, R.T. SARS-CoV-2 persistence and infectivity in COVID-19 corpses: a systematic review. Forensic Science, Medicine, and Pathology. V. 19, p. 94-102, 2022. Disponível em: https://pubmed.ncbi.nlm.nih.gov/36001241/. Acesso em janeiro de 2023.
SAINI, V. et al. Paradigm Shift in Antimicrobial Resistance Pattern of Bacterial Isolates during the COVID-19 Pandemic. Antibiotics, v. 10, p. 954, 2021. Disponível em: https://pubmed.ncbi.nlm.nih.gov/34439004/. Acesso em janeiro de 2023.
SINGH, V. et al. SARS-CoV-2 respiratory co-infections: Incidence of viral and bacterial co-pathogens. International Journal of Infectious Diseases, v.105, p. 617–620, 2021. Disponível em: https://pubmed.ncbi.nlm.nih.gov/33640570/. Acesso em janeiro de 2023.
SHARIFIPOUR, E. et al. Evaluation of bacterial co-infections of the respiratory tract in COVID-19 patients admitted to ICU. BMC Infectious Diseases, p. 20:646, 2020. Disponível em: https://pubmed.ncbi.nlm.nih.gov/32873235/. Acesso em janeiro de 2023.
SULEIMAN, A.S. et al. A meta-meta-analysis of co-infection, secondary infections, and antimicrobial resistance in COVID-19 patients. Journal of Infection and Public Health. V. 16, n. 10, p. 1562-1590, 2023. Disponível em: https://pubmed.ncbi.nlm.nih.gov/37572572/. Acesso em janeiro de 2023.
TIAGO, K.P. et al. Frequência e resistência de uroculturas provenientes de pacientes internados na unidade de terapia intensiva do hospital municipal de Santarém-PA. Revista Brasileira de Análises Clínicas-RBAC. V.52, p 64-70, 2020. Disponível em: https://pesquisa.bvsalud.org/portal/resource/pt/biblio-1104475. Acesso em janeiro de 2023.
TIMSIT, J.F. et al. Update on ventilator-associated pneumonia. F100 Faculty Reviews. V. 6, nov. 2017. Disponível: https://pubmed.ncbi.nlm.nih.gov/29225790/. Acesso em janeiro de 2023.
YANG, X. et al. Molecular epidemiology of Pseudomonas aeruginosa isolated from lower respiratory tract of ICU patients. Brazilian Journal of Biology, v. 81, n. 2 p. 351-360, 2021. Disponível: https://pubmed.ncbi.nlm.nih.gov/32491054/. Acesso em janeiro de 2023.
YESUDHAS, D; SRIVASTAVA, A; GROMIHA, M. COVID-19 outbreak: history, mechanism, transmission, structural studies and therapeutics. Infection. V. 49, n. 2, p.199-213, 2021. Disponível: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7472674/. Acesso em janeiro de 2023.
Currículo do autor

